さて,前回までの直感RDF!でRDFの概要を学び,イカシタRDFを作成したり,情報収集する方法を紹介しました。今回は,外部のデータと繋げて検索する方法をご紹介します。
同内容はブログでも公開しております。
目的ー何するの?
今回は今まで作成したRDFのデータを使って,薬のターゲット情報や,特許情報を取得するまでを書いていきたいと思います。
外部のデータのことを知ろう。
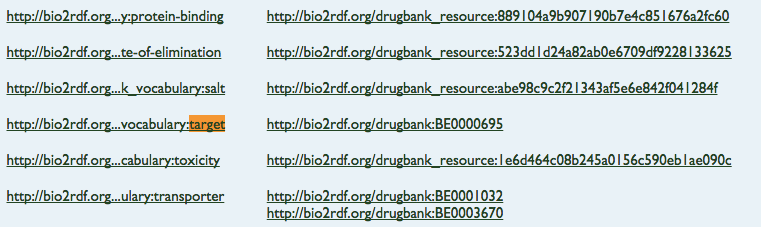
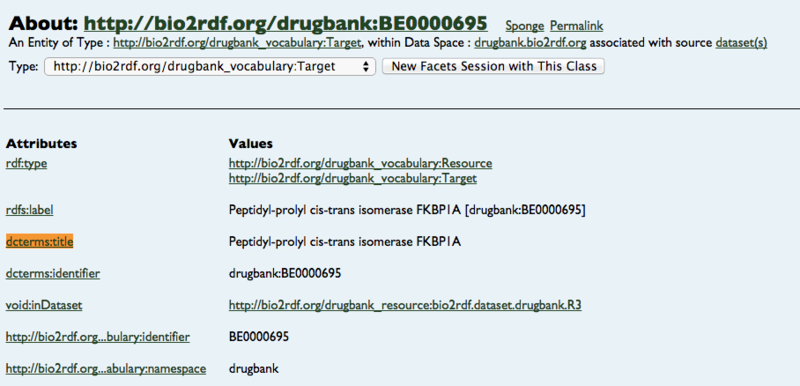
外部のデータと繋げていくためには,外部のRDFデータについて知る必要があります。ここでは,薬のターゲット情報が知りたいので,bio2rdfで提供されているdrugbankの情報を見てみましょう。bio2rdfのトップページから,Vioxx [drugbank:DB00533 ]を選択します。右の検索窓に,drugbank:DB00143が表示されると思うのですが,これを自分の手持ちのデータのdrugbank ID - ここでは, drugbank:DB00864 に変更し,検索します。すると,このようなページが開かれるはずです。ここからtargetを探ります。あ,ありました。
あ,あれ,なんか求めてたのと違う?URIじゃん,と思ったあなた,クリックしてみてください。 targetが見つかりました!このように,ターゲットを1つのURIで表記をしておくと,他の化合物のターゲットだった時にも使いまわすことができるので,URI を使用していると思われます。ここでの例のように, URI が Object になり,今度はそれを Subject としてその先に Predicate とObject が書かれており,詳しい説明(多くの場合文字列)があるいう RDF のデータは良く存在します。自分で RDF のデータを作っていく時にも参考となるはずです。ただあまりグラフが深くなると,どこに何があるのか分からなくなってしまうという恐れもあります。
さて中身をご覧頂くと,おそらくバイオ関係で研究をされている方なら,この時点で遺伝子名,生物種,他の関連するデータベースの ID などが書かれているので興味を持って頂けるかも?と感じています。
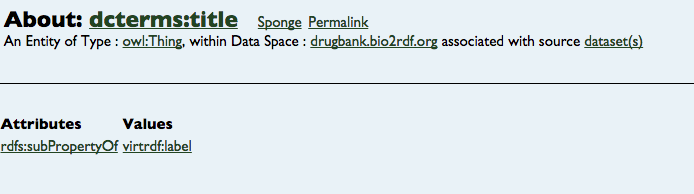
あ,でも,dcterms:titleってなんだろう?と思ったあなた,クリックしてみてください。
レッツ検索!以下の様なクエリを入れてみましょう- bio2rdfで提供している drugbank の SPARQL Endpoint
PREFIX void: <http://rdfs.org/ns/void#>
PREFIX dv: <http://bio2rdf.org/bio2rdf.dataset_vocabulary:>
prefix drgb: <http://bio2rdf.org/drugbank_vocabulary:>
SELECT *
WHERE {
<http://bio2rdf.org/drugbank:DB00864> drgb:target ?o.
?o dcterms:title ?target.
}
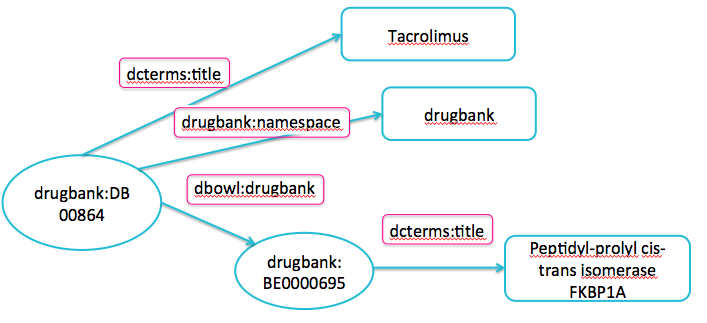
ここでは,http://bio2rdf.org/drugbank:DB00864のURIを主語として,targetとなりうる?o, Objectを探し出し,
そのtitleを出して,?targetとして出力させています。
検索結果
よっしゃ,ビンゴ!
ここまでで,事前準備完了です。手持ちのデータと繋げてみましょう!!
SPARQLで繋げ!
前回ご紹介した,DrugTargetのSPARQL Endpointを開いていください。SERVICEを使って検索をします。
prefix drgb: <http://bio2rdf.org/drugbank_vocabulary:>
prefix dbowl: <http://dbpedia.org/ontology/>
prefix orphan: <http://www.nibio.go.jp/orphanDrugTarget#>
select distinct ?name ?product ?drugbank ?title ?target_name
where {?s drgb:name ?name;
drgb:product ?product;
dbowl:drugbank ?drugbank.
SERVICE <http://drugbank.bio2rdf.org/sparql>
{?drugbank <http://purl.org/dc/terms/title> ?title;
drgb:target ?target.
?target drgb:name ?target_name.
}
}
解説しましょうっ。
まず,select distinct のdistinctは重複を除く,との意味です。その後は,いつもどおり,?sでSubjectを探せ,drgb:name ?nameで手持ちのデータの薬の名前出せ,";"でつなぐと,Subjectが同じままで検索できます。drgb:product ?productで製品名を出せ,dbowl:drugbank ?drugbankで?drugbankのURIを出せ,と指定しています。次の,SERVICE以降の;<http://drugbank.bio2rdf.org/sparql>では,外部のSPARQL Endpointを指定し,先ほど取り出した,?drugbankのURIをSubjectとし,<http://purl.org/dc/terms/title> ?titleで名前を出し(prefixを使わない場合はURIを直接書いてしまってOKです),
drgb:target ?targetで?targetのURIにつなぎ,?target drgb:name ?target_name.でターゲットの名前を出しています。
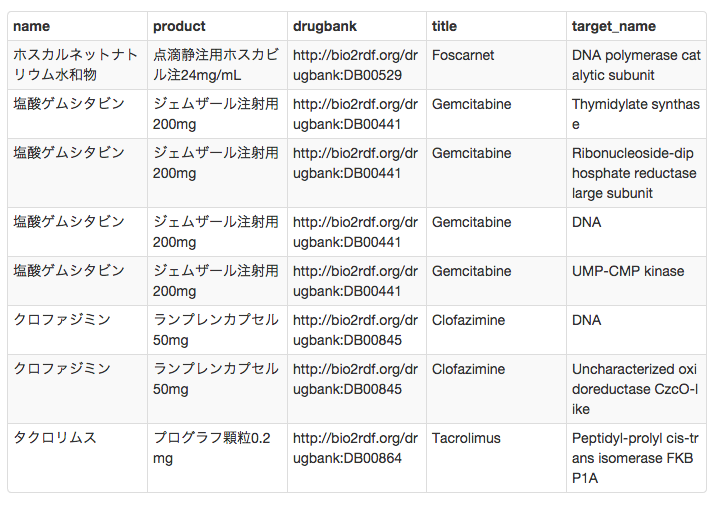
実行結果
もとのデータより表が大きいと感じる方がいらっしゃるかもしれませんが,どこかのデータが多い場合には,その多いデータに合わせて,表の行数も多くなります。ここでは,target_nameの数が多いので,それに合わせてありますね。
ちょっと応用。
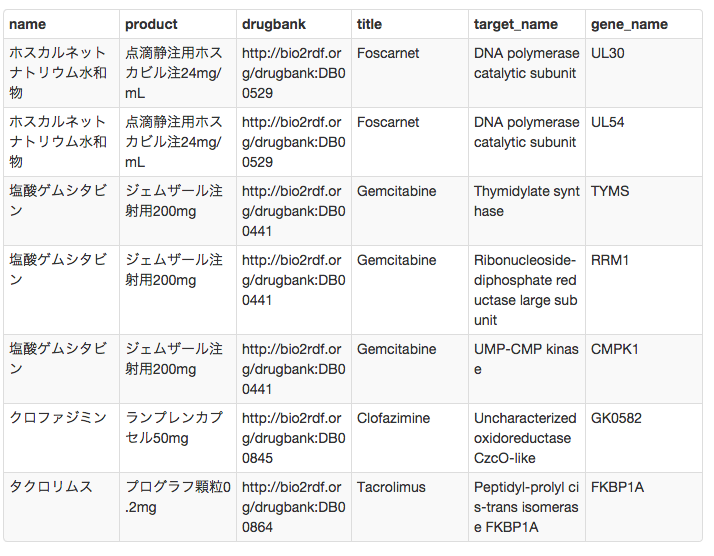
ここで,遺伝子名も見たいなぁと思った場合には,以下のようにチョビっと書き換えればOKです。この辺りで,もとのdrugbankのRDFを調べたことが効いていることが分かると思います。
prefix drgb: <http://bio2rdf.org/drugbank_vocabulary:>
prefix dbowl: <http://dbpedia.org/ontology/>
prefix orphan: <http://www.nibio.go.jp/orphanDrugTarget#>
select distinct ?name ?product ?drugbank ?title ?target_name ?gene_name
where {?s drgb:name ?name;
drgb:product ?product;
dbowl:drugbank ?drugbank.
SERVICE <http://drugbank.bio2rdf.org/sparql>
{?drugbank <http://purl.org/dc/terms/title> ?title;
drgb:target ?target.
?target drgb:name ?target_name;
drgb:gene-name ?gene_name.
}
}
実行結果
特許の情報を知りたし。
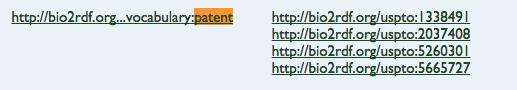
そんな場合には,先ほどのdrugbankの解説ページに戻りましょう。3ページ目に特許らしき記述を発見しました。
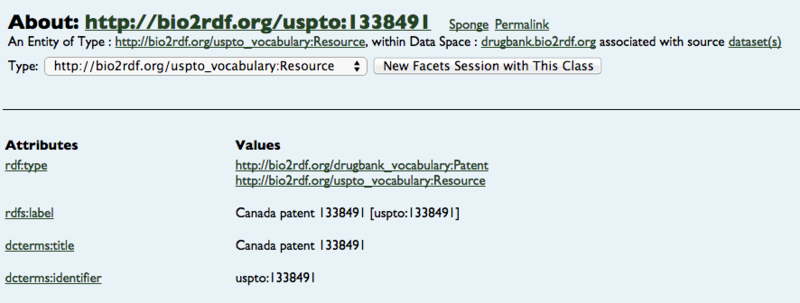
特許番号は何かしら?とクリックすると,ありました♪先ほどのターゲット情報と同様の方法を使えば,特許番号の取得が可能です。ぜひ,試してみてください!
おなか空いたので宿題です。皆さんなら出来るはずです(まさかの丸投げ)。
他にも提供されているSPARQL Endpointを使ったり,自分でトリプルストアを立てて公開されているRDFデータをダウンロードしたりして,色々と検索してみてください。言葉をつなぐように,データをつなぎ,新たな発見を見出すことが出来るかもしれません。また,複数のSPARQL Endpointを繋いで検索をすることもできます。これにより,世界中のデータとどんどこさと繋げていくことが出来ます。そして,それぞれの機関で作られているので,情報は勝手に新しくなってくれます。ティム・バーナーズ=リーが想像した世界に近づけます。やっほー。
ただ,現在のところストレスを感じるくらいに遅いので,技術革新を待つか,必要なデータは自分でダウンロードして入れしまいましょう。
トリプルストアを自分で立てるなら?
最後に簡単に自分でトリプルストアを立てる時に役立ちそうなURLを紹介します。
以下の情報を参考にしてみてください。
-
となんだか,日本語と手前味噌のエントリを紹介してしまいましたが,各トリプルストア(有名ドコロはVirtuoso, GraphDB<以前のOWLIM>, Fusekiといったところでしょうか)の公式ページにも掲載されています。また,Learning SPARQLの書籍をご購入された方は,ARQというコマンドを使用して検索するのも手軽で便利です。こちらのエントリをご参考に,ぜひ導入してみてください。詳しい使い方は,本の記述をご参照ください。それでは,またお会いしましょう!!Adios!!!(多分まだ色々と修正します^^;)
直感RDF!! シリーズ
その1-RDFとは。
その2 -使いやすいRDFを作って,検索しよう。