直感シリーズ第二弾,その2です。えっと,次は書くといいながら,300日弱の時が過ぎました。なんやて!(エセ関西弁)。ある程度ネタが集まったので書いていくことにします。
同内容はブログでも公開しております。
手持ちのデータをRDF化しよう。
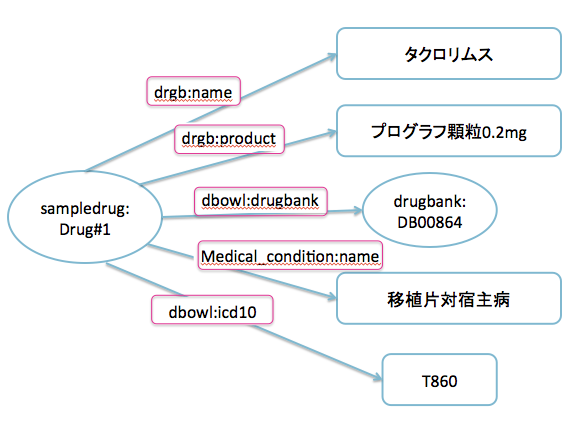
実際にRDFを他のRDFと統合しようと思ったら手元のデータのRDF化が必要です。RDF自体の説明は前回のエントリをご覧頂くとして,ここでは,こんなデータを用意してみました。ある薬のデータです。シチュエーションとしては,ある研究所の研究支援部(あくまでFictionにしてあげてください)が支援している薬のデータをRDF化したとします。中身は,ある薬を基準として,その名前,製品名,drugbankID,疾患名,疾患分類であるicd10のIDをつけています。rdf(記法はttl形式)の中身はこうなります。
@prefix drugbank: <http://bio2rdf.org/drugbank:> .
@prefix drgb: <http://bio2rdf.org/drugbank_vocabulary:> .
@prefix dbowl: <http://dbpedia.org/ontology/> .
@prefix Medical_condition: <http://schema.org/MedicalCondition/> .
<http://sampledrug.jp/Drug#1>
drgb:name "タクロリムス"@ja;
drgb:product "プログラフ顆粒0.2mg"@ja;
dbowl:drugbank drugbank:DB00864;
Medical_condition:name "移植片対宿主病"@ja;
dbowl:icd10 "T860".
最初にprefixで以下の記述で省略したいURIを書いています。今回は可読性のためにもprefixを多用していますが,(drgb:nameは正式には,http://bio2rdf.org/drugbank_vocabulary:name です。ここで使われているURIをクリックすると,そのURI自体について,概略が説明されていることもあります(本当は,そのようなウェブページを作るのが望ましいとされています。ただ,今回はクリックしても真っ白だぜ)。
このRDFのdrgb:name,dbowl:drugbankの部分(<http://sampledrug.jp/Drug#1>の主語(Subject)に対して述語(Predicate)といいます。ちなみに"タクロリムス"@ja は目的語(Object)になります。)は汎用的なものを使うことが望ましいとされています。
上記のRDFをグラフにするとこんな感じになります。(Subjectについては,http://sampledrug.jp/Drug#をsampledrug: に省略しています。)
適切なURI(特にpredicate)を探せ
少しRDFの検索からは横道にそれますが,適切なURIの付与は個人的には,RDF(というかLOD←情報は色々とあると思うので検索してくだされ)において重要であると考えています。また,RDFのトリプルに適切なURIを指定する際の話を詳しく書いてある既存の記事が少ないので,記載します。
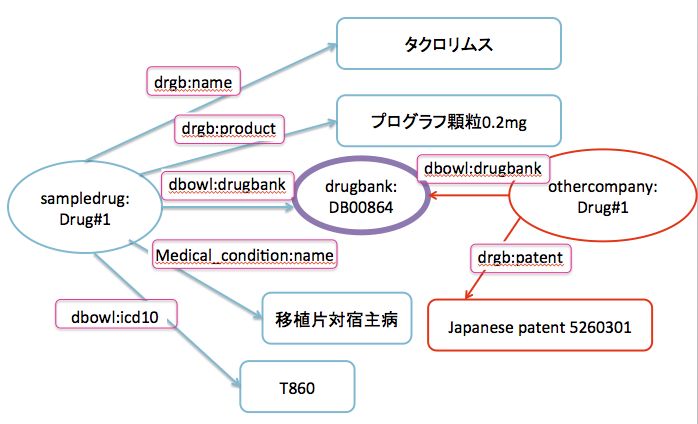
RDFの概念では,SubjectとPredicateには,URIを使うということが前提となっています。それはグラフ式のデータベースのそれぞれのResource(RDFのRのResource,具体的にはSubject, Predicate, Objectに該当するデータ)を他のデータとつなげようとした時に,このURIが同一かどうかで判断しているからです。例えば,先ほど書いた,<http://sampledrug.jp/Drug#1> dbowl:drugbank drugbank:DB00864.のトリプルですが,別のデータベースにおいて<http://othercompany.jp/Drug#29> dbowl:drugbank drugbank:DB00864.と書かれていた場合に,このdrugbank:DB00864は唯一無二のものとして認識することが出来ます。例えば,othercompanyが薬の特許を取得していてその情報をRDFで記述していたなら,
@prefix drugbank: <http://bio2rdf.org/drugbank:> .
@prefix drgb: <http://bio2rdf.org/drugbank_vocabulary:> .
@prefix dbowl: <http://dbpedia.org/ontology/> .
@prefix Medical_condition: <http://schema.org/MedicalCondition/> .
<http://othercompany.jp/Drug#29>
dbowl:drugbank drugbank:DB00864;
drgb:patent "Japanese patent 5260301".
のようになり,先ほどのデータと,drugbank:DB00864を通じてデータを的確に繋げることが出来ます。
グラフにするとこんな感じです。
こうすることで,例えば,移植片宿主病の薬はタクロリムスで,その特許はothercompanyにより取得されていて,その特許番号は日本国特許の場合526301である,ということが取り出せます。(詳しい方法は後述します。)
ここまで,さりげなくRDFを書いてしまいましたが,実際にはこの手持ちのデータをRDF化する過程で時間がかかります(多分これはデータが貯まれば貯まるほど前例が出来るので簡単になっていくはずです)。現状では,適切なURIを割り当てるということがなかなか難しい状況であります。私自身も現在手探りで進めているところではあるのですが,そのための方法(案)を幾つか紹介しましょう。
もっとも,適当に自分の好きなURIを割り当ててしまって後から修正するという考え方もあります。その辺の緩やかさも許容されるのは,RDFの強みです。特に既存のものが見つからなかった場合には無理をしなくてもいいんじゃないのかな?と個人的には考えています。(まともにやるとオントロジーの設計やらをする必要が出てしまうので…)
ここから述べる情報は私が個人的に本を読んだり,教えていただいたりした情報なので,もっと良い方法がありましたら,ご連絡ください〜また,今後掲載する情報は,現在私がいる環境に関連しているため,ライフサイエンスまわりの情報に偏っていることはご容赦ください。
ちなみに,Subjectは手持ちのデータの場合,自分の所属する機関や所有するウェブページのURIを使って生成すると良いと思います。確かその辺りの話は,Linked Data: Webをグローバルなデータ空間にする仕組みという本が詳しいのでご参照ください。
DBpedia
まず,汎用性が高いDBpediaから紹介しましょう。Wikipediaの情報を頑張ってセマンティックウェブに対応してくださっているプロジェクトです。論文を読むと,ライプツィヒ大学の方々が中心となってこのサービスを進めてくださっているようです。
ここでのURIを探す方法ですが,まず,wikipediaで手持ちのデータと関連するデータを検索します。
ここでは,タクロリムスを検索してみました。次に,Englishをクリックして,英語ページを表示します。
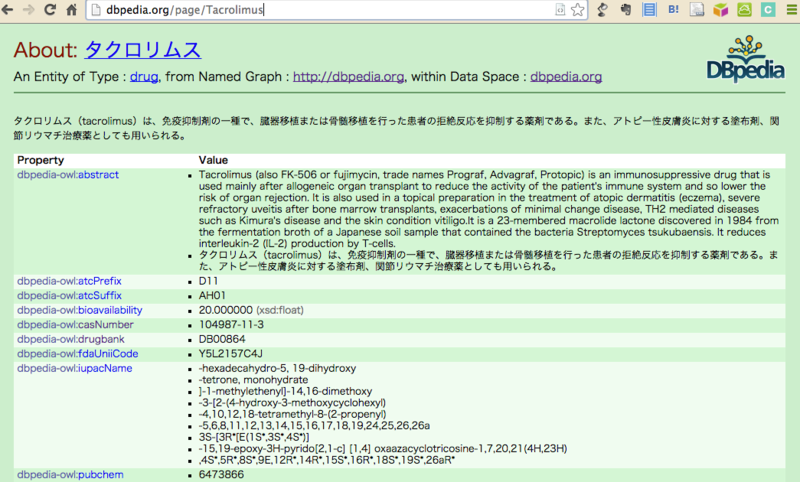
そして,[http://dbpedia.org/page/Tacrolimus]のように,dbpedia.org/page/ 以降に先ほどのコピーした文字列をペーストして,dbpediaのページを開きます。もちろん,直接調べたい情報の文字列を書き込んでいただいても構いません。すると以下の様なページが開かれます。
さて,ここでPropertyやValueをチェックしていきます。RDF的にこのページを捉えると,ページのURIである
http://dbpedia.org/page/Tacrolimus がSubject, PropertyがPredicate, ValueがObjectになります。
先の例では,dbpedia-owl:drugbankを使いましたが,DBpediaから参照して使用しました。ちなみにこのページのdbpedia-owl:drugbankをクリックすると,dbpedia-owl:drugbankをSubjectとしたPredicate, Objectに該当する情報(ここでdbpedia-owlはオントロジーです)を表示してくれます。(DBpediaの中の方のすごい労力を感じる…)。
そうして,適切なURIが見つかれば,[http://dbpedia.org/ontology/drugbank] を手持ちのRDFデータのリソースに付与してあげることが出来ます。
LODSTATS
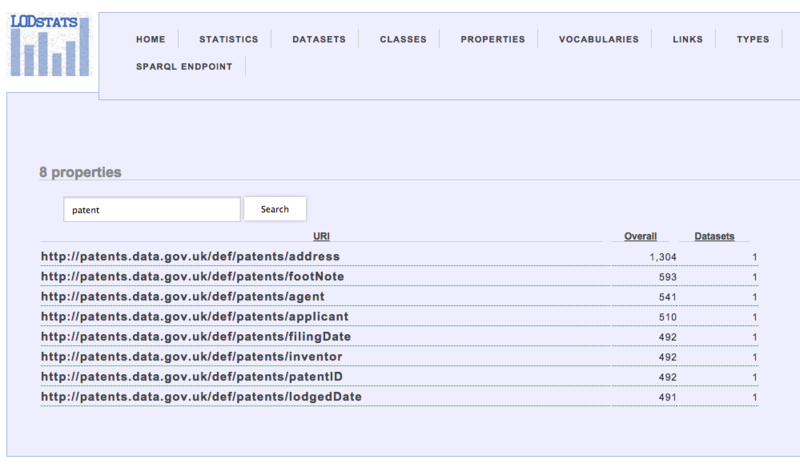
LODSTATSは統計的によく使われているPropertyなどを検索することが出来ます。例えば,ここでpatentを検索するとhttp://patents.data.gov.uk/def/patents/patentID といったPropertyと個数が出てきて汎用的に使われているデータを検索できます。
とはいえ,収録されているデータが網羅的ではないようなので,私にはあまり参考になりませんでした(・・;)。分野によっては参考になることもありそうです。
bio2rdf
bio2rdfは,バイオに特化したプロジェクトではありますが,かなり参考になるデータも多く含まれています。お目当てのデータベースの種類が決まっている場合にはトップ画面のセレクトボックスから,目的のデータベースを選び,検索をしてみると良いです。
すると先ほど紹介したDBpediaと同様に1つのページがSubjectに該当し,参考となるAttributes(Predicate)と内容であるValues(Object)を調べることが出来ます。bio2rdfでは,他にも個別のデータベースのSPARQL Endpointが存在し(例:MGI,Clinical Trials)それぞれのデータベース毎に検索が出来たり,データをまとめてダウンロード(ダウンロードページ)をすることが出来るので,バイオ関連の方は必見といって良いと思います。難点はそのデータベースを作っている機関が作成したRDFではないため,RDF化したデータの捉え方が正確ではない点とリンク切れも結構あります。
データベースを作成している研究機関によるRDF
各国の研究機関では,そのデータベースを作成している研究機関が持っているデータをRDF化して,公開,検索,またはダウンロードできる場合があります。専門的な内容となるので,詳細は省きますが,以下にリンク先を掲載します。
-
BioModels, BioSamples, ChEMBL, Expression Atlas, Reactome, Uniprotの各データベースをRDF化し,公開,検索,ダウンロードできます。検索例も充実しており,SPARQLの勉強にもなります。またそれぞれRDF化されたデータの概要やグラフ画像もあるので,自分の出したクエリがどういった経路を辿って出力されるのか,直感的に理解することも出来ます。例:ChEMBL documentation
-
上記のページに説明が細かく書いてあります。化学的な知識が無いと使いこなすことが難しそうですが(私はひと通りドキュメントを読んだのですが,実験用語の概念を理解して,ほぉ,と思っただけという状況です…すみません…。)専門的な検索が出来そうに見受けられます。検索するための機能は無さそうなので(現時点では),ダウンロードして使用するということが必要のようです。
-
keggの各エントリにはRDFでダウンロードできるページがついており,使用することができます。どうやら,最近SPARQLによる検索もできるようになったようです。とてもシンプルなRDFで構成されているので,実際にデータを繋げていく時にも便利ではないか,という印象を持っています。
-
こちらの検索窓に検索語を入れて,検索をすると,該当するSubjectやPredicateを検索できます。日本語での説明(pdbj)での説明はこちら。また,PDB上のタンパク質のデータは基本的にRDF形式でのダウンロードが可能です。しかし,PDBのデータと見せ方は綺麗だなぁ,といつも思います。
-
理研で公開されているデータをまとめて検索できるページなのですが,バイオリソース系を中心として,RDF形式でのダウンロードが可能です。例えば,シロイヌナズナフェノームデータベースやマウス系統リストのページにある
このテーブルをダウンロードするボタンを押すと,RDF形式でのダウンロードを選択できます。手持ちのデータがこれらのデータと近い場合には,かなり参考となるデータが揃っているはずです。ただ,SPARQL Endpointが公開されていなかったり,Predicateがcria315s1iなど,すぐに意味が分からない言葉が含まれていることが難点です。
他にもあるはずですが,有用なデータベースを紹介していなかったらご連絡ください〜。
全般的に,データを出している機関によるRDF化されたデータは,データの信頼度が高いと思われます。(中の人にもよるかもしれない)前述のbio2RDFはかなり便利なのですが,本家のデータベースのRDF化されたデータとデータリソースがかぶっていた場合,かなり書き方が異なることもあるので,注意が必要です。
ふう。他には何かあったかしら?あ,そうでした!
BioPortal
BioPortalは,どちらかというとオントロジーの集積といった色合いが強く,バイオロジーや医薬関連のオントロジーがこちらに集約されています。登録されているものについては,各オントロジーの詳しい説明や定義されている語彙の階層構造や関係性を検索することが出来ます。自分で開発したオントロジーを登録することも出来るので(やったことないですが,多分ユーザー登録が必要です。ちなみに微生物DBであるMicrobe DBで考えられたVocabularlyはこちらで登録されています。)。
しっかりとしたRDF(というよりオントロジーを記述するための言語のOWLかな、ここでは説明しませんが、オントロジーの設計にはこのフォーマットのほうがRDFより適しています。)を気を入れて作る場合には,大いに役立つサイトだと思います。また,オントロジーによってはSPARQLによる検索をかけることが出来(ということの知名度はあまり高くなかったはず), BioPortal SPARQL betaは,案外検索例も充実しています。疾患分類であるicd10自体のオントロジー検索は,こちらから行うことができました。ただ,個人的にはオントロジーを検索できても,そこからデータを繋げていくのは難しいので,辞書的なもの,あるいはハブ的なものととらえたほうが良いのかなぁと感じています。(そうでもないかもしれないので,そうでもない使い方をされている方は教えて下さい〜)
LODに関するプロジェクトとRDF
直接研究機関で出しているもの,ではないですが,アメリカの特許のRDF化や検索(SPARQL Endpointによる)やRDFを活用したアプリケーションについては,こちらのAKSWから見つけられます。先ほど紹介したDBpediaをはじめとしたRDF(というか,もっとひろくLOD(LODについての情報は転がっていると思うので,調べてあげてください))を活用した様々なプロジェクトがあるので,探しものが見つかるかもしれません。
Schema.org
Schema.orgは,htmlをmicordata, RDFa, JSON-LDでマークアップするための語彙を記述してまとめているサイトなのですが(余裕があったら後述しますが,RDF的に捉えると基本的に各ウェブページをSubjectとし,そこに含まれているデータを指定してObjectとし<文字列が多いですが,URIや画像等も指定できます> Predicate<原則Schema.orgの語彙>を使ってウェブページ1ページでRDFを表現するようなものです),ここで使われているプロパティもRDFに使用することが可能です。前述した手持ちのデータのRDF化でも```@prefix Medical_condition: .```で使用しています。こちらの http://schema.org/MedicalCondition には詳しく説明があります。Propertyの下の方にnameがあることがご確認いただけますでしょうか。今回のRDFのMedical_condition:nameはこちらから使用しています。
@prefix drugbank: <http://bio2rdf.org/drugbank:> .
@prefix drgb: <http://bio2rdf.org/drugbank_vocabulary:> .
@prefix dbowl: <http://dbpedia.org/ontology/> .
@prefix Medical_condition: <http://schema.org/MedicalCondition/> .
<http://sampledrug.jp/Drug#1>
drgb:name "タクロリムス"@ja;
drgb:product "プログラフ顆粒0.2mg"@ja;
dbowl:drugbank drugbank:DB00864;
Medical_condition:name "移植片対宿主病"@ja;
dbowl:icd10 "T860".
ファイルタイプ指定検索
裏技的な方法ですが,google(他の検索エンジンでもそうかもしれない)では,データタイプを指定して検索をかけることができます。通常は pdf, doc, png などを検索するのですが,例:ぐぐれ filetype:pdf
これで,rdf関連のフォーマットで検索してしまうのです。例:癌 filetype:ttl
RDFのフォーマットとしては,他に,rdf, n3, nt, trig, trixがあるので,良かったら,調べてみてください。
もう少し多くのデータを作成して…
さて,今後の検索のために,データが一つだけだとつまらないので4つほどお薬をご用意しました。
@prefix rdfs: <http://www.w3.org/2000/01/rdf-schema#> .
@prefix drugbank: <http://bio2rdf.org/drugbank:> .
@prefix drgb: <http://bio2rdf.org/drugbank_vocabulary:> .
@prefix dbowl: <http://dbpedia.org/ontology/> .
@prefix Medical_condition: <http://schema.org/MedicalCondition/> .
<http://sampledrug.jp/Drug#1>
drgb:name "タクロリムス"@ja;
drgb:product "プログラフ顆粒0.2mg"@ja;
dbowl:drugbank drugbank:DB00864;
Medical_condition:name "移植片対宿主病"@ja;
dbowl:icd10 "T860".
<http://sampledrug.jp/Drug#2>
drgb:name "ホスカルネットナトリウム水和物"@ja;
drgb:product "点滴静注用ホスカビル注24mg/mL"@ja;
Medical_condition:name "サイトメガロウイルス網膜炎"@ja;
dbowl:icd10 "B258","H320";
dbowl:drugbank drugbank:DB00529 .
<http://sampledrug.jp/Drug#3>
drgb:name "塩酸ゲムシタビン"@ja;
drgb:product "ジェムザール注射用200mg"@ja;
Medical_condition:name "膵癌"@ja;
dbowl:icd10 "C259";
dbowl:drugbank drugbank:DB00441 .
<http://sampledrug.jp/Drug#4>
drgb:product "ランプレンカプセル50mg"@ja;
Medical_condition:name "ハンセン病"@ja;
dbowl:icd10 "A309";
drgb:name "クロファジミン"@ja;
dbowl:drugbank drugbank:DB00845 .
RDFを検索!
いやはや,URI談義がこんなに長くなるとは思わなんだ…あの,余分な情報でしたらすっ飛ばしてください。
さて,いよいよ,RDFを検索しましょう!
RDFの検索にはSPARQL という言語を使います。SQLチックに,でも,言葉のようなRDFはそのままに検索することが出来ます。
ブログで紹介できるクエリは限られているので,よりよいクエリや環境構築を学びたい方は,Learning SPARQL という本をご参照ください。残念ながら英語版しか無いのですが,クエリとその実行結果だけでも,読む価値有りです。1st Editionと2nd Editionがありますが,新しい情報も追加されているので,2nd Editionをオススメします。
今回は,こちらに検索窓をご用意しました(一般的にはSPARQL Endpointといいます。)。Let's click!
するとこんな感じの画面が開かれると思います。
何はともあれ,検索実行ボタンを押してみてください。
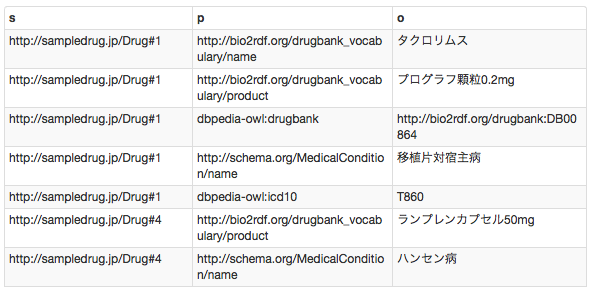
select * where {?s ?p ?o.} LIMIT 100
検索結果
解説しましょうっ。selectは単純に選んでくれといっています。その後の*はワイルドカード,全部選んでくれ!と言っています。次にwhere,どこの何を選ぶのか,以下に指定します。?s ?p ?o.これはあ大変ざっくりしたクエリで,?s ?p ?o という形になるもの全部出せや,と言っています。なので,トリプルすべてを出すということになります。ちなみに,この?s ?p ?o.は自分で指定して良いので,別に?shugo ?jutugo ?mokutekigo.とかにしても構いません。ちなみに,ずぼらな私はselectやwhereを小文字にしてしまっていますが,一般的には大文字を使います。ただ検索上は問題ないので(そのあたりは,SQL系の言語と同じです)ご了承ください。
ちょっと分かりにくい方のために,例えば,以下のクエリを実行してみてください。
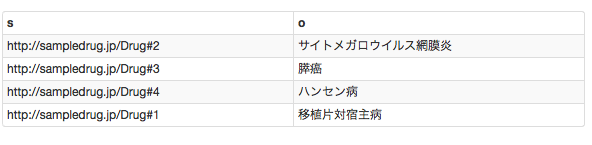
prefix Medical_condition: <http://schema.org/MedicalCondition/>
select * where
{?s Medical_condition:name ?o.}
すると疾患名だけが検索結果に現れるはずです。
もう少し検索してみましょう。
prefix Medical_condition: <http://schema.org/MedicalCondition/>
prefix dbowl: <http://dbpedia.org/ontology/>
prefix drgb: <http://bio2rdf.org/drugbank_vocabulary:>
prefix drugbank: <http://bio2rdf.org/drugbank:>
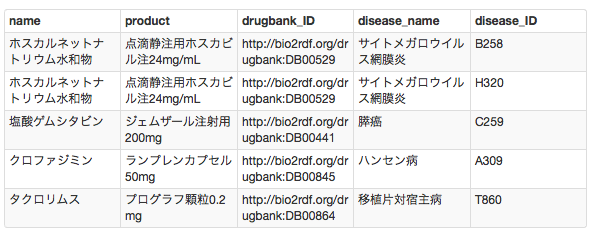
select ?name ?product ?drugbank_ID ?disease_name ?disease_ID
where
{
?s
drgb:name ?name;
drgb:product ?product;
dbowl:drugbank ?drugbank_ID;
Medical_condition:name ?disease_name;
dbowl:icd10 ?disease_ID.
}
?s drgb:name ?name;のように,";"で繋げると,Subject は同じまま,PredicateとObjectを書いていくことが出来ます。
実行結果
え,今まで作ったRDF形式の手持ちのデータを表形式にしただけじゃないか,この…(以下自粛)って?
あ,はい,このままではそうです。次回は,外部のデータと繋げてもっと使える検索をする,
という話をしたいと思います。
ご紹介が遅れましたが,今回使用させて頂いた
http://lodcu.cs.chubu.ac.jp/SparqlEPCU/は中部大学の年岡研究室で提供してくださっているサービスで,手持ちのRDFデータをアップロードすると,SPARQL Endpointという検索窓を提供してくれる太っ腹なサービスです。データ量が少ない場合やちょっと皆さんとデータを共有したい時には,使用してみると大変便利だと思います。
直感RDF!! シリーズ
その1-RDFとは。
その3 -外部のデータと繋げて使える検索をしよう。