はじめに
コンピュータビジョンではホモグラフィ行列(射影変換行列)というのが頻出します。これは、ある画像において、射影変換(拡大・縮小・回転・平行移動など)が行われた場合、元画像の画像座標から変換後の画像に射影することができる3×3の行列です。逆に、2画像間の画像マッチングにおいて、複数の対応点が存在するとき、ホモグラフィ行列を推定することができます。一般に、対応点の座標には誤差があるので、この誤差を最小化するようなホモグラフィ行列は、最小二乗法により推定することができます。
本稿では、具体例により、DLT法を使ってホモグラフィ行列を推定する手順を示すともに、あまりまとまって説明されていることのない特異値分解(SVD)により最小二乗解を求める数学理論について、詳しく説明します。
ホモグラフィ行列
ホモグラフィ行列と、画像座標の射影変換は、以下のように表されます。ホモグラフィ変換(射影変換)は9つの成分を自由に決められますが、今回は回転と平行移動だけのアフィン変換を考えます。
H = \lambda\left(
\begin{array}{ccc}
\cos \theta & -\sin\theta & t_1 \\
\sin\theta & \cos\theta & t_2 \\
0 & 0 & 1
\end{array}
\right)
\\
\vec{x}_1 = (x_1,y_1,1)^T,\ \vec{x}_2 = (x_2,y_2,1)^T \\
\vec{x}_2 = H\vec{x}_1
ここで、画像座標$\vec{x}_1,\vec{x}_2$は同次座標系を用いています。$t_1,t_2$は平行移動を表し、左上の2×2は回転行列です。また、拡大、縮小すなわちスケールによる定数倍の不定性があり、$\lambda$はそれを表す係数です。ホモグラフィ行列の成分は9個ありますが、定数倍の不定性があることから、自由度は8となります。つまり、4点以上の対応点(一つの対応点につきx,yの2つの方程式が立てられる)があればホモグラフィ行列を推定することができます。
例題
元の画像をアフィン変換して2枚の画像を生成します。アフィン変換はopencvの関数を使用しました。対応点は4つ以上で推定ができますが、今回は10点用意します。ホモグラフィ行列によりアフィン変換して座標を求めておきます。
img1
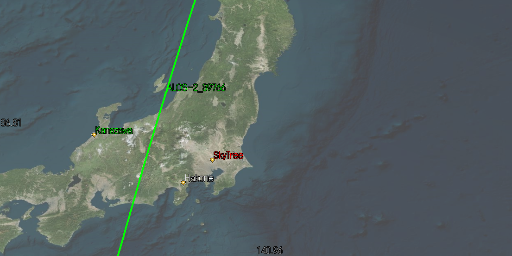
img2
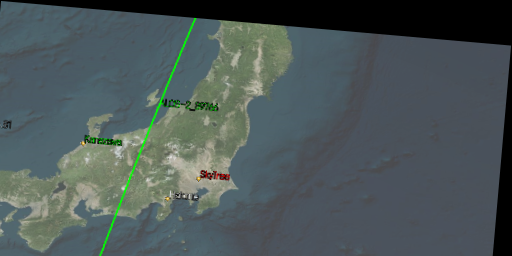
対応点座標
img1 (10, 3)
[[ 66 215 1]
[135 32 1]
[362 185 1]
[479 40 1]
[239 247 1]
[407 147 1]
[253 14 1]
[ 11 103 1]
[474 181 1]
[ 57 222 1]]
img2 (10, 3)
[[ 57 229 1]
[141 53 1]
[354 225 1]
[483 91 1]
[226 276 1]
[402 191 1]
[260 45 1]
[ 11 113 1]
[466 231 1]
[ 47 236 1]]
コード
def main():
os.chdir(PATH_NAME)
src = cv2.imread("./stk.png", cv2.IMREAD_COLOR)
src = cv2.resize(src,(512,256))
cv2.imwrite("src.png", src)
print(src.shape)
# 回転5deg、平行移動(10,10)
theta = np.radians(10)
t1 = 10
t2 = 10
# ホモグラフィ行列
H = np.array([[np.cos(theta),-np.sin(theta),t1]
,[np.sin(theta),np.cos(theta),t2]
,[0,0,1]])
print("Homography matrix=\n" ,H)
# 元画像における画像座標*4 点
img_1 = []
for x in range(4):
v = random.randint(0,src.shape[0])
u = random.randint(0,src.shape[1])
img_1.append([u,v,1])
img_1 = np.array(img_1)
print("img1",img_1.shape)
print(img_1)
# 変換後画像における画像座標N点(射影変換)
img_2 = []
for row in img_1:
x2 = H @ row
x2 = x2.astype('int')
x2[0] = x2[0]
x2[1] = x2[1]
img_2.append(x2)
img_2 = np.array(img_2)
print("img2",img_2.shape)
print(img_2)
# Mは2×3の行列 画像のアフィン変換
M = H[:2,]
affine_img = cv2.warpAffine(src, M, (src.shape[1], src.shape[0]))
cv2.imshow("color", affine_img)
cv2.waitKey(0)
cv2.destroyAllWindows()
cv2.imwrite("affine_img.png", affine_img)
return
ホモグラフィ行列の推定(DLT法)
さて、ここから本題に入っていきます。推定にはDLT法というものを使います。これは、線形方程式の形にして最小二乗法を使える形に式変形するものです。最小二乗法は、$A\boldsymbol{x}=\boldsymbol{b}$で書ける一次連立方程式において、$A\boldsymbol{x}-\boldsymbol{b}=0$の2乗誤差が最小となる$\boldsymbol{x}$を推定します。射影変換の式は
\boldsymbol{x}_2 = H\boldsymbol{x}_1
でした。$A\boldsymbol{x}=\boldsymbol{b}$の形ですが、推定したいのは$x$ではなく、係数行列の$H$です。ここで、射影変換したベクトルは平行であることを使うと、$\boldsymbol{x}_2 \times H\boldsymbol{x}_1 = 0$です。式変形すると、以下のようになります。
\left[
\begin{array}{ccccccccc}
0 & 0 & 0 & -x_1 & -y_1 & -1 & y_2 x_1 & y_2 y_1 & y_2 \\
x_1 & y_1 & 1 & 0 & 0 & 0 & -x_2 x_1 & -x_2 y_1 & -x_2 \\
\end{array}
\right]
\left[
\begin{array}{c}
h_1 \\
h_2 \\
h_3 \\
h_4 \\
h_5 \\
h_6 \\
h_7 \\
h_8 \\
h_9 \\
\end{array}
\right]
=\boldsymbol{0}
ここで、img_1、img_2の対応点の点群を以下にように表します。
\boldsymbol{x}_A=\{\boldsymbol{x}_1^1,\boldsymbol{x}_1^2,\cdots,\boldsymbol{x}_1^k\}\\
\boldsymbol{x}_B=\{\boldsymbol{x}_2^1,\boldsymbol{x}_2^2,\cdots,\boldsymbol{x}_2^k\}
上の式を対応点分拡張すると、
\left[
\begin{array}{ccccccccc}
0 & 0 & 0 & -x_A^1 & -y_A^1 & -1 & y_B^1 x_A^1 & y_B^1 y_A^1 & y_B^1 \\
x_A^1 & y_A^1 & 1 & 0 & 0 & 0 & -x_B^1 x_A^1 & -x_B^1 y_A^1 & -x_B^1 \\
\vdots \\
0 & 0 & 0 & -x_A^k & -y_A^k & -1 & y_B^k x_A^k & y_B^k y_A^k & y_B^k \\
x_A^k & y_A^k & 1 & 0 & 0 & 0 & -x_B^k x_A^k & -x_B^k y_A^k & -x_B^k \\
\end{array}
\right]
\left[
\begin{array}{c}
h_1 \\
h_2 \\
h_3 \\
h_4 \\
h_5 \\
h_6 \\
h_7 \\
h_8 \\
1 \\
\end{array}
\right]
=\boldsymbol{0}
ここで、定数倍の不定性があることから、自由度を減らすため制約条件として$h_9=1$としています。左辺の2k×9の係数行列を$A$とし、ホモグラフィ行列の成分を列ベクトルとして表したものを$\boldsymbol{h}$とすると、$A\boldsymbol{h}=\boldsymbol{0}$となります。これで、最小二乗法を解く準備が整いました。$A\boldsymbol{x}=\boldsymbol{b}$の右辺が0ベクトルの場合に相当します。$A\boldsymbol{h}=\boldsymbol{0}$の誤差を最小化する$\boldsymbol{h}$が求めるホモグラフィ行列です。なお、制約条件は$|\boldsymbol{h}|=0$とする場合もあります。
最小二乗法
さて、最小二乗法の正規方程式は以下のような形でした。
A^{T}A\boldsymbol{x} = A^{T}\boldsymbol{b}
今回、右辺は0となるので、
A^{T}A\boldsymbol{x} = 0
となります。これに対応しています。
さて、
最小二乗法において、
J = \frac{1}{2} \sum_{i=1}^{m}(\sum_{j=1}^{n} a_{ij}x_{j}-b_i)^2 \rightarrow min
の最小値を求めるために偏微分=0とする前に、
J = \frac{1}{2}( x^{T}A^{T}Ax-x^{T}A^{T}b-b^{T}Ax+b^{T}b )\\
となっていました。今回、$\boldsymbol{b=0}$ですので、
J = \frac{1}{2} x^{T}A^{T}Ax
となります。ここで、行列$A^TA$は半正定値対称行列(対称行列であり固有値が0または正)ということが知られており、さらにこのとき、2次形式である$\boldsymbol{x}^T A^TA\boldsymbol{x}$を最小にする$\boldsymbol{x}$は行列$A^TA$の最小固有値に対応する固有ベクトルであるという定理があります。(→線形代数)
今回は、$\boldsymbol{x}$に$\boldsymbol{h}$が対応します。
特異値分解
行列$A^TA$の最小固有値に対応する固有ベクトルは特異値分解(SVD)により求めることができます。$A$の特異値分解は、
A = U\Sigma V^T \\
\Sigma =
\left[
\begin{array}{ccc}
\sigma_1 & & \\
& \sigma_2 & \\
& \cdots & \\
& \ & \sigma_r \\
\end{array}
\right]
です。$U$は$AA^T$の固有ベクトルを正の固有値の大きい順に並べたもの、$V$は$A^TA$の固有ベクトルを正の固有値の大きい順に並べたものとなります。$\sigma_1,\sigma_2,\cdots,\sigma_r$は、特異値と呼ばれ、$A^TA$の固有値の平方根(>=0)となっています。ここで欲しいのは最小二乗解である$A^TA$の最小固有値に対応する固有ベクトルなので、$V$の最終行がそれに対応します。なお、特異値はゼロの場合があります。これは$A$がフルランクでないことを意味しますが、その場合でも$V$の最終行、特異値がゼロに対応する固有ベクトルが最小二乗解となります。つまり、最小固有値を$\lambda_{min}$とすると、
A^TA\boldsymbol{h}=\lambda_{min} \boldsymbol{h}
を満たす解が所望の$\boldsymbol{h}$となります。最終的に$h_{9}$が1になるように全体を除算します。
実験
以下のように、SVDで$H$を推定すると、ほぼ一致していることがわかります。点数が少ないと、4つ以上であれば解はでますが、あまり一致しません。精度よく算出するには画像座標の重心をとり正規化するなど、いろいろテクニックがあるようです。
Homography matrix=
[[ 0.9961947 -0.08715574 10. ]
[ 0.08715574 0.9961947 10. ]
[ 0. 0. 1. ]]
Estimated Homography matrix=
[[ 9.97456897e-01 -8.38633226e-02 8.92359681e+00]
[ 8.68971782e-02 9.98203988e-01 9.29572691e+00]
[ 7.52034139e-07 6.24553736e-06 1.00000000e+00]]
この推定した$H$を使って、画像座標を座標変換してみると、オリジナルの$H$を使った場合とほぼ一致しています。
[382 105 1]
[382 105 1]
コード:
## H行列推定
A = np.zeros((img_1.shape[0]*2,9))
for i, (a, b) in enumerate(zip(img_1,img_2)):
A[i * 2] = np.array([a[0], a[1], 1, 0, 0, 0, -b[0] * a[0], -b[0] * a[1], -b[0]])
A[i * 2 + 1] = np.array([0, 0, 0, a[0], a[1], 1, -b[1] * a[0], -b[1] * a[1], -b[1]])
print(A.shape)
print("A=",A)
print(H.flatten())
print(np.cross(img_2[0],(H @ img_1[0])))
print(A @ H.flatten())
# singular value decomposition
u, s, vh = svd(A)
print('\nSVD result')
print('shape of u, s, vh:', u.shape, s.shape, vh.shape)
print('singular values:', s.round(2))
min = 8
print('minimum eigen vector:', vh[min])
Hest = vh[min].reshape((3,3))
Hest = Hest / Hest[2,2]
print("Estimated Homography matrix=\n" ,Hest)
print((H @ img_1[0] / (H @ img_1[0])[2]).astype('int'))
print((Hest @ img_1[0] / (Hest @ img_1[0])[2]).astype('int'))