はじめに
初めてQiita書いてみます。
基本的にはバイオインフォマティクス中心のトピックスとなると思います。
非エンジニアですので、コードが乱雑なのはお手柔らかに・・・。また、未来の自分へ向けた備忘録的な書き方をしているのでちょっとくどめに見えるかもしれませんが宜しくおねがいします。
基本的にはWindowsとLinuxに**「Python」の「anaconda」を入れ、「jupyter notebook」をメインに使ってます。
今回はモデルデータを用いて 「乳癌患者の生存率を示すカプランマイヤー曲線」 を描きます。
基本的には下記リンクの方「junyasato」さんや「sinhrks」**さんのトレースです。
<参考>
カプランマイヤー曲線について
[Python lifelines で生存分析]
(http://sinhrks.hatenablog.com/entry/2014/11/01/223504)
anacondaにはpythonの** lifelines ライブラリが入っていなかったのでまずインストールします(以下はLinuxの場合)。
・Linuxターミナルのコンソールで次のように打ってlifelines**ライブラリが入っていないことを確認。
conda list
---結果省略---
その後、同じくコンソールで次のようにインストール。
conda install -c conda-forge lifelines
<参考>
lifelinesライブラリ
インストール後、コンソールで jupyter notebook を立ち上げ。
jupyter notebook
これ以降は** jupyter notebook **での作業となります。
Lifelinesライブラリを用いたカプランマイヤー曲線の作成
プログラム環境
import sys
sys.version
'3.6.5 |Anaconda, Inc.| (default, Apr 29 2018, 16:14:56) \n[GCC 7.2.0]'
使用ライブラリ(モデルデータ含む)
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
from lifelines import KaplanMeierFitter
from lifelines.datasets import load_gbsg2 # test data of breast cancer with hormone treatment
from lifelines import CoxPHFitter # Cox model
%matplotlib inline
モデルデータについて
モデルデータとなる乳癌患者の治療データを入手します。
# test data for breast cancer with hormone treatment
df = load_gbsg2()
type(df)
本データはpythonのpandasのDataFrame形式のデータです。
type of "df" data
pandas.core.frame.DataFrame
(一応下記に参考文献を載せてますが読んでません・・・)
|Original paper: Schumacher, M., Basert, G., Bojar, H., et al. “Randomized 2 × 2 trial evaluating hormonal treatment and the duration of chemotherapy in node-positive breast cancer patients.” Journal of Clinical Oncology 12, 2086–2093. (1994) |
|--:|:--|
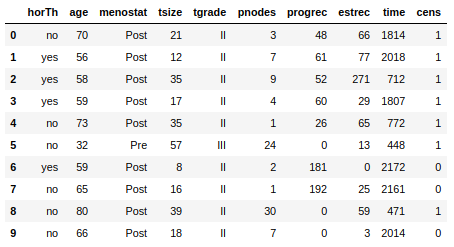
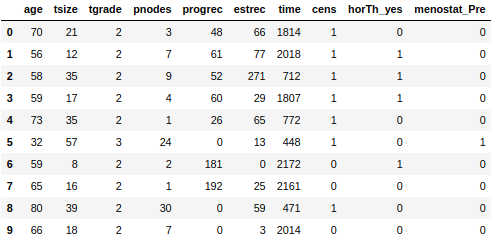
まず、dfデータフレームの最初の10行をざっくり見てみます。
```python
df.head(10)
主なパラメータとして、「horTh」がホルモン治療の有無,
「tgrade」は(恐らく)癌のグレード、timeが生存期間、
「cens」がcensoring、つまりある時点での打ち切りがあったかどうかです。
もう少し詳細にパラメータを見てみると、
print(df.shape)
686症例、10パラメータ。
(686, 10)
```python
print(df.nunique())
例えば「horTh」は2種類,「tgrade」は3種類、「cens」は2種類のuniqueパラメータが存在します。
horTh 2
age 54
menostat 2
tsize 58
tgrade 3
pnodes 30
progrec 242
estrec 244
time 574
cens 2
dtype: int64
df.isnull().all()では各列の要素に欠損があるかを調べます。
boolの返り値が"False"の場合は、「欠損がある、わけではない」、つまり要素に欠損がない、という意味です。
```python
# 欠損値の判定
print(df.isnull().all())
本データには欠損値はないためこのまま進めますが、実施中の臨床データでは欠損値が出てくるケースがあると思います。そのため、実臨床データの解析では、ここから更にデータクリーニングのステップが入ります。
horTh False
age False
menostat False
tsize False
tgrade False
pnodes False
progrec False
estrec False
time False
cens False
dtype: bool
3つのパラメータの内訳。dtype(データ型)もついで調べている理由は後述します。
```python
# パラメータの種類
print("horTh: ", df["horTh"].unique(), df["horTh"].dtype)
print("tgrade: ", df["tgrade"].unique(), df["tgrade"].dtype)
print("cens: ", df["cens"].unique(), df["cens"].dtype)
「horTh」は治療の有無として'no'と'yes'、「tgrade」として'I'、'II'、'III'、「cens」では打ち切りの有無として'1'と'0'、のパラメータが存在することがわかります。
horTh: ['no' 'yes'] object
tgrade: ['II' 'III' 'I'] object
cens: [1 0] int64
どれくらいの生存期間の患者を対象にしているかをざっくり把握します。
```python
# 臨床研究期間における最小と最大のsurvival time
print(" min of viable time: ", df["time"].min())
print(" max of viable time: ", df["time"].max())
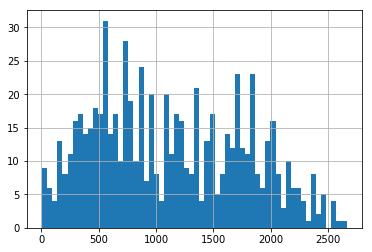
最大7年間の観察期間のようです。
min of viable time: 8
max of viable time: 2659
686人を60分割してsurvival timeのヒストグラムを描くと次のようになります。
```python
df["time"].hist(bins=60)
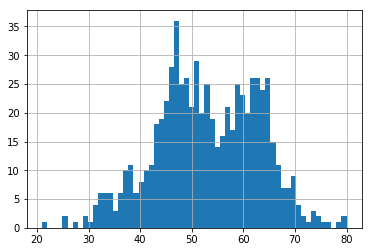
686人を60分割して年齢層のヒストグラムを描くと次のようになります。
df["age"].hist(bins=60)
40代後半から60代前半にかけての患者が多いようですね。
基本的に大半の癌は高齢者の方が罹患率が高い傾向にあるということがここからわかりますね。
50代半ばの患者の方がやや少ないのは何かしら理由があるのでしょうか。
ちなみにこれは1994年のデータセットなので、治療法が改善されている現在では
これとはやや異なるプロファイルになる可能性はあります。
カプランマイヤー曲線の作成前のデータ分割結果の検証
「horTh」がホルモン治療の有無により、生存率が変わるかをカプランマイヤー曲線によりチェックします。
その前に、groupby機能により、どのようにデータを分割できるかを見てみます。
# group classification by with or without hormone therapy("horTh")
for name, group in df.groupby('horTh'):
print("name =", name)
print("group =")
print(group.shape)
print(group.head(10))
print("")
name = no
group =
(440, 10)
horTh age menostat tsize tgrade pnodes progrec estrec time cens
0 no 70 Post 21 II 3 48 66 1814 1
4 no 73 Post 35 II 1 26 65 772 1
5 no 32 Pre 57 III 24 0 13 448 1
7 no 65 Post 16 II 1 192 25 2161 0
8 no 80 Post 39 II 30 0 59 471 1
9 no 66 Post 18 II 7 0 3 2014 0
13 no 50 Post 27 III 1 16 12 1842 0
15 no 54 Post 30 II 1 135 6 1371 1
16 no 39 Pre 35 I 4 79 28 707 1
19 no 55 Post 65 I 4 312 76 865 1
>```python
name = yes
group =
(246, 10)
horTh age menostat tsize tgrade pnodes progrec estrec time cens
1 yes 56 Post 12 II 7 61 77 2018 1
2 yes 58 Post 35 II 9 52 271 712 1
3 yes 59 Post 17 II 4 60 29 1807 1
6 yes 59 Post 8 II 2 181 0 2172 0
10 yes 68 Post 40 II 9 16 20 577 1
11 yes 71 Post 21 II 9 0 0 184 1
12 yes 59 Post 58 II 1 154 101 1840 0
14 yes 70 Post 22 II 3 113 139 1821 0
17 yes 66 Post 23 II 1 112 225 1743 0
18 yes 69 Post 25 I 1 131 196 1781 0
「horTh」の状態(ホルモン治療が'no' or 'yes')によりデータを分割し、"shape"により分割後のデータ数(行×列)を表示させたのち、それぞれデータを10症例ずつ表示させた結果です。
分割した2つのグループ「no」と「yes」の行数の合計が(440 + 246) = 686、とオリジナルデータの行数と一致しているのがわかります。
これをみるとホルモン治療を行っていない対称群の方が症例数が2倍程度多いようですね。数百症例規模の研究だと大きな問題にはならない気がしますが、 症例数が少ない場合は「Stratified Sampling (層化サンプリング) 」等をした方が良いのかも・・・
# tgrade after grouping by with or without hormone therapy("horTh")
for name, group in df.groupby('horTh'):
print("name =", name)
print(group["tgrade"].value_counts())
print("")
name = no
II 281
III 111
I 48
Name: tgrade, dtype: int64
name = yes
II 163
III 50
I 33
Name: tgrade, dtype: int64
ホルモン治療の有無によりグループ分けしたあとの、「tgrade」の3種類のグレードの割合について見てみました。多少ばらつきはあるものまあ許容範囲なのかもしれません。
これが例えば、別治療群のみステージIの比率が圧倒的に多い、とかいう結果になると、そもそも比較が成立しない可能性がありますしね。このあたりはじっくりチェックしておきたいところです。
### カプランマイヤー曲線の作成
いよいよカプランマイヤーまできました。
「horTh」がホルモン治療の有無により、生存率が変わるかをカプランマイヤー曲線によりチェックします。
先ほどgroupby機能により「horTh」によりグループ分けした結果がほどほどにきちんと分割されている(ような気がする)ことを確認したので、この2種類のグループでカプランマイヤー曲線を描写します。
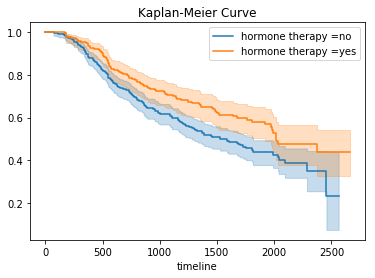
```python
kmf = KaplanMeierFitter()
ax = None
for name, group in df.groupby('horTh'):
kmf.fit(group['time'], event_observed=group['cens'],
label = 'hormone therapy =' + str(name))
# 描画する Axes を指定。None を渡すとエラーになるので場合分け
if ax is None:
ax = kmf.plot()
else:
ax = kmf.plot(ax=ax)
plt.title('Kaplan-Meier Curve')
plt.show()
なんとなく、ホルモン治療群の方が生存率は高そうな感じですね。
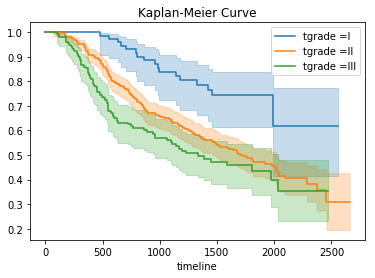
次は、「tgrade」で層別化してカプランマイヤーを描いてみます。
ax = None
for name, group in df.groupby('tgrade'):
kmf.fit(group['time'], event_observed=group['cens'],
label = 'tgrade =' + str(name))
# 描画する Axes を指定。None を渡すとエラーになるので場合分け
if ax is None:
ax = kmf.plot()
else:
ax = kmf.plot(ax=ax)
plt.title('Kaplan-Meier Curve')
plt.show()
こちらもtgrade=IIとIIIの差はやや微妙ですが、Iの予後が良いのは間違いなさそうですね。
ちなみに下記でtgradeで分類したグループI,II,IIIそれぞれの年齢に関する要約統計量を算出していますが、だいたい均等分布していることがわかります。
この2点の情報から、(本検証の結果からは)年齢に関わらず、癌は早期発見するほど予後は良くなるであろう、ということが推測できます。
# age after grouping by ("tgrade")
for name, group in df.groupby('tgrade'):
print("name =", name)
print(group["age"].describe())
print("")
name = I
count 81.000000
mean 54.000000
std 9.648834
min 37.000000
25% 47.000000
50% 51.000000
75% 61.000000
max 79.000000
Name: age, dtype: float64
name = II
count 444.000000
mean 53.364865
std 10.059742
min 21.000000
25% 46.000000
50% 53.000000
75% 62.000000
max 80.000000
Name: age, dtype: float64
name = III
count 161.000000
mean 51.714286
std 10.452529
min 29.000000
25% 45.000000
50% 52.000000
75% 60.000000
max 77.000000
Name: age, dtype: float64
しかし、年齢についてこのままカプランマイヤー曲線を描写するととんでもないことに・・・・
```python
ax = None
for name, group in df.groupby('age'):
kmf.fit(group['time'], event_observed=group['cens'],
label = 'age =' + str(name))
# 描画する Axes を指定。None を渡すとエラーになるので場合分け
if ax is None:
ax = kmf.plot()
else:
ax = kmf.plot(ax=ax)
plt.title('Kaplan-Meier Curve')
plt.show()
この理由はuniqueな年齢ごとにカプランマイヤーが層別化してしまうからです。
前の方で本データセットに関して、「age」には54種類の異なる年齢が登録されていることがわかっていましたので、カプランマイヤーでは54種類の層別化をおこなっている(はず)です。
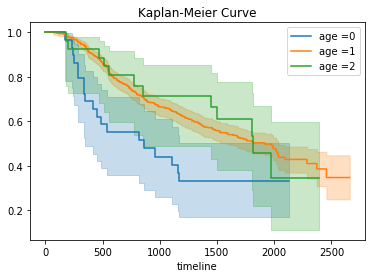
そこで、データセットに関して年齢を35歳区分で区切って、3分割(0〜34,35〜69,70〜)してみます。
3分割した「age」の要素(df2["age"])はそれぞれ(0,1,2)へと変換されていることがわかると思います。
df2 = df.copy()
df2["age"] = (df2["age"]//35) # 35歳ずつ区切る
print(df2["age"].value_counts())
df2.head(10)
1 631
0 29
2 26
Name: age, dtype: int64

```python
# tgrade after grouping by ("age")
for name, group in df2.groupby('age'):
print("name =", name)
print(group["tgrade"].value_counts())
name = 0
II 18
III 11
Name: tgrade, dtype: int64
name = 1
II 409
III 144
I 78
Name: tgrade, dtype: int64
name = 2
II 17
III 6
I 3
Name: tgrade, dtype: int64
しかし分割した年齢区分には厄介な問題が・・・・。
上記で3分割した年齢層ごとにtgradeのサブタイプの数を見てみたのですが、若年層(0〜34歳)のサブタイプにはtgradeがIの患者が含まれていません。つまり、分割後の若年層グループは潜在的に癌が進行している患者が多くを占めていると予想されます。
そのため、年齢3分割後のカプランマイヤーを描写すると・・・
```python
ax = None
for name, group in df2.groupby('age'):
kmf.fit(group['time'], event_observed=group['cens'],
label = 'age =' + str(name))
# 描画する Axes を指定。None を渡すとエラーになるので場合分け
if ax is None:
ax = kmf.plot()
else:
ax = kmf.plot(ax=ax)
plt.title('Kaplan-Meier Curve')
plt.show()
一見、age=0(0〜34歳までの患者)の生存率が悪いような気がするのですが、これは前述したように層別化後に若年層ではtgrade=Iの患者がいない影響もあるためだと考えられます(筆者の勝手な推測です)。
かといって、tgrade=Iの患者を排除して解析すると症例数が少なくなっていきますし、なかなか臨床試験の解析は難しいですね(とりあえずこの解析は年齢に関してはこれ以上深入りしません)。
ここで言いたかったのは、分類項目が多すぎるものに関しては、何らかの方法で分類項目を減らして解析することが可能だということです(その結果が良いか悪いかは別として)。
コックス比例ハザードモデル(Cox proportional hazard model)
上記まででカプランマイヤー曲線を作成して、なんとなくホルモン治療の有無やtgradeの違いによって、患者の生存率(≒予後)に影響を与えることがわかってきましたが、では** 「どの程度」 影響があるのでしょうか?
それを調べる手法の一つが「コックス比例ハザードモデル」**です。
コックス比例ハザードモデルでは、各パラメータが目的変数(今回だと生存率)に与える影響度を数値で比較します(間違ってたらすみません・・・)。
そして、数値で比較することにより、ではこの疾患ではこのパラメータに注目してフォローアップしていこう、という治療方針が立ちます(と思います)。
しかし、ここで元データの要素に少し厄介な性質があることに気づきます。
それは「文字データ」の取り扱いです。
前の方でdtype(データ型)を調べていた時に、いくつかのパラメータは数値型(例:int64)ではなく、object型だったことを覚えているでしょうか?
色々なコマンドにデータを引き渡す場合、結構な頻度でobject型のデータは拒絶されることあります。
horTh: ['no' 'yes'] object
tgrade: ['II' 'III' 'I'] object
cens: [1 0] int64
実際に今回のオリジナルデータをコックス比例ハザードモデルで分析してみると、、、
cph = CoxPHFitter()
cph.fit(df, duration_col='time', event_col='cens', show_progress=True)
TypeError Traceback (most recent call last)
...<中略>...
TypeError: DataFrame contains nonnumeric columns:
['horTh', 'menostat', 'tgrade']. Try 1) using pandas.get_dummies to convert
the non-numeric column(s) to numerical data, 2) using it in stratification
strata=, or 3) dropping the column(s).
でた!素人殺しのエラーメッセージ。。。
心が折れて諦めそうになりますが、いやいや読むとどうも['horTh', 'menostat', 'tgrade']、の3種類が問題なんじゃないかという予想がつきます。
そして、その3種類のデータはobjectデータです。そしてよくよく読むと、Pythonもそれらについて、
1) using pandas.get_dummies to convert the non-numeric column(s) to numerical data
2) using it in stratification `strata=`,
3) dropping the column(s).
をTryしろよ、って言っていることに気づきます。でもこういうのって、エラー出た直後は冷静に読めないよね。
手順としては、
A) ['horTh', 'menostat', 'tgrade']の3種類のobject型データに関して、「get_dummies」を使用して、要素の種類に関してカテゴリ変数から数値型(→ダミー変数(例:0、1))に変換します。
通常、get_dummies()関数ではk個の要素がk個のダミー変数に変換されるため、引数でdrop_first=Trueし、k-1個のダミー変数に変換するようにします。
k-1個にする理由は下記リンク参照して頂ければと思います。
><参考>
>[Pandasでダミー変数を作成するget_dummies関数の使い方]
(https://deepage.net/features/pandas-get-dummies.html)
ただし、**「junyasato」**さんのブログでは、['horTh', 'menostat']に関しては、ダミー変数化しているのですが ['tgrade']に関しては列ごと削除してCox比例ハザードモデルに投入しています。
その理由はいくつかある気がするのですが、理由の一つは['tgrade']の要素の種類としては「I, II, III」の3種類があり、そのままダミー変数化してしまうと、オリジナルデータでは「'tgrade'」が1列だったのが2列になってしまうためだと思います。
ごちゃごちゃ書いていますが実際のデータを見たほうが早いので、実際のデータに関して変換前後の10行のデータを見てみましょう。
```python
df3_pre = df.copy()
df3_pre2 = pd.get_dummies(df3_pre,columns=['horTh','menostat','tgrade'],drop_first=True)
# ダミーデータ変換前
df3_pre.head(10)
# ダミーデータ変換後
df3_pre2.head(10)
df3_pre #ダミーデータ変換前
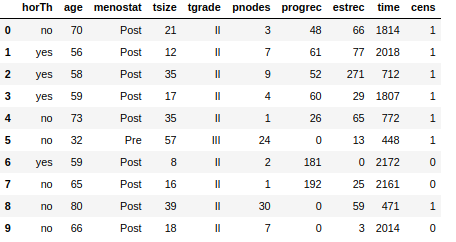
df3_pre2 #ダミーデータ変換後
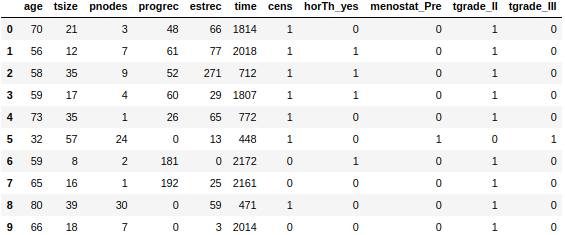
これを見ると、例えば「horTh」に関しては、ホルモン治療があった人(='yes')の人が「1」、治療がなかった人が「0」になっていることがわかります。
また、「menostat」に関しては、'Pre'が「1」、'Post'が「0」になっていることがわかります。
一方、「tgrade」に関しては、列が「tgrade_II」,[tgrade_III]の2列にわかれています。
そして、元々のtgrade='II'の人は、「tgrade_II,tgrade_III:1,0],元々のtgrade='III'の人は、「tgrade_II,tgrade_III:0,1],そしてここには表示していませんが、元々のtgrade='I'の人は、「tgrade_II,tgrade_III:0,0]に変換されることになります。
ただこう変換すると、Cox比例ハザードモデルは通常、列データごとの影響度を調べることになるため、恐らくですが、「tgrade_II」の列ではtgrade='II'の人と、'I'+'III'の人の比較、「tgrade_III」の列ではtgrade='III'の人と、'I'+'II'の人の比較をすることになります。
ブログを書きながら、まあでもそれぞれtgradeの影響度を判別できるからこれはこれで良いのかも・・・、と思ったのですが(汗)、元々の自分のポリシー(謎)は「tgrade」という変数そのものを評価したいということでした。
<参考:この状態でCOX比例ハザードモデルに投入した結果>
cph = CoxPHFitter()
cph.fit(df3_pre2, duration_col='time', event_col='cens', show_progress=True)
cph.print_summary()
cph.plot()
duration col = 'time'
event col = 'cens'
number of subjects = 686
number of events = 299
log-likelihood = -1735.73
time fit was run = 2019-05-12 08:25:34 UTC
coef exp(coef) se(coef) z p -log2(p) lower 0.95 upper 0.95
age -0.01 0.99 0.01 -1.02 0.31 1.69 -0.03 0.01
tsize 0.01 1.01 0.00 1.98 0.05 4.39 0.00 0.02
pnodes 0.05 1.05 0.01 6.55 <0.005 34.03 0.03 0.06
progrec -0.00 1.00 0.00 -3.87 <0.005 13.14 -0.00 -0.00
estrec 0.00 1.00 0.00 0.44 0.66 0.60 -0.00 0.00
horTh_yes -0.35 0.71 0.13 -2.68 0.01 7.10 -0.60 -0.09
menostat_Pre -0.26 0.77 0.18 -1.41 0.16 2.65 -0.62 0.10
tgrade_II 0.64 1.89 0.25 2.55 0.01 6.55 0.15 1.12
tgrade_III 0.78 2.18 0.27 2.90 <0.005 8.08 0.25 1.31
Concordance = 0.69
Log-likelihood ratio test = 104.75 on 9 df, -log2(p)=59.01

そこで、元々の自分のポリシーに従うため、「tgrade」の要素「'I','II','III'」をダミー変数ではなく、数値型「'1','2','3'」に変換します。
```python
df3 = df.copy()
df3["tgrade"] = df3["tgrade"].replace({'I': 1,'II': 2,'III': 3})
df3_2 = pd.get_dummies(df3,columns=['horTh','menostat'],drop_first=True)
df3_2.head(10)
[tgrade]がobject型から数値型に変換されているのが確認できます。
それではこの状態でこの状態でCOX比例ハザードモデルに投入してみましょう。
cph.fit(df3_2, duration_col='time', event_col='cens', show_progress=True)
cph.print_summary()
cph.plot()
coef exp(coef) se(coef) z p -log2(p) lower 0.95 upper 0.95
age -0.01 0.99 0.01 -1.01 0.31 1.68 -0.03 0.01
tsize 0.01 1.01 0.00 1.95 0.05 4.30 -0.00 0.02
tgrade 0.28 1.32 0.11 2.64 0.01 6.93 0.07 0.49
pnodes 0.05 1.05 0.01 6.73 <0.005 35.82 0.04 0.06
progrec -0.00 1.00 0.00 -3.89 <0.005 13.26 -0.00 -0.00
estrec 0.00 1.00 0.00 0.37 0.71 0.50 -0.00 0.00
horTh_yes -0.34 0.71 0.13 -2.61 0.01 6.81 -0.59 -0.08
menostat_Pre -0.27 0.77 0.18 -1.46 0.14 2.79 -0.63 0.09
Concordance = 0.69
Log-likelihood ratio test = 101.86 on 8 df, -log2(p)=58.96

めでたく「tgrade」が一つの指標として評価されましたね!
ただ、データをよく見ると、「age」の項はそんなにCOXモデルで生存率に影響を与える指標としては評価されていません。
そのため、前述したように「35歳区分」で「age」を分けて、再び評価してみましょう。
```python
df3_3 = df3_2.copy()
df3_3["age"] = (df3_3["age"]//35) # 35歳ずつ区切る
cph.fit(df3_3, duration_col='time', event_col='cens', show_progress=True)
cph.print_summary()
cph.plot()
coef exp(coef) se(coef) z p -log2(p) lower 0.95 upper 0.95
age -0.35 0.70 0.21 -1.66 0.10 3.37 -0.77 0.06
tsize 0.01 1.01 0.00 2.09 0.04 4.77 0.00 0.02
tgrade 0.28 1.32 0.11 2.60 0.01 6.73 0.07 0.48
pnodes 0.05 1.05 0.01 6.62 <0.005 34.69 0.03 0.06
progrec -0.00 1.00 0.00 -3.88 <0.005 13.25 -0.00 -0.00
estrec 0.00 1.00 0.00 0.45 0.65 0.61 -0.00 0.00
horTh_yes -0.34 0.71 0.13 -2.67 0.01 7.04 -0.60 -0.09
menostat_Pre -0.19 0.83 0.13 -1.45 0.15 2.76 -0.44 0.07
Concordance = 0.69
Log-likelihood ratio test = 103.58 on 8 df, -log2(p)=60.14

プロファイルが大分かわりましたね!
まあ、いずれのグラフでもlog(HR)(95% CI)をまたいでいるので、「age」は影響度がそんなに大きいパラメータではなさそうですが、これを見るともうちょっと層別化して解析してみようかな、という気にもなりますね。
このように、今回は同一データから出発しても、データの区切り方を変えるだけで、全く別の結果が得られる、ということを確認できたためよいモデル実験となりました。それにしてもやはり臨床データの解析はなかなか難しいですね。。。
ちなみに、cox比例ハザードモデルは仮定のもとに成立しているようなので、本来は仮定が成立しているか検証すべきようですがちょっとそこまでフォローできていませんので悪しからず・・・