NetworkXは、グラフ/ネットワーク理論系の計算を行うためのPythonのパッケージです。
最近(2017-09-21)、メジャーアップデートしてバージョン2.0が公開されました。
NetworkX 2.0は1.x系からAPIが大きく整理されています。
これを機に使い方をまとめようと思います。
インストール
pipでインストールしてください。
pip install networkx
古いバージョンをインストール済みなら-Uオプションで更新できます。
pip install -U networkx
グラフの構築
networkx.Graphクラスおよびその派生クラスに頂点と辺を追加していきます。
下の例ではintを頂点としていますが、hashableならなんでも頂点として使えます。
import networkx as nx
G = nx.DiGraph() # 有向グラフ (Directed Graph)
# 頂点の追加
G.add_node(1)
G.add_nodes_from([3, 4, 5])
# 辺の追加 (頂点も必要に応じて追加されます)
G.add_edge(1, 2)
G.add_edges_from([(1, 3), (2, 5), (3, 4), (4, 5)])
# 辺の削除
G.remove_edge(3, 4)
G.remove_edges_from([(1, 3), (2, 5)])
# 頂点の削除 (削除された頂点に接続されている辺も削除されます)
G.remove_node(5)
G.remove_nodes_from([3, 4])
# 指定したパス上の頂点と辺を追加
nx.add_path(G, [1, 2, 3, 4, 5]) # 1 → 2 → 3 → 4 → 5
# 指定した閉路上の頂点と辺を追加
nx.add_cycle(G, [1, 2, 3, 4, 5]) # 1 → 2 → 3 → 4 → 5 → 1
# 放射状に頂点と辺を追加
nx.add_star(G, [1, 2, 3, 4, 5]) # 1 → 2, 1 → 3, 1 → 4, 1 → 5
グラフの可視化
グラフの可視化には、Matplotlibを使う方法とPyGraphvizを使う方法があります。
設定可能な項目が多いのでこの記事では軽い説明に留めます。
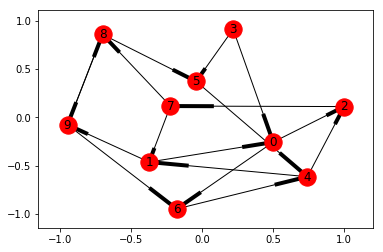
Matplotlibを使う
Matplotlibを使う方法の利点は、インストールが簡単なことです。
欠点は曲線的な辺を描画してくれないことで、複雑なグラフになると頂点や辺やラベルが盛大に重なります。長いラベルをつけるのには向いていません。
import matplotlib.pyplot as plt
import networkx as nx
G = nx.DiGraph()
nx.add_path(G, [3, 5, 4, 1, 0, 2, 7, 8, 9, 6])
nx.add_path(G, [3, 0, 6, 4, 2, 7, 1, 9, 8, 5])
nx.draw_networkx(G)
plt.show()
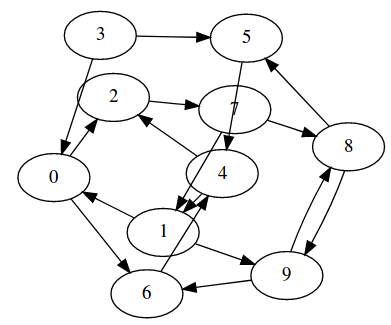
PyGraphvizを使う
PyGraphvizを使うと、Matplotlibを使うよりも気の利いた見た目になります。
欠点は、WindowsにPyGraphvizをインストールするのが多少面倒なことです。
Ubuntuなら apt install graphviz libgraphviz-dev して pip install pygraphviz でOK。
インストール手順(Windows)
- Graphvizをインストールする。( http://wingraphviz.sourceforge.net/wingraphviz/ )
- Githubで公開されているビルド済みwheelを使ってPyGraphvizをインストールする。
# Python 3.6
pip install "https://github.com/CristiFati/Prebuilt-Binaries/blob/master/PyGraphviz/v1.5/Graphviz-2.42.2/pygraphviz-1.5-cp36-cp36m-win_amd64.whl?raw=true"
# Python 3.7
pip install "https://github.com/CristiFati/Prebuilt-Binaries/blob/master/PyGraphviz/v1.5/Graphviz-2.42.2/pygraphviz-1.5-cp37-cp37m-win_amd64.whl?raw=true"
# Python 3.8
pip install "https://github.com/CristiFati/Prebuilt-Binaries/blob/master/PyGraphviz/v1.5/Graphviz-2.42.2/pygraphviz-1.5-cp38-cp38-win_amd64.whl?raw=true"
(参考:Installing pygraphviz on Windows 10 64-bit, Python 3.6)
使い方
import networkx as nx
G = nx.DiGraph()
nx.add_path(G, [3, 5, 4, 1, 0, 2, 7, 8, 9, 6])
nx.add_path(G, [3, 0, 6, 4, 2, 7, 1, 9, 8, 5])
nx.nx_agraph.view_pygraphviz(G, prog='fdp') # pygraphvizが必要

Jupyter Notebookを使っている場合は、SVGで表示することができます。
import networkx as nx
from IPython.display import SVG, display
G = nx.DiGraph()
nx.add_path(G, [3, 5, 4, 1, 0, 2, 7, 8, 9, 6])
nx.add_path(G, [3, 0, 6, 4, 2, 7, 1, 9, 8, 5])
svg = SVG(nx.nx_agraph.to_agraph(G).draw(prog='fdp', format='svg'))
display(svg)
基本的な情報の取得
この辺は1.xから変更が多いようです。
import matplotlib.pyplot as plt
import networkx as nx
G = nx.DiGraph()
nx.add_cyle(G, [1, 2, 3, 4, 5])
nx.add_star(G, [1, 2, 3, 4, 5])
# 頂点の一覧
print(list(G.nodes))
# [1, 2, 3, 4, 5]
# 辺の一覧
print(list(G.edges))
# [(1, 2), (1, 3), (1, 4), (1, 5), (2, 3), (3, 4), (4, 5), (5, 1)]
# 指定した始点に対する、終点の一覧と辺の一覧
print(list(G.succ[1]), G.out_edges(1))
# [2, 3, 4, 5] [(1, 2), (1, 3), (1, 4), (1, 5)]
# 指定した終点に対する、始点の一覧と辺の一覧
print(list(G.pred[5]), G.in_edges(5))
# [4, 1] [(4, 5), (1, 5)]
# 指定した頂点に対する、隣接している頂点の数とその一覧
print(G.degree(4), list(nx.all_neighbors(G, 4)))
# 3, [3, 1, 5]
頂点や辺にデータをもたせる
G.nodesとG.edgesは、頂点および辺をキー、その属性値の辞書を値とした、Mapping (辞書っぽいオブジェクト) になっています。
さらにG.succはネストしたMappingになっており、辺の始点→終点→属性辞書の順に辿ることができます。同様にG.predは辺の終点→始点→属性辞書の順に辿ることができます。
import matplotlib.pyplot as plt
import networkx as nx
G = nx.DiGraph()
G.add_edges_from([(1, 2), (1, 3), (2, 3)])
# G.nodes[頂点][属性キー] = 属性値
G.nodes[1]['a'] = 'Alice'
# G.edges[辺][属性キー] = 属性値
G.edges[1, 2]['b'] = 'Bob'
# G.succ[始点][終点][属性キー] = 属性値
G.succ[2][3]['c'] = 'Carol'
# G.pred[終点][始点][属性キー] = 属性値
G.pred[3][1]['d'] = 'Dave'
print(dict(G.nodes))
# {1: {'a': 'Alice'}, 2: {}, 3: {}}
print(dict(G.edges))
# {(1, 2): {'b': 'Bob'}, (1, 3): {'d': 'Dave'}, (2, 3): {'c': 'Carol'}}
print(G.succ)
# {1: {2: {'b': 'Bob'}, 3: {'d': 'Dave'}}, 2: {3: {'c': 'Carol'}}, 3: {}}
print(G.pred)
# {1: {2: {'b': 'Bob'}, 3: {'d': 'Dave'}}, 2: {3: {'c': 'Carol'}}, 3: {}}
nx.set_node_attributes()やnx.get_edge_attributes()を使うと属性値をまとめて取得したり設定したりできます。
import networkx as nx
G = nx.DiGraph()
G.add_edges_from([(1, 2), (1, 3), (2, 3)])
# すべての頂点に同じ属性値を設定する.
nx.set_node_attributes(G, name='a', values='Alice')
# 頂点ごとに属性値を個別に設定する. キーは共通.
nx.set_node_attributes(G, name='b', values={2: 'Bob', 3: 'Bravo'})
# まるごと渡す.
nx.set_node_attributes(G, values={1: {'c': 'Carol'}, 2: {'d': 'Dave'}})
print(nx.get_node_attributes(G, 'b'))
# {2: 'Bob', 3: 'Bravo'}
print(nx.get_node_attributes(G, 'c'))
# {2: 'Carol'}
print(dict(G.nodes))
# {1: {'a': 'Alice', 'c': 'Carol'}, 2: {'a': 'Alice', 'b': 'Bob', 'd': 'Dave'}, 3: {'a': 'Alice', 'b': 'Bravo'}}
探索系の関数
import matplotlib.pyplot as plt
import networkx as nx
G = nx.DiGraph()
nx.add_cyle(G, [1, 2, 3, 4, 5])
nx.add_star(G, [1, 2, 3, 4, 5])
# 2点間の最短経路
print(nx.shortest_path(G, source=2, target=1))
# [2, 3, 4, 5, 1]
# 指定した点から各点への最短経路
print(nx.shortest_path(G, source=2))
# {1: [2, 3, 4, 5, 1], 2: [2], 3: [2, 3], 4: [2, 3, 4], 5: [2, 3, 4, 5]}
# 各点から指定した点への最短経路
print(nx.shortest_path(G, target=1))
# {1: [1], 2: [2, 3, 4, 5, 1], 3: [3, 4, 5, 1], 4: [4, 5, 1], 5: [5, 1]}
# 各点から各点への最短経路
print(nx.shortest_path(G))
# ...
# ゴール地点までの最短経路長が、ある長さ未満に収まるスタート地点とその経路
print(nx.single_target_shortest_path(G, target=1, cutoff=2))
# {1: [1], 4: [4, 5, 1], 5: [5, 1]}
# スタート地点からの最短経路長が、ある長さ未満に収まるゴール地点とその経路
print(nx.single_source_shortest_path(G, source=3, cutoff=2))
# {3: [3], 4: [3, 4], 5: [3, 4, 5]}
# スタート地点からゴール地点までのすべての単純路 (同じ頂点を通らないパス)
print(list(nx.all_simple_paths(G, source=1, target=5)))
# [[1, 2, 3, 4, 5], [1, 3, 4, 5], [1, 4, 5], [1, 5]]
さいごに
他にも1.xからの変更点がいろいろとあるようです。
暇を見つけてちょこちょこ更新していこうと思います。