この記事は Stan Advent Calendar 2017 17日目の記事です。
はじめに
モデルを立ててパラメータを推定した後で,事後分布から乱数を生成し,将来のデータと観測データの整合性を確認する,事後予測チェックを行うことが推奨されています。立てたモデルから生成したデータが,実際に得られた観測データと整合していたとしたら,そのモデルは現象をとらえていると考えることが出来るからです。
本記事では,視覚的に事後予測チェックを行う方法の一つを紹介します。
モデル
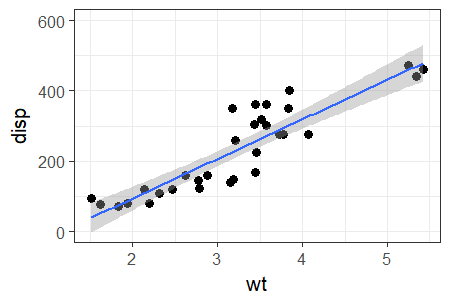
種々の車両に関するデータセットである,mtcarsを使用して例示します。車の排気量(displ)を重量(wt)で予測してみましょう。重い車両ほど排気量が増えると考えられます。実際,データはほぼ一直線上に乗っているように見えます。
この単回帰モデルをStanで書くと以下の通りとなります(StanとRでベイズ統計モデリング7章のコードを使用させていただきました)。generated quantitiesブロック内で,事後分布から乱数を生成しています。
data {
int N;
real X[N];
real Y[N];
int N_new;
real X_new[N_new];
}
parameters {
real a;
real b;
real<lower=0> sigma;
}
model {
for (n in 1:N)
Y[n] ~ normal(a + b*X[n], sigma);
}
generated quantities {
real y_new[N_new];
for (n in 1:N_new)
y_new[n] = normal_rng(a + b*X_new[n], sigma);
}
Rのコードは以下の通りです。
X軸に割り当てる重量(wt)の下限より少し下から,上限より少し上の範囲まで,0.1刻みで新しいX(X_new)を作っています。これらをモデルに投入して生み出される新しいY(y_new)は,今回の観測データと整合するのでしょうか。
library(rstan)
stanmodel <- rstan::stan_model("stan_advent2017.stan")
X_new <- seq(from = min(mtcars$wt) - 1,
to = max(mtcars$wt) + 1,
by = 0.1)
data <- list(N = nrow(mtcars),
Y = mtcars$disp,
X = mtcars$wt,
X_new = X_new,
N_new = length(X_new))
fit <- rstan::sampling(object = stanmodel,
data = data,
seed = 1234)
ggfanパッケージを用いて事後予測チェックを行う
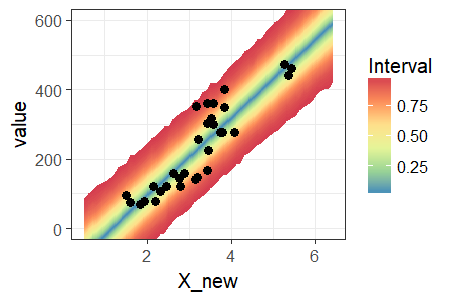
ggplot2パッケージのgeom_ribbon()などを用いて,冒頭のグラフに予測区間を重畳してもいいのですが,少しだけ手間がかかります。そこでggfanパッケージを使用すると簡便に描画することが出来ます。ggfanパッケージの詳しい使用方法については,こちらのページをご参照ください。
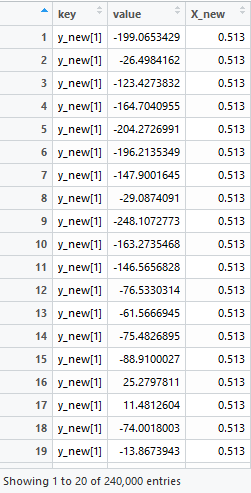
まずは生成された乱数を,ロング形式に並び替えましょう。
library(tidyverse)
temp <- as.data.frame(fit) %>%
dplyr::select(starts_with("y_new")) %>%
tidyr::gather(key = key, value = value) %>%
dplyr::mutate(X_new = rep(X_new, each = 4000))
今回は,新しいX(X_new)の長さは60でした。MCMCのサンプリングにおいて,iterとwarmupは特に指定しなかったので,デフォルトの設定になります(iter = 2000, warmup = 1000, chain = 4)。すなわち各X_newの値について,4,000個のy_newが生成されるわけですから,ロング形式に並べると60 * 4,000 = 240,000行になります。
あとはggplot()に渡してやり,ggfanパッケージのgeom_fan()を使用するだけです。geom_ribbon()は必要ありません。
library(ggfan)
ggplot()+
theme_bw(base_size = 15)+
geom_fan(data = temp, aes(y = value, x = X_new))+
geom_point(data = mtcars, aes(y = disp, x = wt), size = 3)+
scale_fill_distiller(palette = "Spectral")+
coord_cartesian(ylim = c(0, 600))
簡単に予測区間を描画できました。scale_fill_distiller()以降は見栄えを調整しているだけなので,必須ではありません。
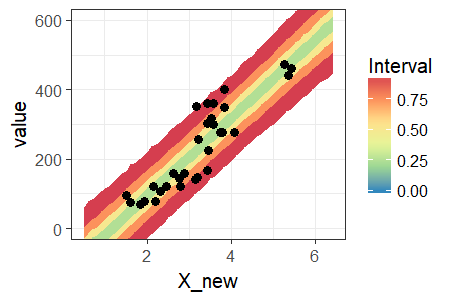
もちろん,特定の分位数を指定することも可能です。0%, 25%, 50%, 75%, 95%の区間を明示してみましょう。
ggplot()+
theme_bw(base_size = 15)+
geom_fan(data = temp, aes(y = value, x = X_new), intervals = c(0, 0.25, 0.5, 0.75, 0.95))+
geom_point(data = mtcars, aes(y = disp, x = wt), size = 3)+
scale_fill_distiller(palette = "Spectral")+
coord_cartesian(ylim = c(0, 600))
一部のデータが95%予測区間をはみ出ているので,まだモデルに改良の余地があるのかもしれませんが,今回立てた単回帰モデルはそんなに悪くなさそうです。
おわりに
以上のように,事後予測区間を可視化することで,モデルを改善する必要性があるかどうかを,直感的に把握することが出来ます。ggfanパッケージはその一助となるかもしれません。もっとも,単回帰以外の時はどう使うのかなあ...