処理速度が遅い・・・
以前の記事にした密度を色で表す散布図において、処理速度が遅いという問題があった。原因はカーネル密度推定の演算に時間がかかること。Rで行う場合はdensCols関数を用いるが、これがPythonでやるよりも圧倒的に速い(しかも標準搭載だから手軽)。
Pythonでも高速でプロットする方法を調べた。
Pythonでの実装方法
カーネル密度推定の高速化方法
仕組み
高速化のカギは「ビン化」、「2D高速フーリエ変換」らしい。
ビン化概算
2Dカーネル密度の正確な推定値の近似により密度計算を高速化します。最初の2Dビン化が(x、y)ポイントで実行され、ビンカウントの行列を取得します。 次に、2D高速フーリエ変換を使用して、各グリッドの密度値を計算するための離散畳み込みを実行します。密度値の4乗根は、密度スケールをカラースケールにマッピングするために計算されます。
https://www.originlab.com/doc/ja/Origin-Help/Create-2D-Kernel-Density
上記サイトに図があるが、2Dビン化ではデータを格子状に分割してマス目ごとのデータ数を数えるイメージ。百聞は一見にしかず。
RのdensCols関数の場合
densColsの公式ドキュメントによると、パッケージKernSmoothのbkde2D関数を用いているらしい。
densColscomputes and returns the set of colors that will be used in plotting, callingbkde2D(*, bandwidth, gridsize = nbin, ..)from package KernSmooth.
次に、bkde2Dの公式ドキュメントを見ると、ビン化(Linear binning)と高速フーリエ変換(Fast Fourier Transform)を用いているらしい。
This is the binned approximation to the 2D kernel density estimate. Linear binning is used to obtain the bin counts and the Fast Fourier Transform is used to perform the discrete convolutions.
ついでに、パッケージKernSmoothの公式ドキュメント(PDF版)とソースコード。
Pythonでの実装方法
ビン化と高速フーリエ変換FFTによって実装できそうだが、難しそうなので先人が作ったものを紹介する。
1. kern_smoothを用いる
ソースコードを見ると、Rのbkde2DをPythonで書き換えているみたい。
- 使い勝手が良い
- 処理速度は
fastkde(後述)より遅い
インストール
pip install kern-smooth
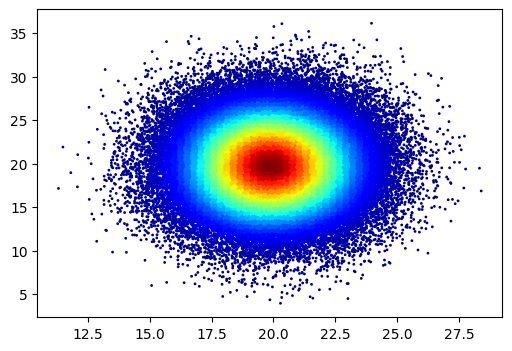
インポート、乱数データの生成
from matplotlib import pyplot as plt
import numpy as np
np.random.seed(0)
# create two 'bulbs' with normal distributions
mean1 = [0, 0]
cov1 = [[5, 0], [0, 30]] # diagonal covariance
x1, y1 = np.random.multivariate_normal(mean1, cov1, 50000).T
mean2 = [5, 17]
cov2 = [[30, 0], [0, 5]] # diagonal covariance
x2, y2 = np.random.multivariate_normal(mean2, cov2, 50000).T
x = np.hstack([x1,x2])
y = np.hstack([y1,y2])
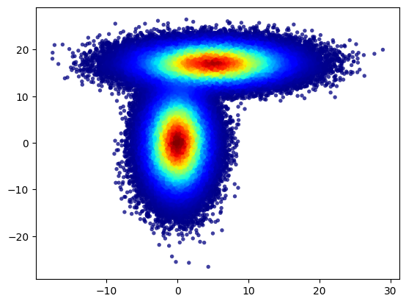
kern_smooth densColsの実行
推定した密度値が返される。
from kern_smooth import densCols
densities = densCols(x, y, nbin = 128)
散布図のプロット
c=densitiesを指定してプロットする。
plt.figure(figsize=(6, 4))
plt.scatter(x, y, c=densities, s=15, edgecolors='none', alpha=0.75, cmap='jet')
plt.colorbar()
plt.show()
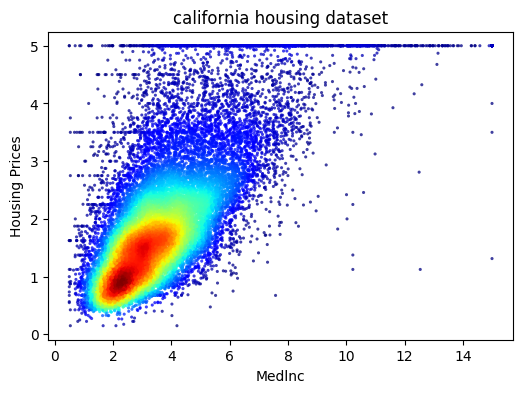
california housing を用いた実装例
2. fastkdeを用いる
- 処理速度が非常に速い。
- 図を描画するためのメソッドが用意されている。
- ほかにも色々な機能がある。
- けど、算出された密度値を散布図に適用するのには向かない(理由は後述)。
機能例
https://github.com/LBL-EESA/fastkde/blob/main/examples/readme_test.ipynb
https://github.com/LBL-EESA/fastkde/tree/main/examples
インストール
!pip install fastkde
インポート、乱数データの生成
import numpy as np
import fastkde
import matplotlib.pyplot as plt
from sklearn.datasets import fetch_california_housing
california_data = fetch_california_housing()
x = california_data.data[:,0]
y = california_data.target
fastkdeの実行
ビン化した際のエッジ(境界)や推定された密度値が返される。あとでこれらを取り出すのだが、この時の出力される配列の形状shapeが一定でない(自動で調整されている?)ため、後の操作でエラーになりがち。
#Do the self-consistent density estimate
PDF = fastkde.pdf(x,
y,
var_names = ['x', 'y'],
num_points=[129, 129],
ecf_precision=1)
密度の値の取り出し
edges_x: x軸方向のビンのエッジ(境界)
edges_y: y軸方向のビンのエッジ(境界)
[x_loc, y_loc]: ビン化後において、元データの各プロットがどのビンに含まれるかの位置
density: 各プロットに対応する密度値
edges_x = np.array(PDF.coords['x'])
edges_y = np.array(PDF.coords['y'])
x_loc = np.digitize(x, edges_x, right=True)
x_loc = np.where(x_loc==edges_x.shape[0], edges_x.shape[0] -1 , x_loc)
y_loc = np.digitize(y, edges_y, right=True)
y_loc = np.where(y_loc==edges_y.shape[0], edges_y.shape[0] -1, y_loc)
density = PDF.data[y_loc, x_loc]
散布図のプロット
# scatterplots colored based on density
plt.figure(figsize=(6, 4))
plt.scatter(x, y, c=density, s=1, marker='o', cmap='jet', alpha=1)
#ラベルの表示
plt.title('california housing dataset')
plt.xlabel('Medlnc')
plt.ylabel('Housing Prices')
# プロットを表示
plt.show()
california housing を用いた実装例
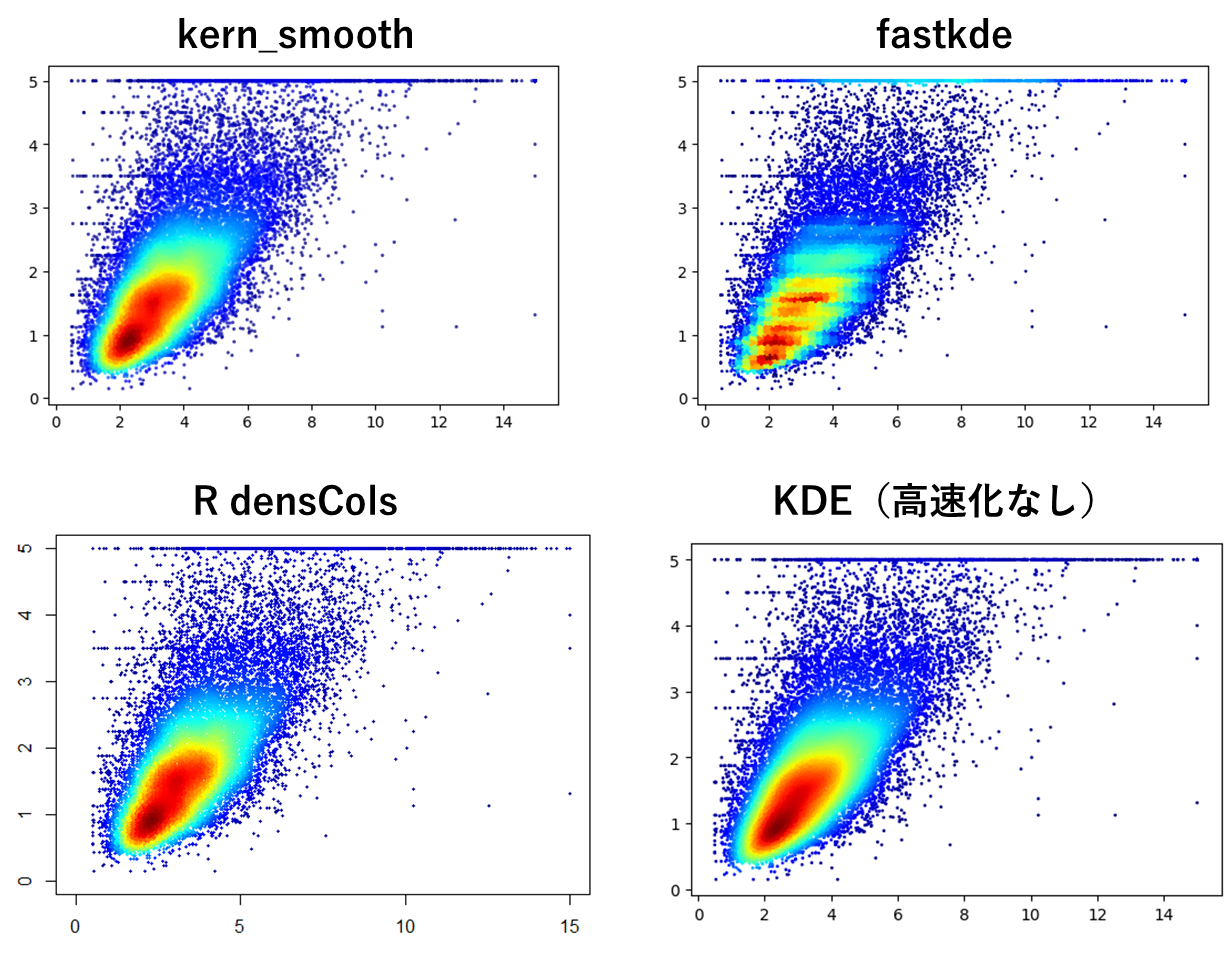
比較
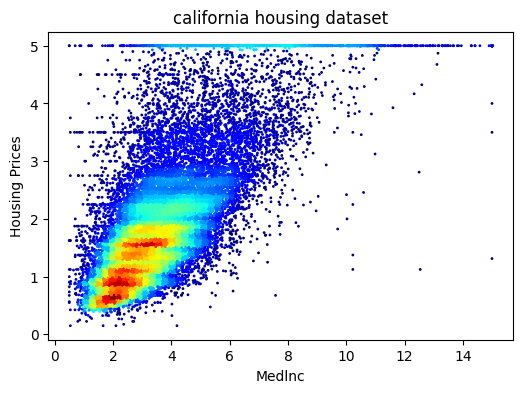
各手法によるプロットの比較は以下の通り。
ker_smoothは良い感じ。
fastkdeはイマイチ。
もちろん高速化しないのが一番きれい。
でもやっぱり総合的にはRdensColsが優秀。
実装コード
kern_smooth : california housing
import numpy as np
import matplotlib.pyplot as plt
from kern_smooth import densCols
from sklearn.datasets import fetch_california_housing
# california housing データの読み込み
california_data = fetch_california_housing()
x = california_data.data[:,0] #平均収入のデータをxに入れる。
y = california_data.target #住宅価格は.targetに入っている。yに入れる。
#密度の計算を実行
densities = densCols(x, y, nbin = 128)
#プロットの描画
plt.figure(figsize=(6, 4))
plt.scatter(x, y, c=densities, s=5, edgecolors='none', alpha=0.75, cmap='jet')
#ラベルの表示
plt.title('california housing dataset')
plt.xlabel('Medlnc')
plt.ylabel('Housing Prices')
# プロットを表示
plt.show()
fastkde: california housing
import numpy as np
import fastkde
import matplotlib.pyplot as plt
from sklearn.datasets import fetch_california_housing
california_data = fetch_california_housing()
x = california_data.data[:,0]
y = california_data.target
#Do the self-consistent density estimate
PDF = fastkde.pdf(x, y, var_names = ['x', 'y'], num_points=[129, 129], ecf_precision=1, do_save_marginals=True)
edges_x = np.array(PDF.coords['x'])
edges_y = np.array(PDF.coords['y'])
x_loc = np.digitize(x, edges_x, right=True)
x_loc = np.where(x_loc==edges_x.shape[0], edges_x.shape[0] -1 , x_loc)
y_loc = np.digitize(y, edges_y, right=True)
y_loc = np.where(y_loc==edges_y.shape[0], edges_y.shape[0] -1, y_loc)
fast_density = PDF.data[y_loc, x_loc]
# scatterplots colored based on density
plt.figure(figsize=(6, 4))
plt.scatter(x, y, c=fast_density, s=1, marker='o', cmap='jet', alpha=1)
#ラベルの表示
plt.title('california housing dataset')
plt.xlabel('Medlnc')
plt.ylabel('Housing Prices')
# プロットを表示
plt.show()
R: california housing
# scikit-learnのデータセットと同じDataframeの準備
# Based on scikit-learn function of the same name!
# Download and untar data in a temporary directory
setwd(tempdir())
url <- "http://www.dcc.fc.up.pt/~ltorgo/Regression/cal_housing.tgz"
download.file(url, destfile = "cal.tar.gz")
untar("cal.tar.gz")
untar("cal.tar.gz", files = "cal_housing.pkz")
# Read the data into R and provide column names
cal_housing <- read.csv("CaliforniaHousing//cal_housing.data", header = FALSE)
columns_index <- c(8, 7, 2, 3, 4, 5, 6, 1, 0) + 1
cal_housing <- cal_housing[, columns_index]
names(cal_housing) <- c("Price", "MedInc", "HouseAge", "AveRooms",
"AveBedrms", "Population", "AveOccup", "Latitude",
"Longitude")
# Transform/rescale the data
cal_housing$AveRooms <- cal_housing$AveRooms / cal_housing$AveOccup
cal_housing$AveBedrms <- cal_housing$AveBedrms / cal_housing$AveOccup
cal_housing$AveOccup <- cal_housing$Population / cal_housing$AveOccup
cal_housing$Price <- cal_housing$Price / 100000
# 平均収入と住宅価格の取り出して配列に変換
hoge <- cal_housing[2:1]
colnames(hoge) <- NULL
x <- as.matrix(hoge[1])
y <- as.matrix(hoge[2])
# 色の指定
jet_palette <- colorRampPalette(c("#00008F", "#0000FF", "#0080FF", "#00FFFF", "#80FF80", "#FFFF00", "#FF8000", "#FF0000", "#800000"))
# 密度と色の計算
col1 <- densCols(x, y, colramp = jet_palette)
# プロットの描画
plot(0, 0, type = "n", xlim = c(0, max(x)), ylim = c(0, max(y)),xlab = "x", ylab = "y")
points(x, y, col = col1, pch = 16, cex = 0.4)