初めに
scikit learnのbreast cancer datasetsを利用しPCAをやってみます。
これはそのコードを保存するために作成しました。
予備知識
Scalingについて理解しましょう。まず、mglearnをインストールします。
pip install mglearn
pip install joblib==1.1.0
import mglearn
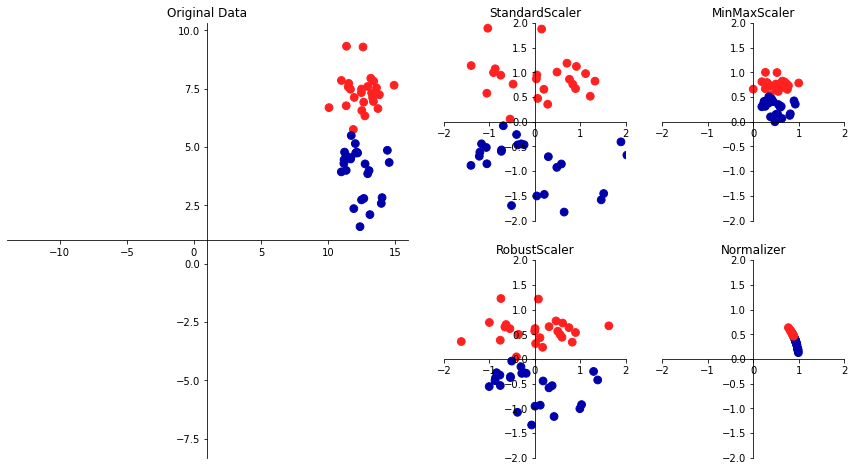
mglearn.plots.plot_scaling()
結果
良く使うのは、1. StandardScaler と 2. MinMax Scalerです。
Breast Cancer Datasets
Datasetsを入手し、Scalingしてみましょう。
from sklearn.datasets import load_breast_cancer
cancer = load_breast_cancer()
中身
print(type(cancer))
<class 'sklearn.utils.Bunch'>
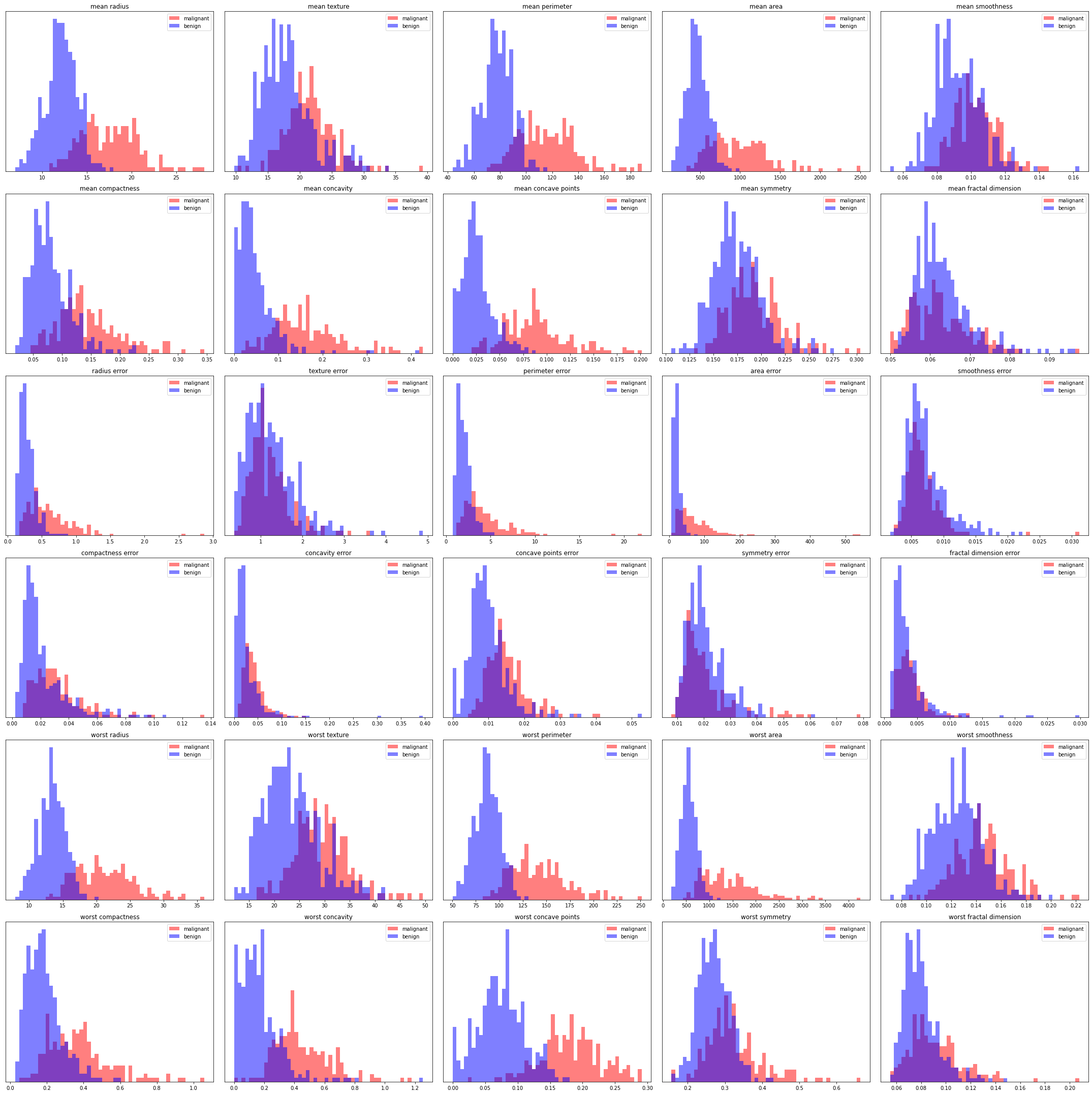
可視化してみましょう。 axes.ravel()には注目してください。これを使うとnested loopを使わなくてもいいです。便利です。
import matplotlib.pyplot as plt
import numpy as np
fig, axes = plt.subplots(6,5, figsize = (30,30))
malignant = cancer.data[cancer.target==0]
benign = cancer.data[cancer.target==1]
ax = axes.ravel()
from numpy import histogram
for i in range(30):
_, bins = np.histogram(cancer.data[:,i], bins=50)
ax[i].hist(malignant[:,i], bins=bins, color='red',alpha=0.5)
ax[i].hist(benign[:,i], bins=bins, color='blue',alpha=0.5)
ax[i].set_title(cancer.feature_names[i])
ax[i].legend(['malignant', 'benign'], loc='best')
ax[i].set_yticks(())
fig.tight_layout()
plt.show()
Scaling
cancer dataをスケールしましょう。
from sklearn.preprocessing import StandardScaler
scaler = StandardScaler()
scaler.fit(cancer.data)
X_scaled = scaler.transform(cancer.data)
PCA
それではPCAを始めます。
from sklearn.decomposition import PCA
pca = PCA(n_components=2)
pca.fit(X_scaled)
X_pca = pca.transform(X_scaled)
結果
print(f'original data: {X_scaled.shape}')
print(f'after PCA data: {X_pca.shape}')
print(f' Explaiend variance ratio : {pca.explained_variance_ratio_}')
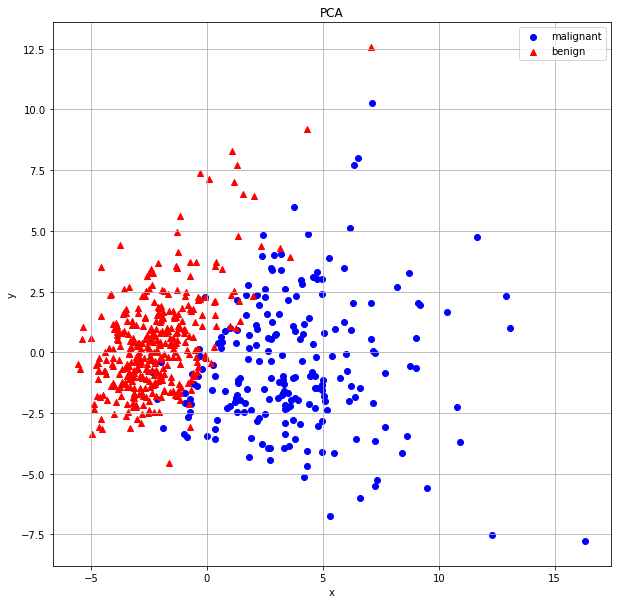
PCAの後、次元が30から2に削減されています。そして、説明可能な分散の比は、PCA1が44.2%, PCA2が18.9%で、合計63.1%です。
結果
original data: (569, 30)
after PCA data: (569, 2)
Explaiend variance ratio : [0.44272026 0.18971182]
可視化のために、pandasを利用し、dataframeの操作を行います。
ガンの陰性と陽性に関する別々のdataframeを作ります。これは最後の散布図で陰性と陽性の色分けをしたいからです。
import pandas as pd
df_X_pca = pd.DataFrame(X_pca, columns=['PC1','PC2'])
df_target = pd.DataFrame(cancer.target, columns=['target'])
df_X_pca = pd.concat([df_X_pca, df_target] , axis=1)
df_X_pca_malignant = df_X_pca[df_X_pca['target'] ==0]
df_X_pca_benign = df_X_pca[df_X_pca['target'] ==1]
可視化のコードです。
fig, ax = plt.subplots( figsize = (10,10))
ax.scatter(x = df_X_pca_malignant['PC1'], y = df_X_pca_malignant['PC2'], color='blue')
ax.scatter(x = df_X_pca_benign['PC1'], marker='^',y = df_X_pca_benign['PC2'], color='red')
ax.legend(['malignant', 'benign'], loc='best')
ax.set_title('PCA')
ax.set_xlabel('x')
ax.set_ylabel('y')
ax.grid()
fig.show()
感想
PCA、教師なし学習として、なかなか良さそうですね。ロジックも確かでシンプルであることが気に入ります。
参考資料
2017年の本ですが、自分の中で機械学習ベスト1の教材です。
https://www.amazon.co.jp/Python%E3%81%A7%E3%81%AF%E3%81%98%E3%82%81%E3%82%8B%E6%A9%9F%E6%A2%B0%E5%AD%A6%E7%BF%92-%E2%80%95scikit-learn%E3%81%A7%E5%AD%A6%E3%81%B6%E7%89%B9%E5%BE%B4%E9%87%8F%E3%82%A8%E3%83%B3%E3%82%B8%E3%83%8B%E3%82%A2%E3%83%AA%E3%83%B3%E3%82%B0%E3%81%A8%E6%A9%9F%E6%A2%B0%E5%AD%A6%E7%BF%92%E3%81%AE%E5%9F%BA%E7%A4%8E-Andreas-C-Muller/dp/4873117984