はじめに
本記事では、二次元コロイド結晶画像を用いて、下のような解析を行います。
- コロイド位置の検出
- 動径分布関数(のようなもの)の導出
- 第1近接原子の計数とプロット
- 近接原子の配位角度を指標とした多結晶粒可視化
コロイド画像は以下のものを使いました![]()

作者 Zephyris [CC BY-SA 3.0 (https://creativecommons.org/licenses/by-sa/3.0) または GFDL (http://www.gnu.org/copyleft/fdl.html)], ウィキメディア・コモンズより(リンク)
使用するライブラリ一覧
以下の通りです。
import cv2
import numpy as np
import matplotlib.pyplot as plt
import matplotlib.cm as cm
from scipy import ndimage as ndi
from sklearn.neighbors import NearestNeighbors
コロイド位置の検出
コロイド位置の検出には、最大値フィルタを用います。
この方法では、ImageJのFind Maximaとほぼ同じことをやります。
まず以下のように画像を読み込み、グレースケールに変換します。
ついでに画像サイズも取得しておきます。
# 画像読み込み
img_raw = cv2.imread('ColloidCrystal_10xBrightField_GlassInWater.jpg', 1)
# グレースケール化
img = cv2.cvtColor(img_raw, cv2.COLOR_BGR2GRAY)
# 画像の高さ, 幅を取得
h, w = img.shape[:2]
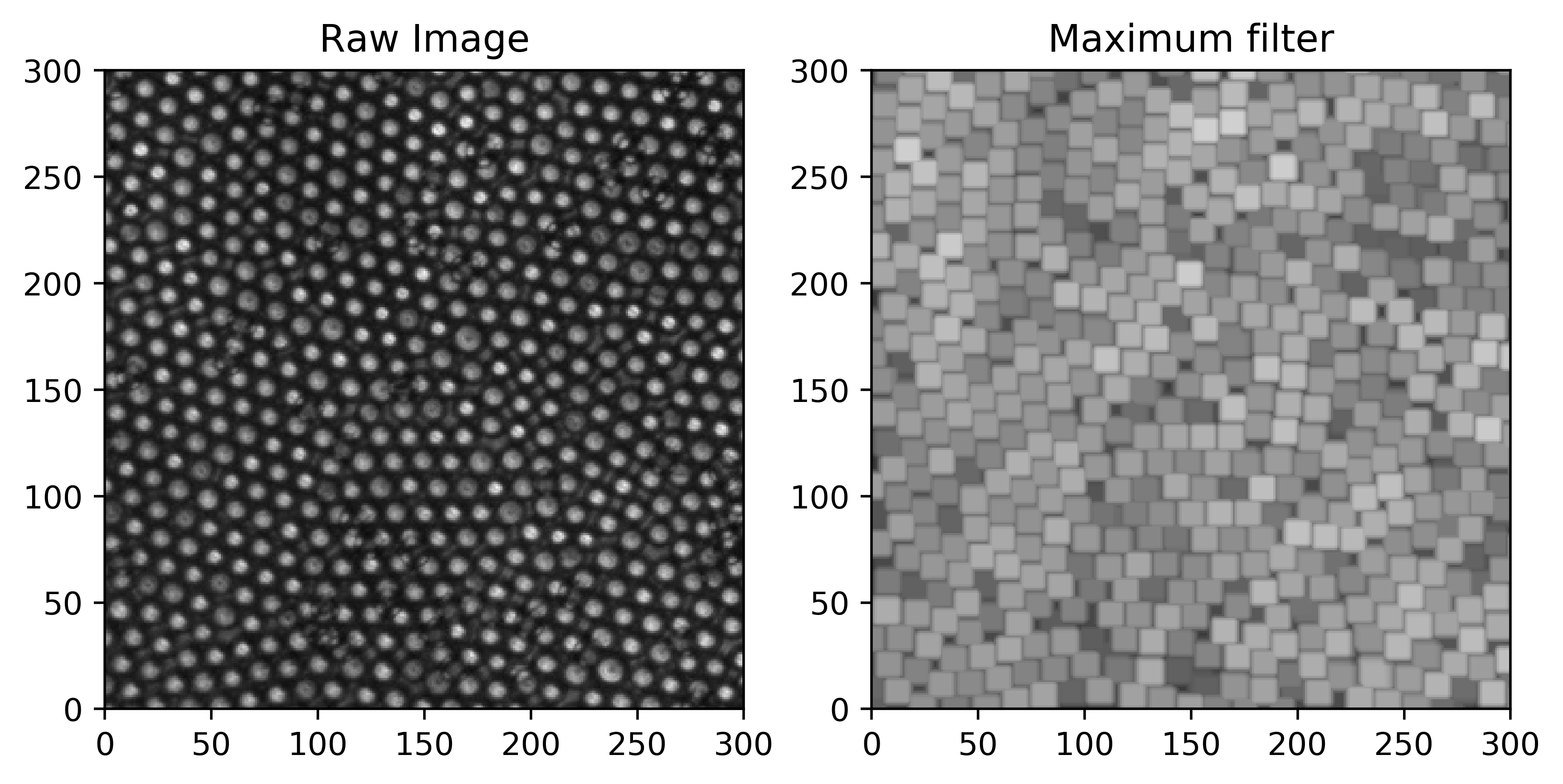
最大値フィルタを用いた粒子検出
最大値フィルタは名前の通り、注目画素の周りのある範囲内における最大輝度を参照するフィルタです。
これを用いることで画像の局所最大値、つまりコロイド粒子の位置を検出することができます。
注目範囲(カーネル)の大きさを9×9としてフィルターを適用してみます。
(適用前にスムージングでノイズ除去を行っています)
# ぼかし(スムージング)によるノイズ除去
img = cv2.blur(img,(3,3))
# 最大値フィルタの適用
img_max = ndi.maximum_filter(img, size=9, mode='constant')
この2つの画像の中で、輝度の値が同じ位置が局所最大値、つまり粒子の位置となります。(参考)
これはnumpy.whereを使えば簡単に求められます。
# 局所最大値の抽出
pts_list = np.where(img == img_max)
pts_list = np.array(pts_list)
# 元画像上へプロット
fig = plt.figure(dpi=150,facecolor='white')
plt.gray()
plt.imshow(img)
plt.scatter(pts_list[1],pts_list[0],s=3,c='red',alpha=0.7)
plt.xlim(0,300)
plt.ylim(0,300)
このように求めた点を元画像の上に表示したものを下に示します。
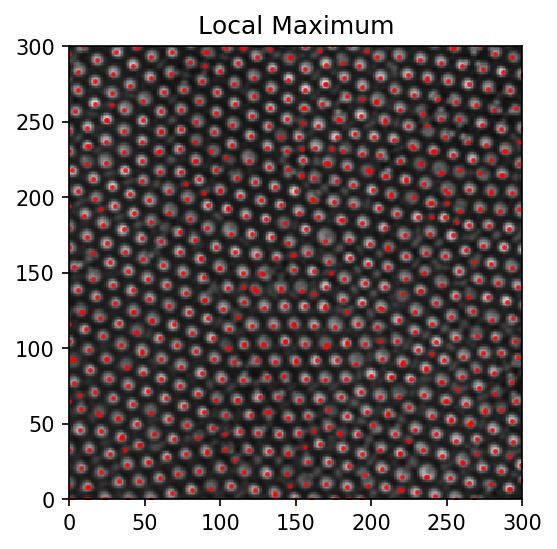
おおむね粒子の位置を検出できていますね。
ただ、いくつかの粒子が2重に検出されてしまっています。
これを取り除くために、ある程度近接した点はその重心を代表としてまとめてしまうことにします。
重複点の削除
点と点との距離の計算には、sklearn.neighbors.NearestNeighborsを使います。
nbrs = NearestNeighbors(n_neighbors=10, algorithm='ball_tree').fit(pts_list.T)
distances, indices = nbrs.kneighbors(pts_list.T)
Nearest Neighborでは、第何近接目までかを指定することで、近接点までの距離とインデックスを返してくれます。
今回は、3ピクセル以内に存在する点は同じ粒子に由来するとして計算していきます。
# 近接点の閾値
th = 3
# 閾値以下の近接点との重心を計算。
center_list = []
for d,i in zip(distances,indices):
i = i[np.where(d < 3)]
pts = pts_list.T[i]
center = np.mean(pts,axis=0)
center_list.append(center)
center_list = np.array(center_list).astype(np.int32)
# 重複を削除
center_list = np.unique(center_list,axis=0)
center_list = center_list.T
このように求めた点を同様に図示すると下のようになります。
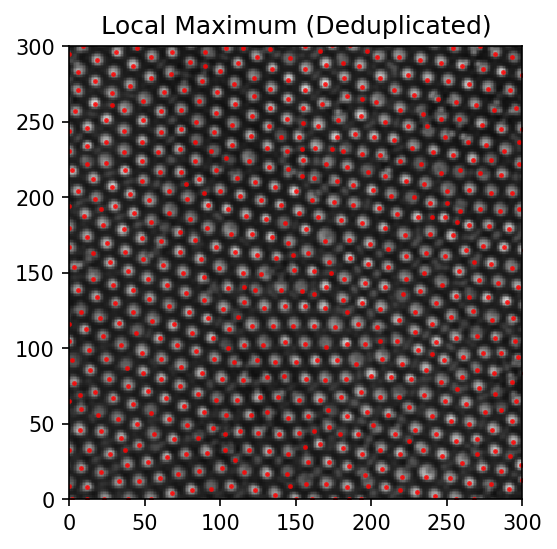
1つの粒子内で重複していた点がまとめられています。
これで粒子検出は完了です ![]()
![]()
動径分布関数(のようなもの)の導出
それでは次に、動径分布関数(RDF: Radial Distribution Function)の導出を行います。
RDFは、ある粒子からの距離に対して、他の粒子が存在する確率を示すものであり、結晶の周期構造を記述するものとしてよく用いられています。
RDFの導出とその計算法に関しては以下を参照してください。
動径分布関数の計算 - Koishi's Page
近接原子の計算には、先ほど用いたsklearn.neighbors.NearestNeighborsを使います。
# 近接原子の位置と距離を40個まで計算
nbrs = NearestNeighbors(n_neighbors=40, algorithm='ball_tree').fit(center_list.T)
distances, indices = nbrs.kneighbors(center_list.T)
# 50ピクセル以内に存在する他粒子との距離を計算し、ヒストグラムを作成
dist_list = []
for d,i in zip(distances,indices):
d = d[np.where(d < 50)][1:]
dist_list.extend(d)
dist_list = np.array(dist_list)
dist_hist = np.histogram(dist_list,range=(0,50),bins=50)
# RDFの計算
rdf = dist_hist[0]/(4*np.pi*np.power(dist_hist[1][1:],2))
これだけでオッケーです。
今回は50ピクセルまでの範囲でRDFを計算しました。
(最後に密度で割っていないので厳密には異なりますが、今回はピーク値がわかればOKとします)
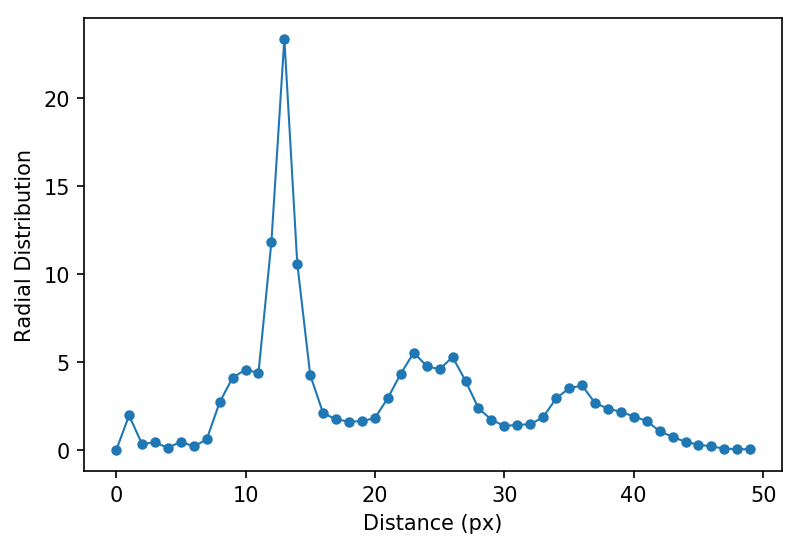
結果を図示すると以下のようになります。
第1, 2, 3近接までのピークがわかりますね。
今回のコロイド結晶は密充填していますが、ところどころに欠陥があるので、ピークが分裂したりブロードになったりしています。
とりあえずこれで、第1近接粒子までの距離はだいたい10~18ピクセルぐらいであることが分かりました。
それでは次に、この第1近接粒子の数を数えてみましょう。
第1近接原子の計数とプロット
球状コロイドが2次元に最密充填する場合、第1近接粒子との配位数は6となります。
これ以下となる部分は、コロイドがきれいに配列していない場所です。
つまり、第1近接粒子の数をプロットすれば、コロイド結晶の欠陥を図示することができます。
ではやっていきましょう![]()
![]()
使うのは同じくsklearn.neighbors.NearestNeighborsです。
第1近接原子とみなす閾値は16 pxとします。
# 第1近接原子の計数
co_list = []
for d,i in zip(distances,indices):
d = d[np.where(d < 16)][1:]
co_list.append(d.shape[0])
また、第1近接粒子の個数をヒストグラムで表示した図を下に示します。
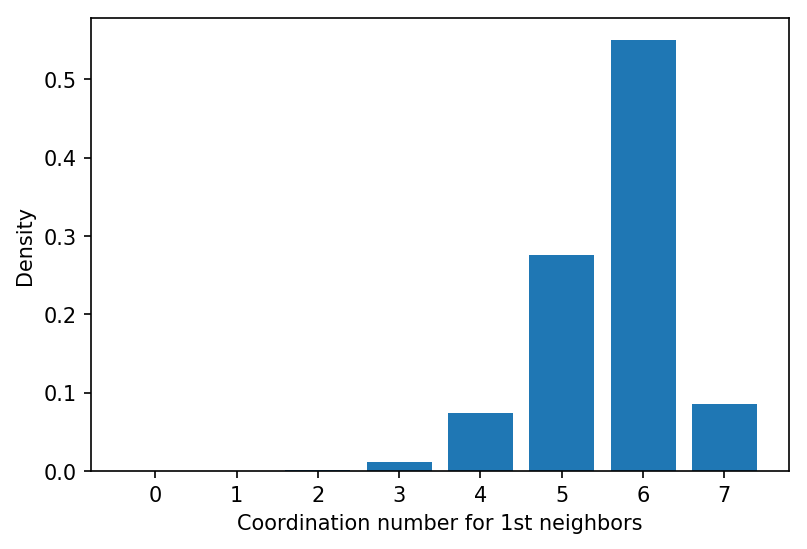
だいたい最近接粒子数は3~7の間に分布していますね。
ではとりあえす、(3,6)の範囲でプロットしてみます。
fig = plt.figure(dpi=150,facecolor='white')
plt.imshow(img)
plt.scatter(center_list[1],center_list[0],s=3,c=co_list,cmap='rainbow',alpha=0.7)
plt.colorbar()
plt.clim(3,6)
plt.xlim(0,1000)
plt.ylim(0,1000)
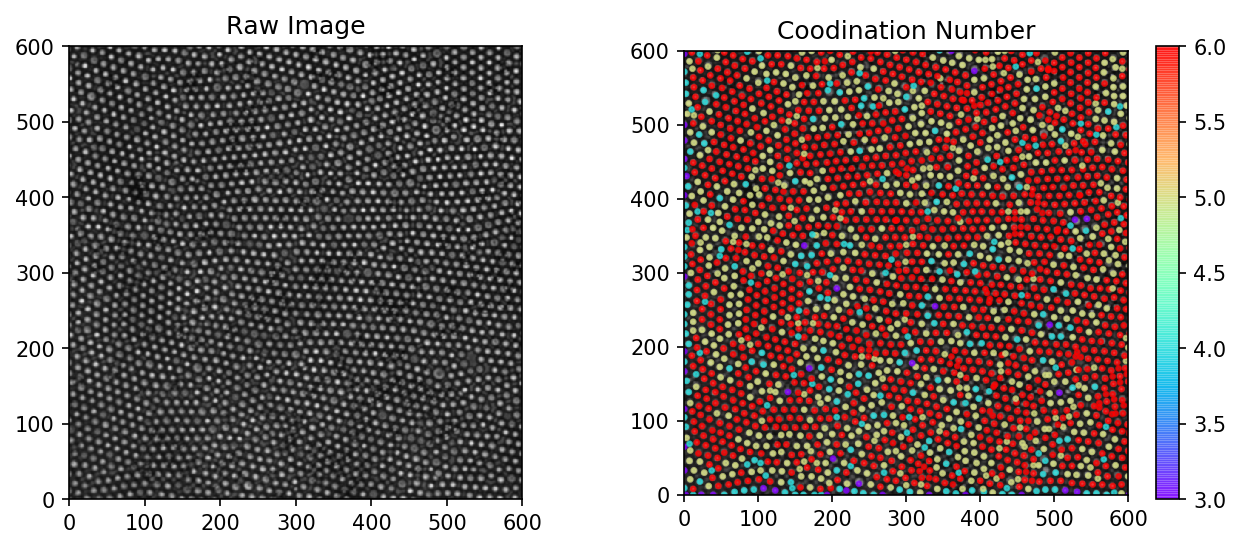
元画像を左に、各粒子を配位数で色分けしたプロットを右に示しています。
配列が乱れている部分 = 欠陥の場所が図示できてます ![]()
![]()
近接原子の配位角度を指標とした多結晶粒可視化
では最後に、コロイド結晶の多結晶粒の可視化を行っていきます。
具体的には、EBSD(電子線後方散乱回折)で得られるような結晶粒の2次元的なマッピング(下図参考)を、コロイド結晶についても行います。
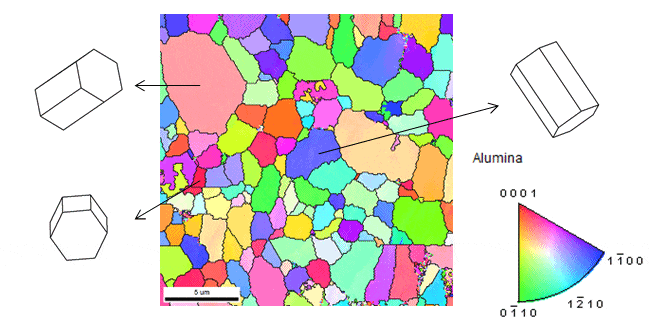
(引用元:https://www.ube-ind.co.jp/usal/documents/m1303_550.htm)
このような解析はコロイド結晶に関する論文でも行われていて、例えば以下の論文のFig.1では、注目粒子⇒第1近接粒子のなすベクトルを、X軸とのなす角度で分類して結晶粒の可視化を行っています。
同様の手法で可視化を行ってみましょう!
theta_list = []
for d,i in zip(distances,indices):
#自身を除く近接粒子を抽出
idx = i[np.where(d < 16)][1:]
cnts = center_list.T[idx]
#最もXの値が大きい粒子の位置を取得
top = cnts[np.argmin(cnts[:,1])]
#注目している粒子の位置を取得
center = center_list.T[i[0]]
#X軸との角度を計算
axis = np.array([0,center[1]])
u = top - center
v = axis - center
c = np.inner(u, v) / (np.linalg.norm(u) * np.linalg.norm(v) + 1e-99)
a = np.rad2deg(np.arccos(np.clip(c, -1.0, 1.0))) - 90
theta_list.append(a)
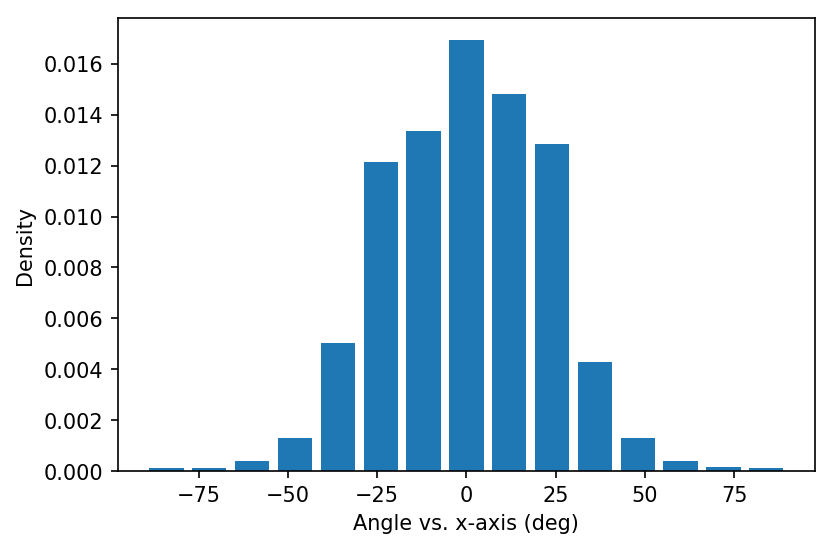
このように得られた角度のヒストグラムを以下に示します。
角度は、だいたい±30 degの範囲内に収まっているみたいです。
それではプロットしてみましょう!
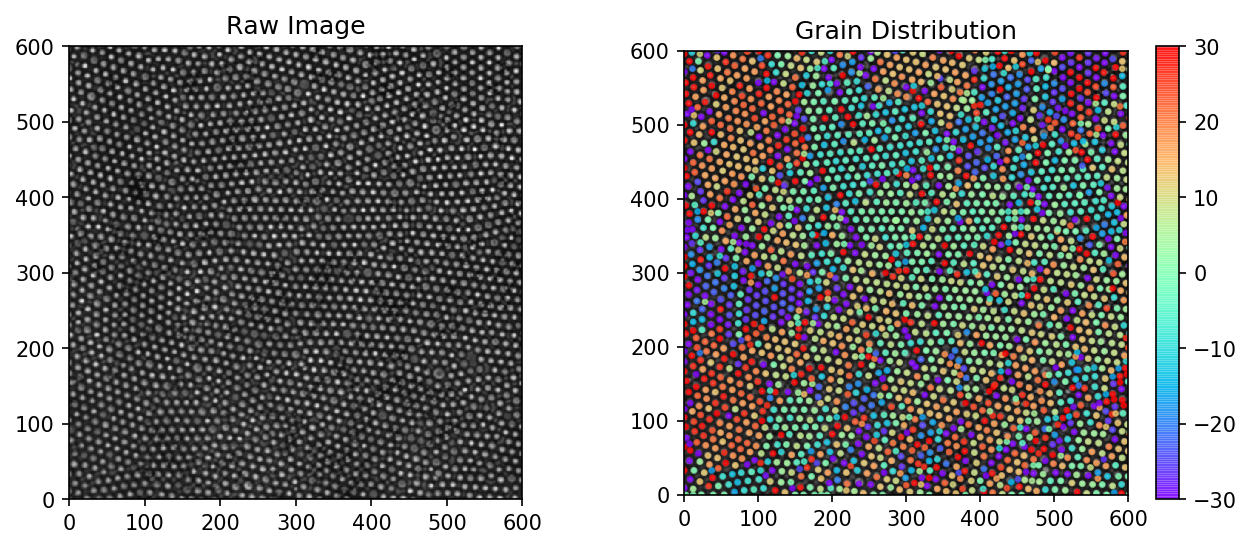
元画像を左に、第1近接原子の角度を色分けしてプロットした図を右に示しています。
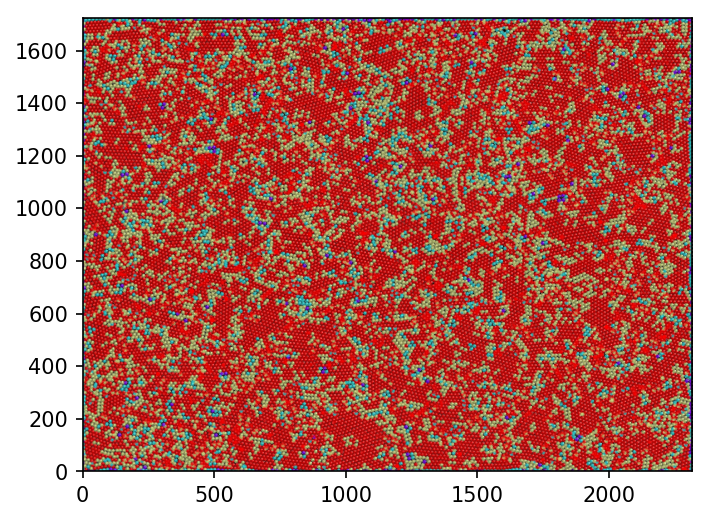
最後に、画像全体でこの色分けを行ってみます!
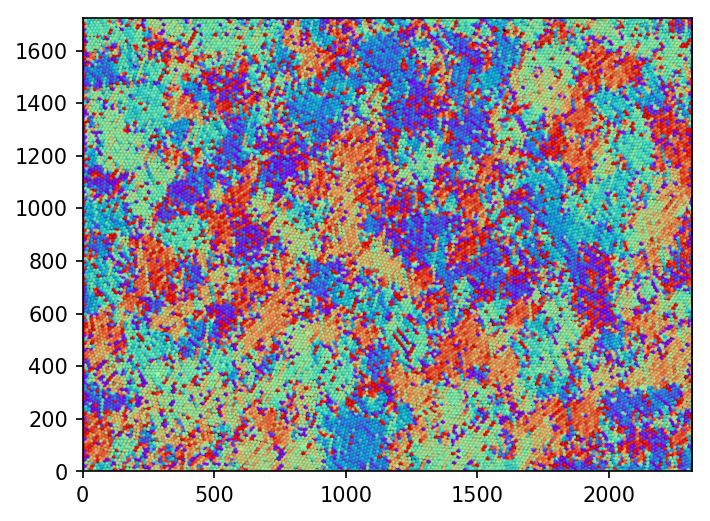
コロイド結晶の結晶粒がはっきりとわかりますね ![]()
![]()
参考にした論文のマッピングと比べるとノイズが大きい気もしますが、やっつけ仕事にしては十分でしょう。
カラフルな図でライバルにアピールだ!
最後に
最近、コロイド関係の研究をされている方から画像解析の依頼があり、家に引きこもりながらいろいろと解析をやってました。
とりあえずひと段落ついたので、ネット上に転がっていたコロイド結晶の画像を題材にノウハウをまとめてみた感じです。
実際、実験で得られる画像にはノイズが激しい場合も多いので、適切なフィルター処理やデノイズが必要となったりします。
こっちに関しては、基本的に出たとこ勝負というか、試行錯誤が必要な部分になります。
そのうち役に立つ手法をまとめたいなーと思ってます。とっ散らかった記事になりそうです。
それでは~。