概要
一般的な二値化(例えば大津の方法)がうまくいかない場合でも使える領域検出方法、Local standard deviation filterの紹介です。

SEM(電子顕微鏡)像からの領域検出を例として、実際の使用方法を解説します!
はじめに ~一般的な領域検出手法~
たとえば下のような画像から領域の検出をしたいとします。
(from 31 downloadable sample images and stacks - ImageJ)
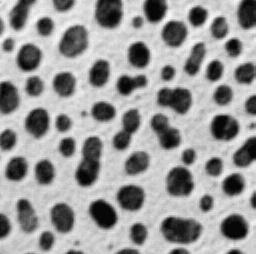
画像処理に慣れている人であれば、ガウシアンフィルタで適当にノイズ処理⇒大津の方法で二値化をするのではないでしょうか?
この流れをPython(OpenCV)で書いてみるとこんな感じです。
# 画像の読み込み
img = cv2.imread('blobs.png',0)
# (5×5)のガウシアンフィルタで平滑化
img = cv2.GaussianBlur(img,(5,5),0)
# 大津の方法で二値化
__,th = cv2.threshold(img,0,255,cv2.THRESH_BINARY_INV+cv2.THRESH_OTSU)
# 二値化領域をRGB化
dec = cv2.cvtColor(th, cv2.COLOR_GRAY2RGB)
dec[np.where(th == 255)] = [255,0,0]
# 元画像をRGB化して重ね合わせ
img_color = cv2.cvtColor(img, cv2.COLOR_GRAY2RGB)
dst = cv2.addWeighted(img_color, 0.5, dec, 0.5, 0)
# 結果を保存
cv2.imwrite('result.png',dst)
結果は下のようになります。
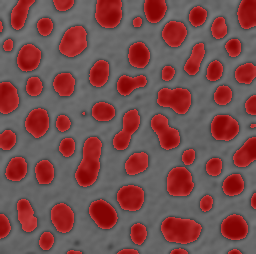
領域検出ができれば、あとは目的に応じて、この部分の面積分布やアスペクト比とかが分析できるようになります。
SEM画像への適用
ところが、微細組織の観察によく用いられるSEM(走査型電子顕微鏡)で撮影された画像の場合は、なかなか領域検出がうまくいかない場合が多いです。
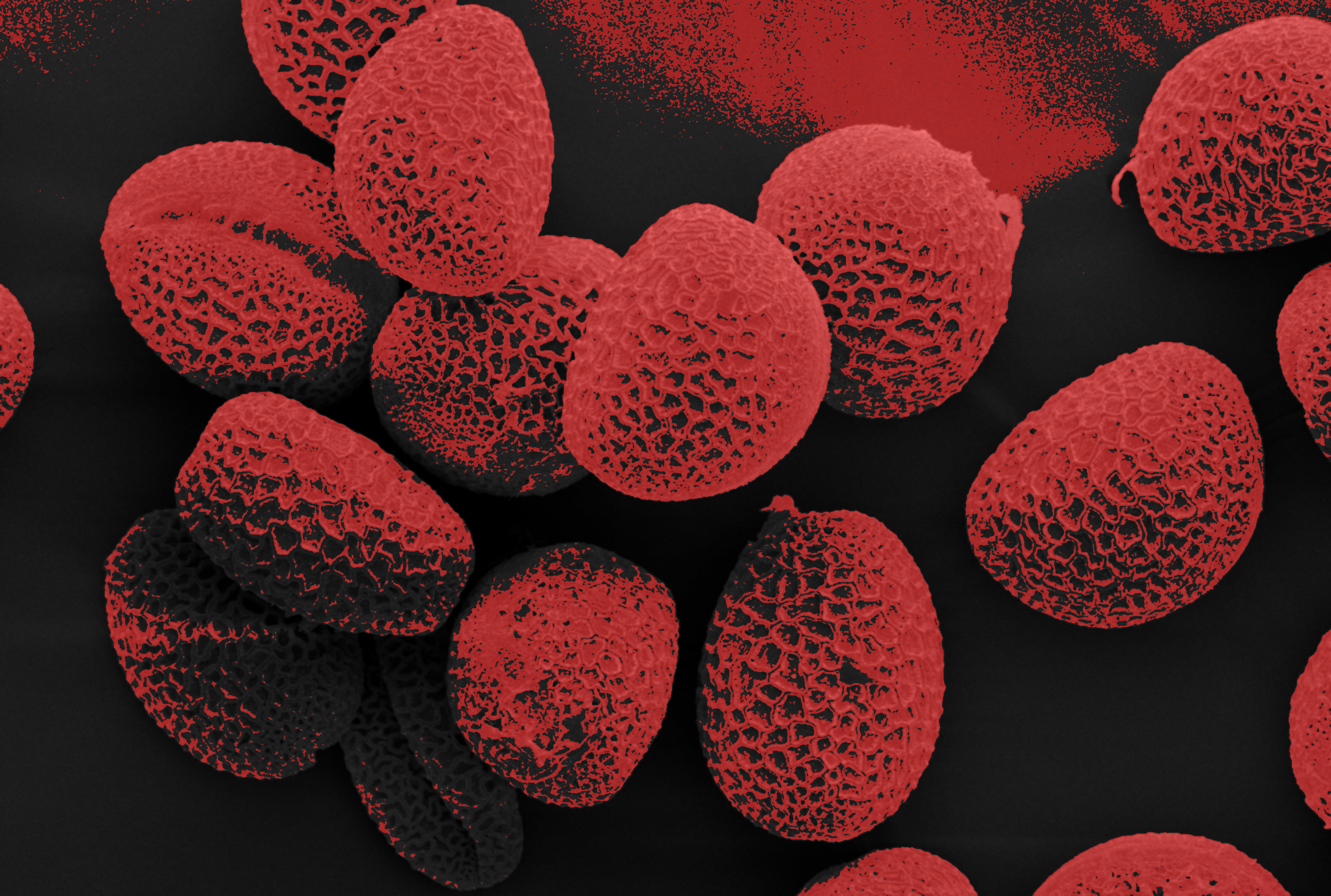
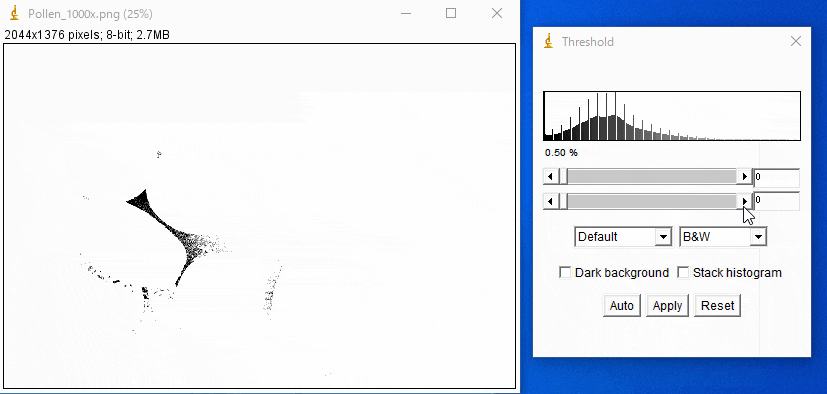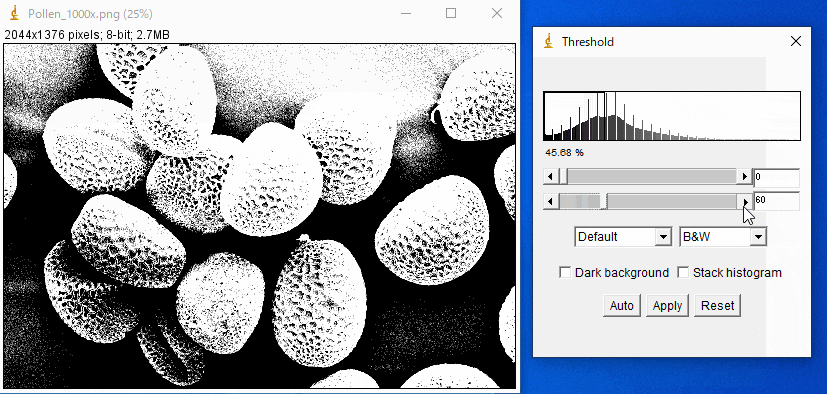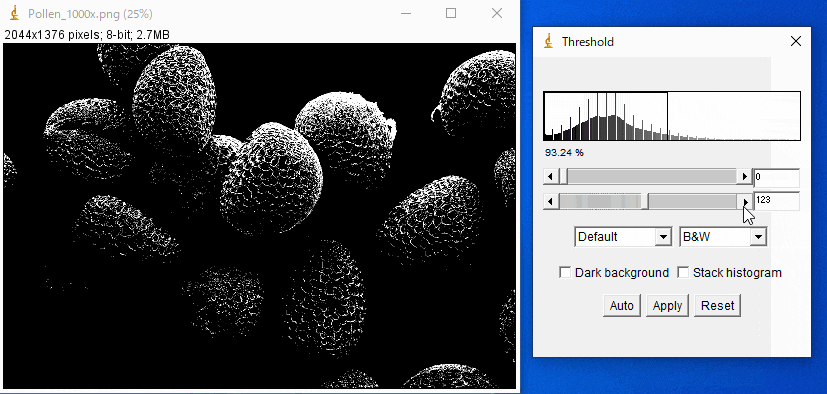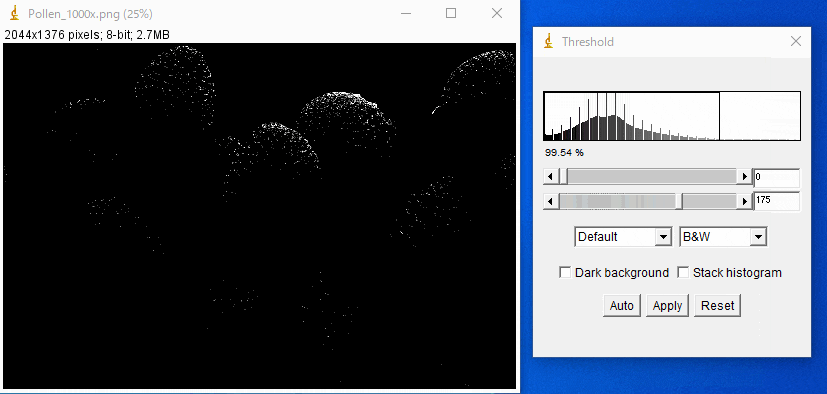
例えば下の花粉の画像を例に考えてみましょう。
(from wikimedia commons: Pollen 1000x - SEM MUSE)
この画像を同様の方法で二値化すると、以下のようになります。
うまく分離できていませんね。
SEM観察像は試料の角で明るくなる特徴(エッジ効果)があり、領域の内外でコントラストが明確に分かれないことが多いです。
ImageJのようなソフトを使って二値化の閾値(Threshold)の値を色々と変えてみればわかるのですが、実際、この画像を輝度だけで分離することは不可能です。
画像の局所的な標準偏差を用いた領域検出
しかし、人の目で見てみると、花粉の領域とバックグラウンドが明確に分かれて見えるのも確かです。なぜそれがわかるかといえば、花粉の表面には特徴的な模様があるからですね。
このように、表面の模様・質感(テクスチャ)の違いをうまいこと使う方法として、画像の局所的な標準偏差(Local standard deviation filter)を用いた領域分離が存在します。
これは、ガウシアンフィルタのような空間フィルタリングと同じように、注目画素から設定した幅の内部領域に注目します。
例えば中央値フィルタはこの領域内の画素の中央値を、ガウシアンフィルタは中心からの距離の重み付き平均を取る操作を行います。
一方、Local standard deviation filterでは、画像の局所的なテクスチャを比較するために、この領域内の輝度の標準偏差を取る操作を行います。
Local std filterのPythonへの実装
MATLABではstdfiltという関数がこの操作に対応しますが、OpenCVには実装されていないようなので、自分で以下のように関数として実装しました。
def LocalStdFilter(img,width):
if(width%2 == 0):
print('width size should be odd.')
return None
trim_width = int((width - 1)/2)
h,w = img.shape[:2]
img_trim = img[trim_width:h-trim_width,trim_width:w-trim_width]
result = np.zeros_like(img_trim)
for n,i in enumerate(tqdm(img_trim)):
for m,j in enumerate(i):
y,x = n+trim_width,m+trim_width
img_window = img[y-trim_width:y+trim_width,x-trim_width:x+trim_width]
result[n,m] = np.std(img_window)
result = np.pad(result,(trim_width,trim_width),'constant')
return result
これを使って、領域検出をやってみます。
フィルターのサイズはとりあえず21に設定しました。
(画像サイズは半分にしてます)
# 画像の読み込みとリサイズ
img = cv2.imread('Pollen_1000x.png',0)
h,w = img.shape[:2]
img = cv2.resize(img, (int(w/2),int(h/2)))
# Local standard deviation filterの適用
result = LocalStdFilter(img,21)
# Std = 5を閾値に二値化
__,th = cv2.threshold(result,5,255,cv2.THRESH_BINARY)
# モルフォロジー変換で検出領域を縮小後、RGBに変換
th[np.where(th == 255)] = 1
th = cv2.erode(th,np.ones((9,9),np.uint8),iterations = 1)
dec = cv2.cvtColor(th, cv2.COLOR_GRAY2RGB)
dec[np.where(th == 1)] = [255,0,0]
# 元画像と重ね合わせて表示
img_color = cv2.cvtColor(img, cv2.COLOR_GRAY2RGB)
dst = cv2.addWeighted(img_color, 0.5, dec, 0.5, 0)
1枚あたり15秒ぐらいかかります。かなり遅いですね・・・。
では、結果を表示してみます。
fig = plt.figure(figsize=(9,7),dpi=200)
plt.subplot(221)
plt.title('Raw Image',fontsize=14)
plt.imshow(img)
plt.subplot(222)
plt.title('Local Std Filter',fontsize=14)
plt.imshow(result)
plt.subplot(223)
plt.title('Thresholding (Std = 5)',fontsize=14)
plt.imshow(dec)
plt.subplot(224)
plt.title('Result',fontsize=14)
plt.imshow(dst)
花粉の領域がうまく検出できています!
やったぜ![]()
![]()
![]()
まとめ
普通の二値化では領域の分離が難しいようなSEM像でも使えるLocal standard deviation filterの紹介を行いました。
今回は領域の特徴量として標準偏差(std)を用いましたが、他の特徴量(2次元FFTとか)を使ってみても面白いかもしれませんね。
正直Pythonで回すのは重いですが、ハマれば強いフィルターです。
ぜひ試してみてください!