結論から書くと、BioRuby の TogoWS クラスを使えばよいです。
KEGG とは
KEGG 京都大学化学研究所でつくられた、代謝経路のデータベースです。パスウェイ解析に使用することが多いようです。
BioRuby とは
Ruby で遺伝子情報などを扱うライブラリです。
https://github.com/bioruby/bioruby
インターネット上にあるBioRubyの情報は古いことが多いので、リファレンスは最新のものを見た方がよいと思われます。
https://www.rubydoc.info/gems/bio
インストール
gem install bio
TogoWS とは
(NCBI, EBI) および (DDBJ, KEGG, PDBj, CBRC) から提供される主要データベースと解析サービスを統合して利用するためにライフサイエンス統合データベースセンターが作成したサービスです。
http://togows.dbcls.jp/
Ruby から TogoWS を使う
まず最初に使えるデータベースを確認します。
エントリーから検索できるデータベースのリストを表示する。
Bio::TogoWS::REST.entry_database_list
検索できるデータベースのリストを表示する。
Bio::TogoWS::REST.search_database_list
grep で KEGG に関係するデータベースを抽出します。
Bio::TogoWS::REST.search_database_list.grep(/kegg/)
| kegg-compound | compound |
| kegg-drug | drug |
| kegg-enzyme | enzyme |
| kegg-genes | genes |
| kegg-glycan | glycan |
| kegg-orthology | orthology |
| kegg-reaction | reaction |
| kegg-module | module |
| kegg-pathway | pathway |
KEGG で PD-1 を検索してみる
今回は、2018年本庶先生のノーベル賞を記念して "PD-1" を検索してみることにします。
PD-1 について書いておくと、この分子は、白血球の表面に生えているスイッチのようなもので、がん細胞が PD-L1 という専用のタンパク質でこれと結合すると、白血球の攻撃を回避することができるそうです。よって、がん細胞に白血球のスイッチを触らせないことが大事になります。そこで、血液中にスイッチのカバーを大量に流すことで、白血球のスイッチを先に覆ってしまい、がん細胞にスイッチを触らせないようにしてしまおうという薬がオプシーボと理解しています。(間違っていたらすみません。)
TogoWS から kegg-gene のなかの PD-1 を検索してみます。
puts Bio::TogoWS::REST.search("kegg-genes", "PD-1")
# たくさん情報が出るので、人間(hsa)にしぼる
puts Bio::TogoWS::REST.search("kegg-genes", "PD-1").split("\n").grep(/hsa/)
結果
hsa:100526842 RPL17-C18orf32, PD-1, RPL17; RPL17-C18orf32 readthrough
hsa:6139 RPL17, L17, PD-1, RPL23; ribosomal protein L17
hsa:64115 VSIR, B7-H5, B7H5, C10orf54, DD1alpha, GI24, PD-1H, PP2135, SISP1, VISTA; V-set immunoregulatory receptor
hsa:5133 PDCD1, CD279, PD-1, PD1, SLEB2, hPD-1, hPD-l, hSLE1; programmed cell death 1
4件ヒットしました。一番下のエントリーが求める PD-1 であると思われます。hsa:5133 は hsapiens (人間)で KEGG エントリーナンバーが 5133 番という意味だと思われます。次にこのエントリーナンバーを使って PD-1 の情報を表示します。
pd1 = Bio::TogoWS::REST.entry("kegg-genes", "hsa:5133")
pd1.class
# => String
puts pd1
以下のように表示されると思います。(フォント:みかちゃん)

これは文字列なので、プログラムで扱いやすいように KEGG::GENES クラスのインスタンスを作成します。
pd1 = Bio::KEGG::GENES.new(pd1)
pd1.class
# => Bio::KEGG::GENES
pd1.entry
=> {"id"=>"5133", "division"=>"CDS", "organism"=>"T01001"}
pd1.name
# => "PDCD1, CD279, PD-1, PD1, SLEB2, hPD-1, hPD-l, hSLE1"
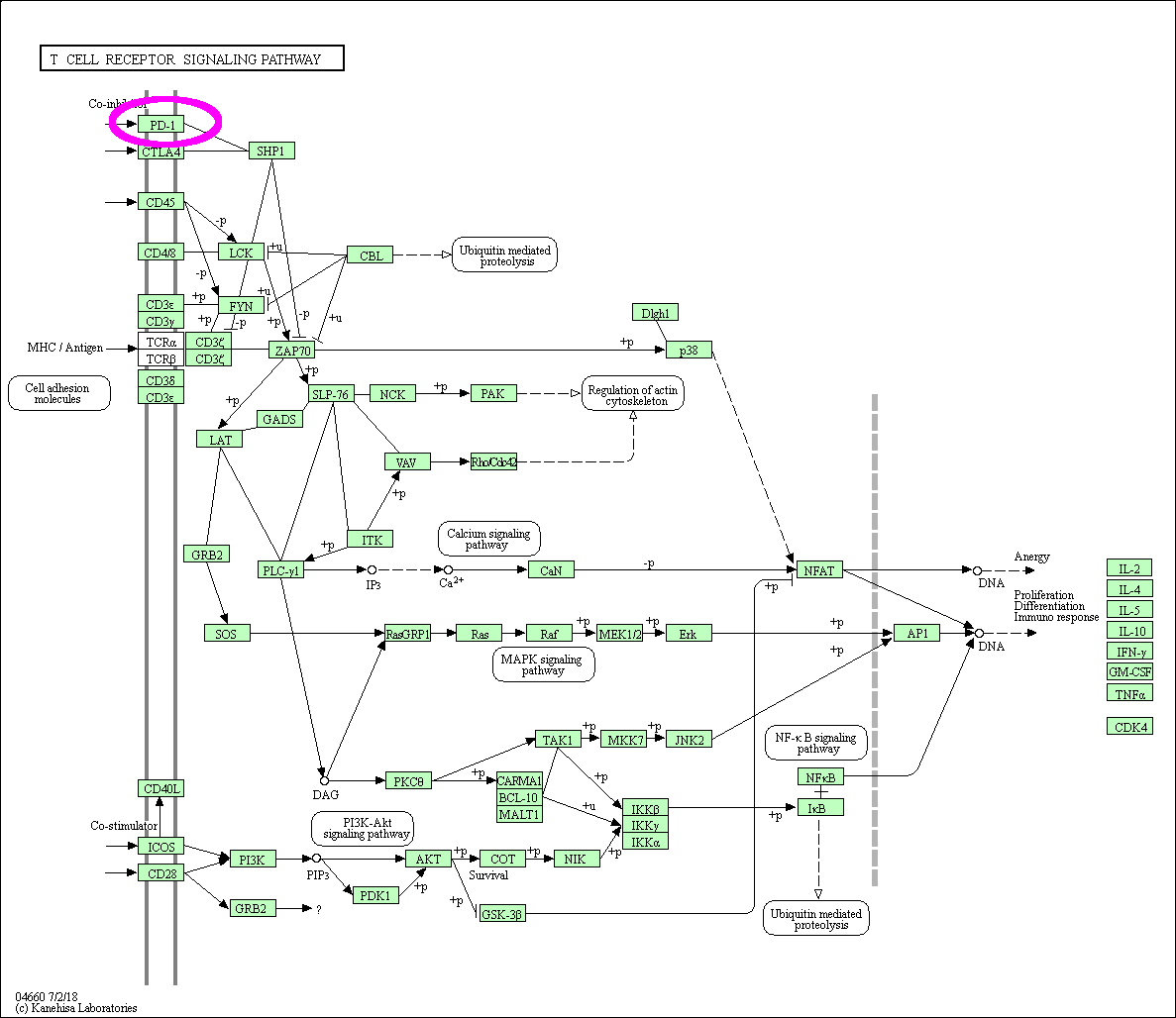
PD-1 の含まれているパスウェイを見てみます。
pd1.pathways
# => {"hsa04514"=>"Cell adhesion molecules (CAMs)",
# "hsa04660"=>"T cell receptor signaling pathway"}
2つあります。このうちの T cell receptor signaling pathway (T細胞受容体シグナルパスウェイ)の情報を取得してみます。
その前に、パスウェイの画像を見てみたいところですが、残念ながら現行(1.5.2)の BioRuby でパスウェイの画像を取得する方法がわかりませんでした。そこで wget で手動で取得します。左上にPD-1があるのがわかると思います。
wget https://www.genome.jp/kegg/pathway/hsa/hsa04660.png
再び TogoWS にアクセスして、Bio::KEGG::PATHWAYクラスのインスタンスを作成します。
str = Bio::TogoWS::REST.entry("kegg-pathway","hsa04660")
tcell_pathway = Bio::KEGG::PATHWAY.new(str)
この pathway に含まれる遺伝子を列挙してみます。
tcell_pathway.genes
# => {"915"=>"CD3D; CD3d molecule [KO:K06450]",
# "916"=>"CD3E; CD3e molecule [KO:K06451]",
# "917"=>"CD3G; CD3g molecule [KO:K06452]",
# "919"=>"CD247; CD247 molecule [KO:K06453]",
# "920"=>"CD4; CD4 molecule [KO:K06454]",
# "925"=>"CD8A; CD8a molecule [KO:K06458]",
# "926"=>"CD8B; CD8b molecule [KO:K06459]",
# "5788"=>"PTPRC; protein tyrosine phosphatase, receptor type C [KO:K06478] [EC:3.1.3.48]",
# "3932"=>"LCK; LCK proto-oncogene, Src family tyrosine kinase [KO:K05856] [EC:2.7.10.2]",
# "2534"=>"FYN; FYN proto-oncogene, Src family tyrosine kinase [KO:K05703] [EC:2.7.10.2]",
# "7535"=>"ZAP70; zeta chain of T cell receptor associated protein kinase 70 [KO:K07360] [EC:2.7.10.2]",
# 以下略
なかなかスムーズにはいかないようですが、Ruby で TogoWS を介して KEGG を検索する方法を書きました。
ここはこうした方がよい、などありましたらコメント欄などで指摘していただけると幸いです。