はじめに
この記事はすごいテキトーな記事なので真に受けるなよろし。
目的
ヒトゲノム計画では、人のDNAの配列が全部明らかにされたと言われていますが、実際にはわかっていない部分もかなりあるはずです。
どこの部分がわかっていないのか、グラフを描きます。
ヒトゲノムのfastaのダウンロード
Gencodeからヒトゲノムの配列がダウンロードできます。
https://www.gencodegenes.org/human/
ここでは、贅沢にALLをダウンロードします。
wget ftp://ftp.ebi.ac.uk/pub/databases/gencode/Gencode_human/release_35/GRCh38.p13.genome.fa.gz
解凍してもいいのですが、zcat コマンドを使えばある程度見ることができますね。
まずは head を表示してみます。
zcat GRCh38.p13.genome.fa.gz | head
>chr1 1
NNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNN
NNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNN
NNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNN
NNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNN
NNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNN
NNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNN
NNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNN
NNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNN
NNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNN
はい、早速出てきました。Nの山です。つまり、配列が特定できていない部分がのっけの頭から来ているわけですね。
次に、一応染色体を確認しておきましょう。
zcat GRCh38.p13.genome.fa.gz | grep "^>"
まあ、こんな感じで大量に出てきます。
次に、本当に配列がTCGANだけの5種類の文字列から構成されているのか確認してみることにします。
きっとコマンドラインツールを使う方法もあると思うのですが、ここはCrystal言語を使って小さなプログラムを書いてしまうことにします。なぜCrystal言語かというと爆速だからですね。
counter = Hash(Char, Int32).new(0)
while l = gets
l.chomp.each_char do |c|
counter[c] += 1
end
end
p counter
はい、Rubyわかる人には何の変哲もないコードですね。ポイントは、Hashの生成時に型を指定しているところだけです。ほかは全部Rubyと同じです。これがCrystalです。ビルドします。
crystal build counter.cr --release
このビルド時間を含めると実行速度でJulia等とあまり変わらないかも知れませんが気にしないことにします。
では、TCGANの数を数えてみましょう
zcat GRCh38.p13.genome.fa.gz | grep -v "^>" | ./count
grep -v "^>" で行頭の染色体などを定義している行を省いています。結果は
{'N' => 161331703, 'T' => 916993511, 'A' => 914265135, 'C' => 635937481, 'G' => 638590158}
となりました。確かに,TCGAおよびN以外の文字列が存在しないことが確認できました。次はNの連続の分布を数えてみましょう。つまりNが何文字連続するか、を調べてみてみることにします。
temp = 0
while l = gets
l.chomp.each_char do |c|
if c == 'N'
temp += 1
elsif temp != 0
puts temp
temp = 0
end
end
end
puts temp if temp != 0
ビルドします。
crystal build nseq.cr --release
まずはNの連続する箇所がいくつあるか調べてみましょう。
zcat GRCh38.p13.genome.fa.gz | grep -v "^>" | ./nseq | wc -l
1234
そんなに多くないことがわかります。これを長い順に並べて表示するとこんな感じになります。
1っていうのが不思議な気がするのと、100とか50000とかが多いなあといった印象ですかね。
本題
さて、適当に10000文字ごとに、N, AT, CGの割合を計算してくれるようなプログラムを書きます。
tcgan = Hash(Char, Int32).new(0)
target = ARGV[0]
chr = ""
loc = 1
flag = false
while l = gets
if l.starts_with?(">")
exit if flag
if l == target
puts "loc\tN\tAT\tCG"
flag = true
end
next
end
if flag
l.chomp.each_char do |c|
tcgan[c] += 1
loc += 1
if loc % 10000 == 0
total = tcgan.values.sum.to_f
ta = (tcgan['A'] + tcgan['T']) / total
cg = (tcgan['G'] + tcgan['C']) / total
n = tcgan['N'] / total
puts "#{loc}\t#{n}\t#{ta}\t#{cg}"
tcgan = Hash(Char, Int32).new(0)
end
end
end
end
ためしに実行します。
zcat GRCh38.p13.genome.fa.gz | ./n2 ">chr1 1" | head
loc N AT CG
10000 1.0 0.0 0.0
20000 0.0001 0.4076 0.5923
30000 0.0 0.4826 0.5174
40000 0.0 0.5288 0.4712
50000 0.0 0.6439 0.3561
60000 0.0 0.6346 0.3654
70000 0.0 0.6669 0.3331
80000 0.0 0.6199 0.3801
90000 0.0 0.6294 0.3706
だいたいうまくいってるようですね。Rubyで同じ動作のプログラムを作成すると、実行時間がすごくがかかってしまいますがCrystalは超高速です。これをコマンドラインでuplotに投げていきます。
uplotというのは、UnicodePlots.rbを使ってターミナル上にグラフを表示できる私が個人的に作っているRuby製のツールです。
ここからターミナルにグラフを描いて、それをスクリーンショットを取ってQiitaに貼り付けるというかなり意味のわからないことをしています。
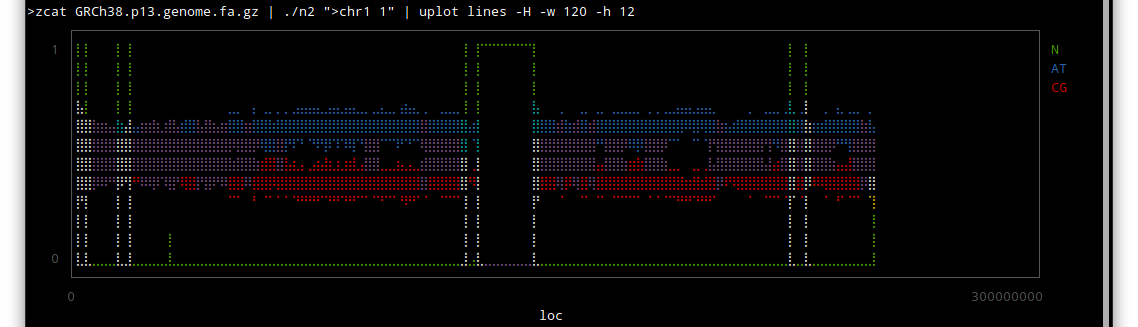
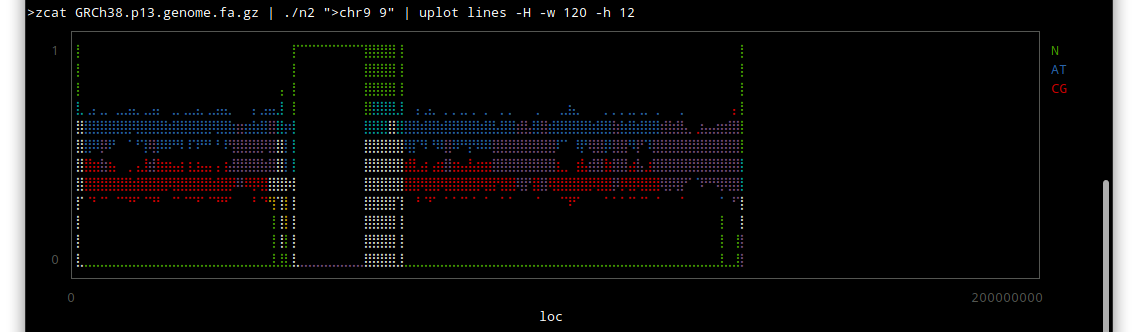
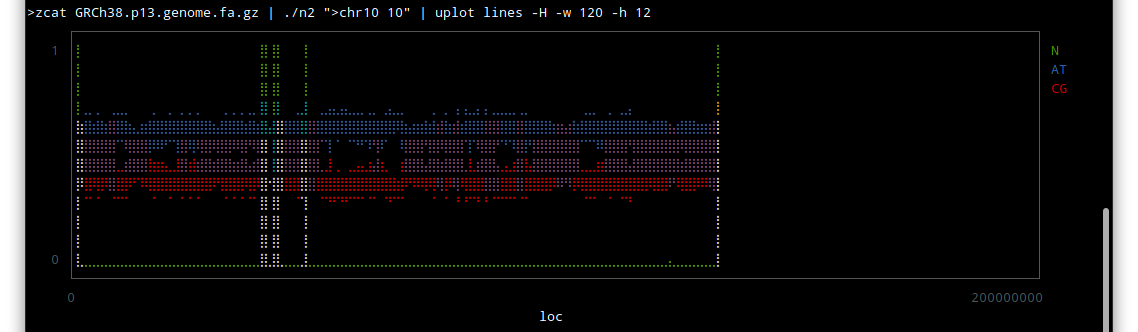
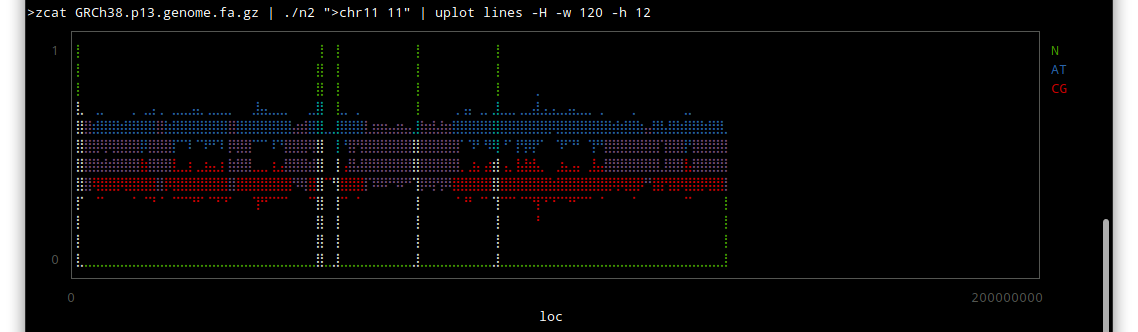
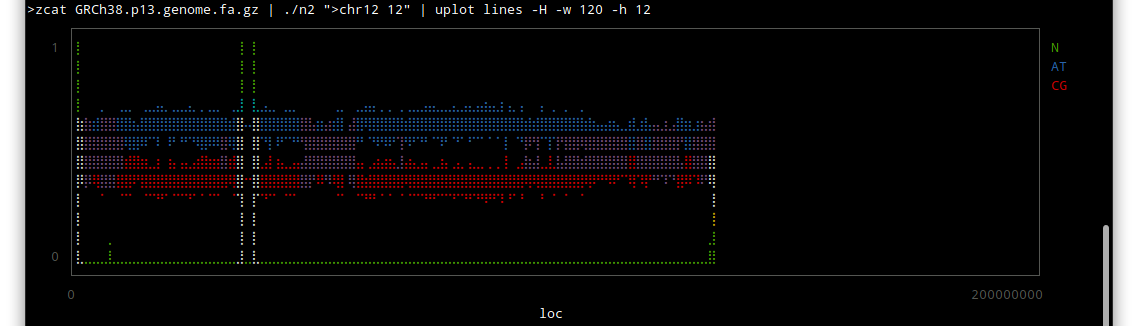
chr1
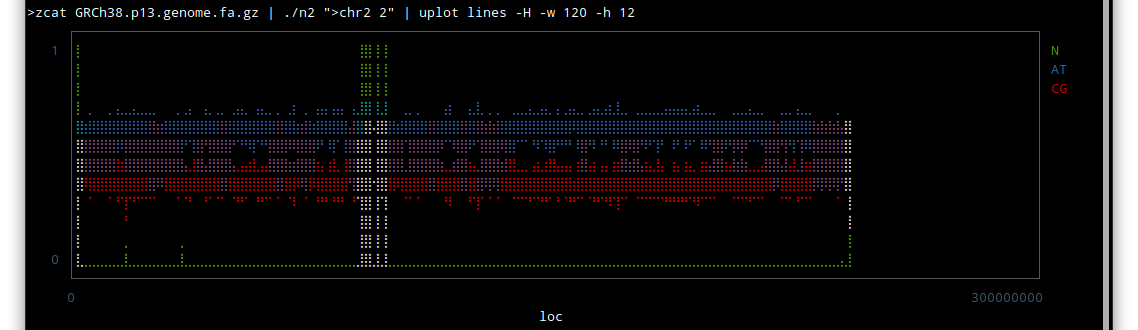
chr2
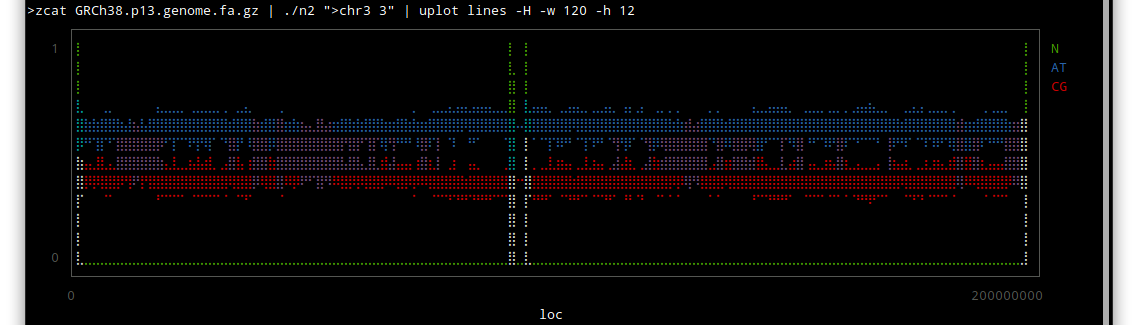
chr3
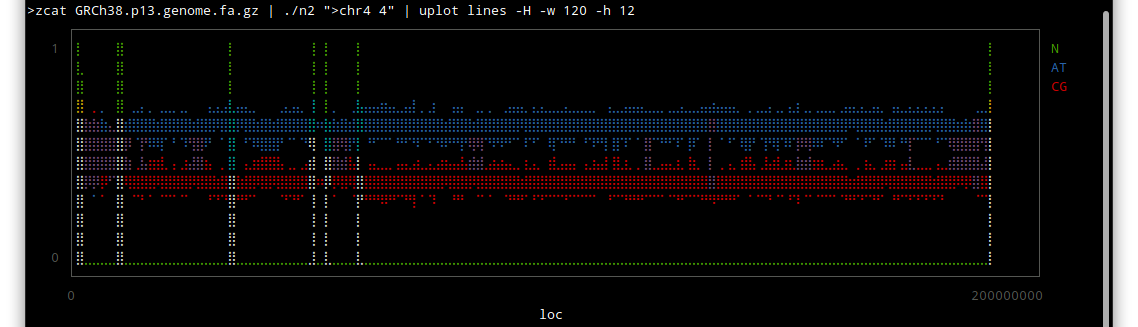
chr4
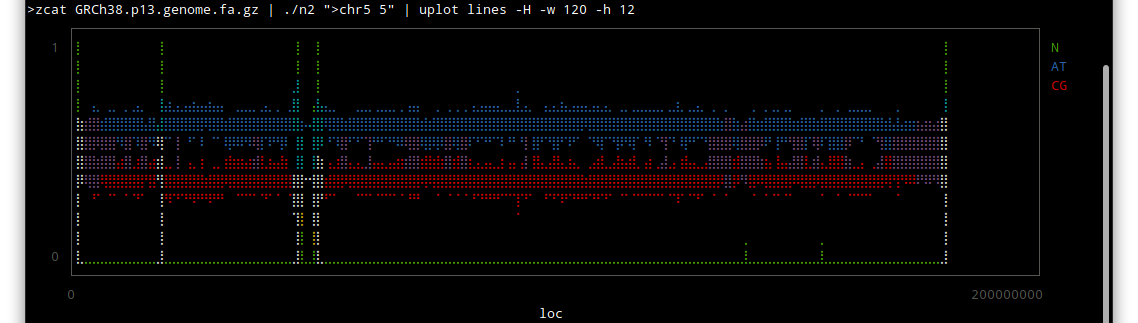
chr5
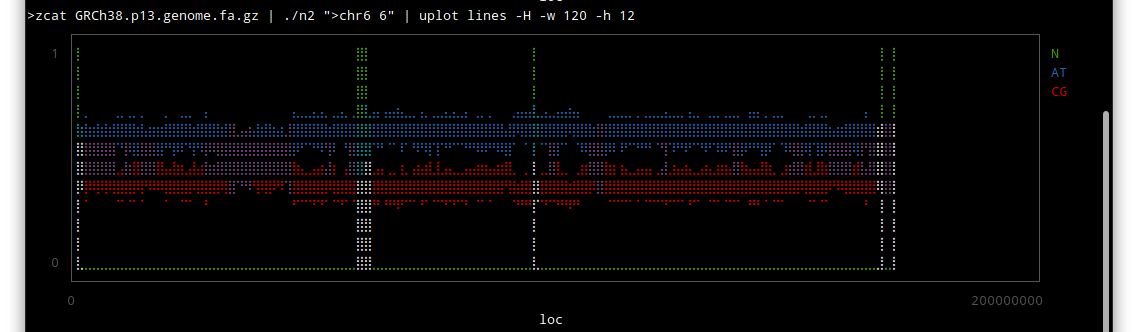
chr6
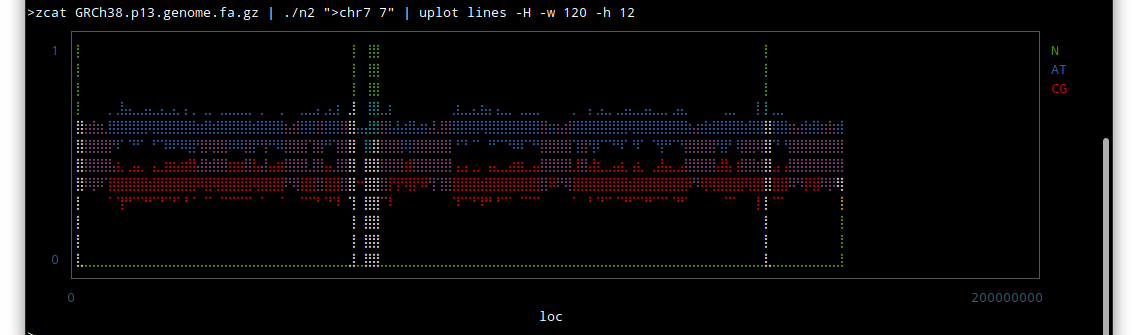
chr7
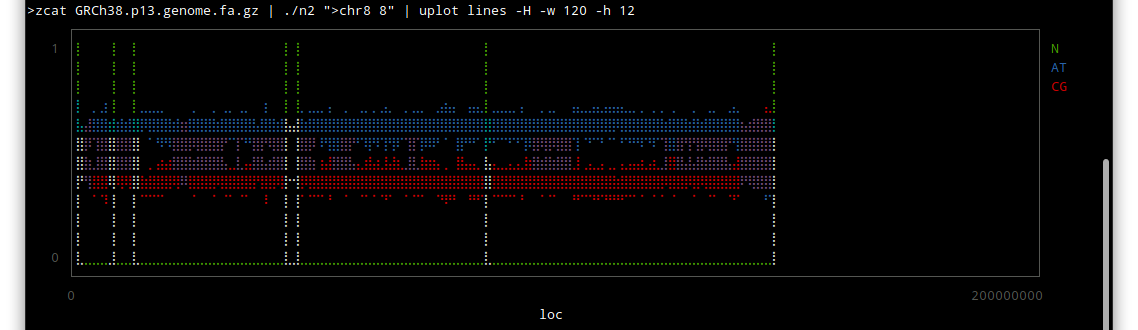
chr8
chr9
chr10
chr11
chr12
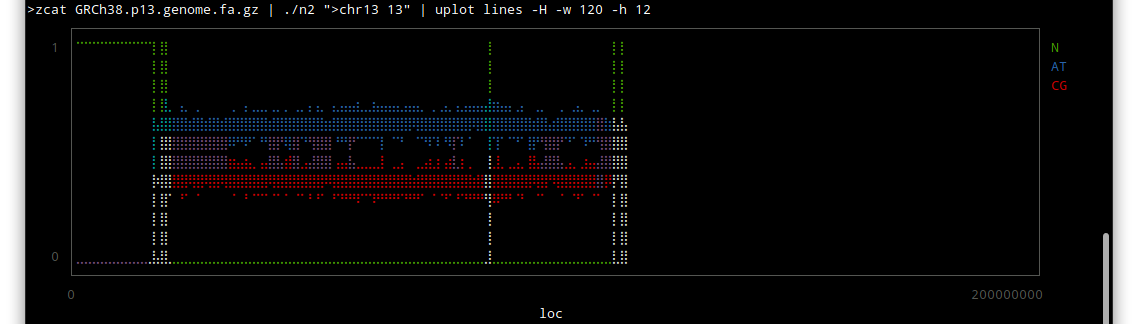
chr13
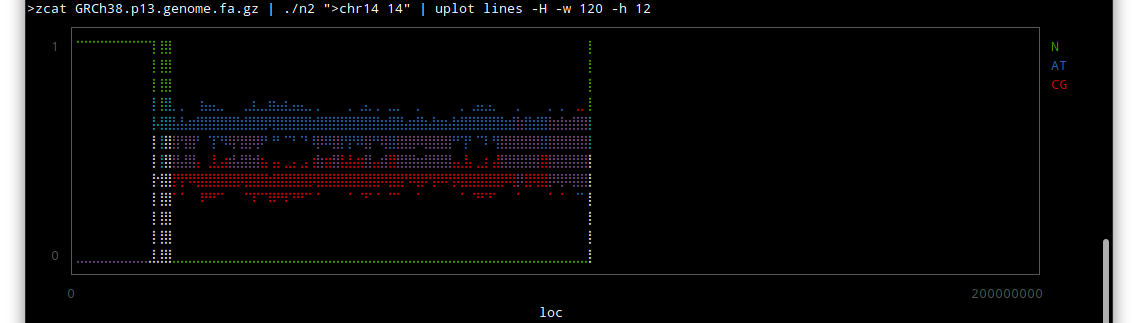
chr14
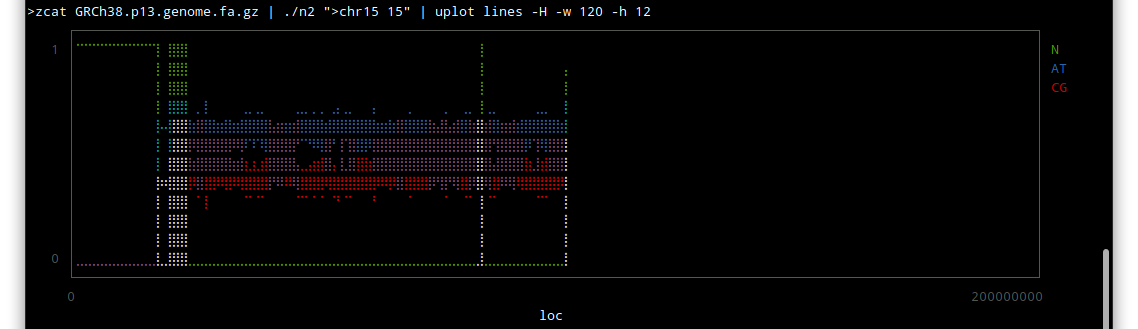
chr 15
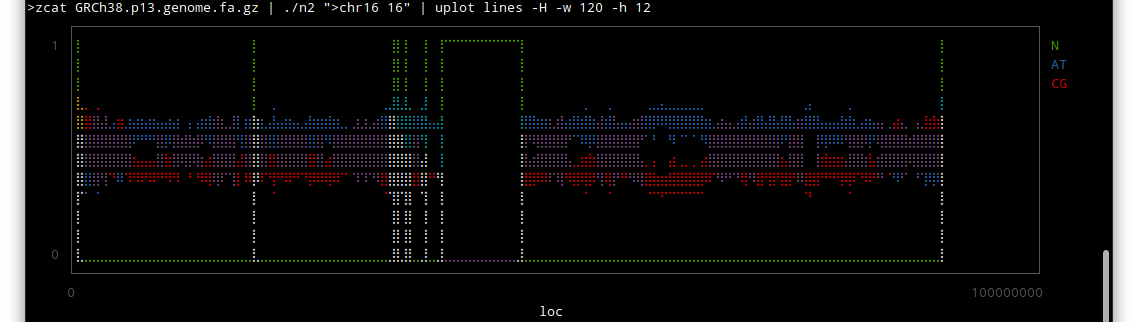
chr 16
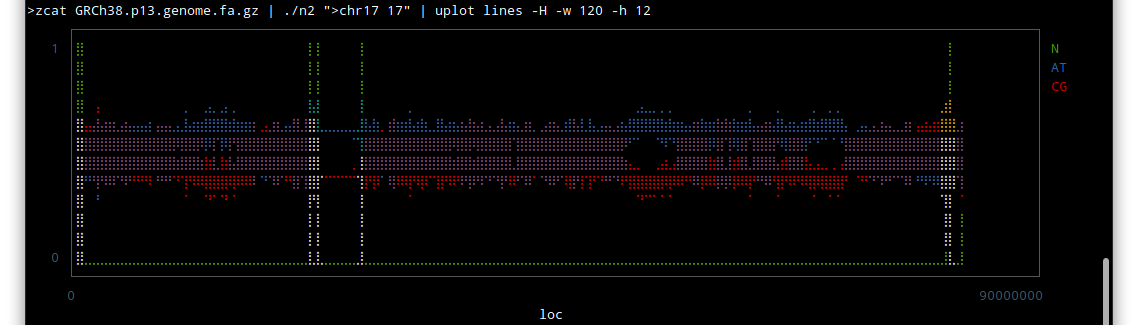
chr 17
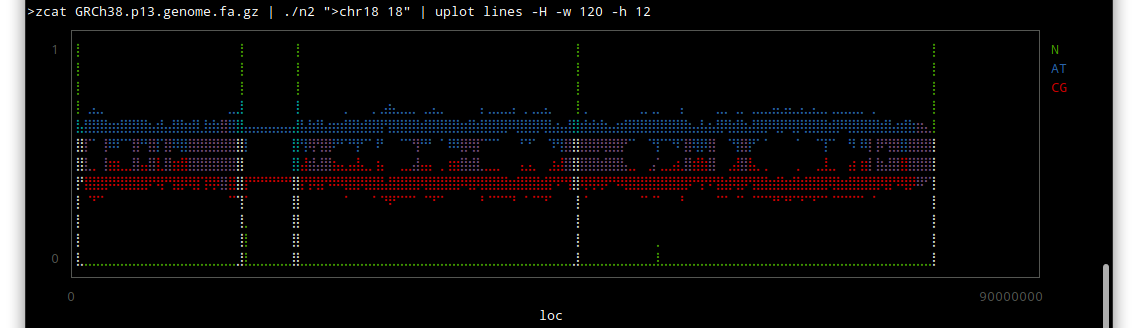
chr 18
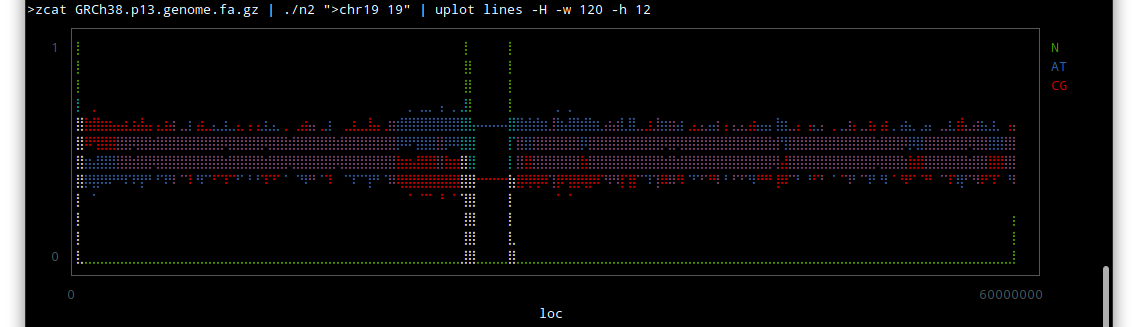
chr 19
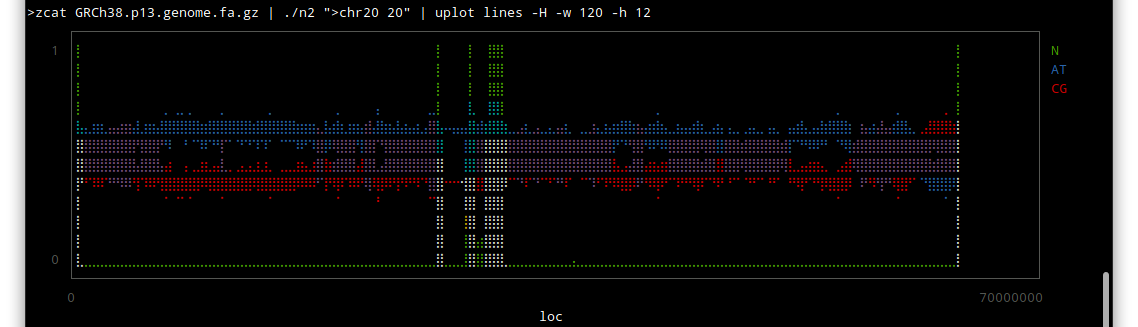
chr 20
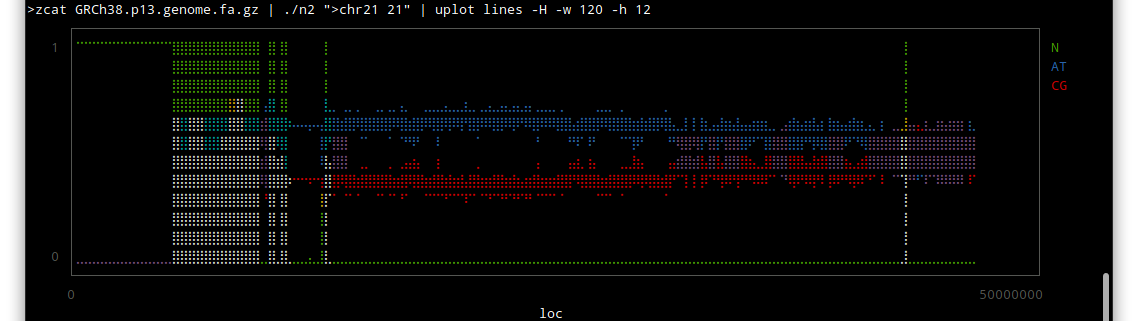
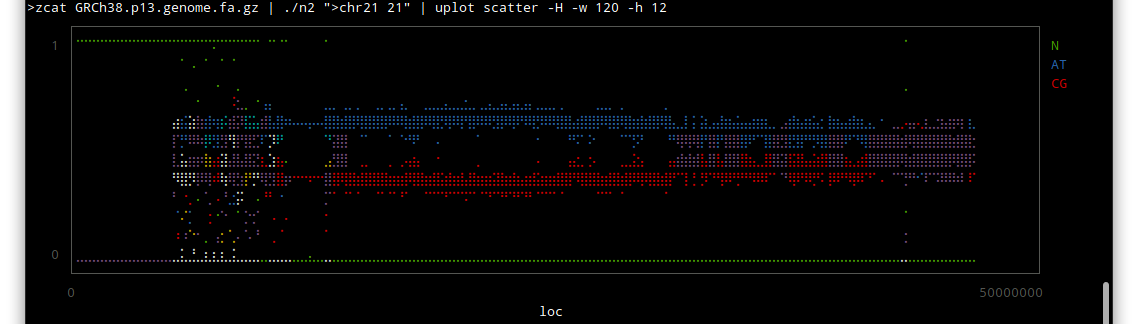
chr 21
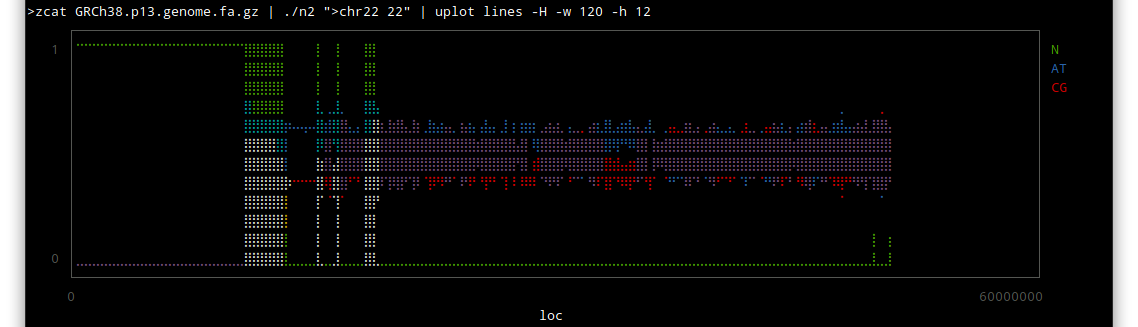
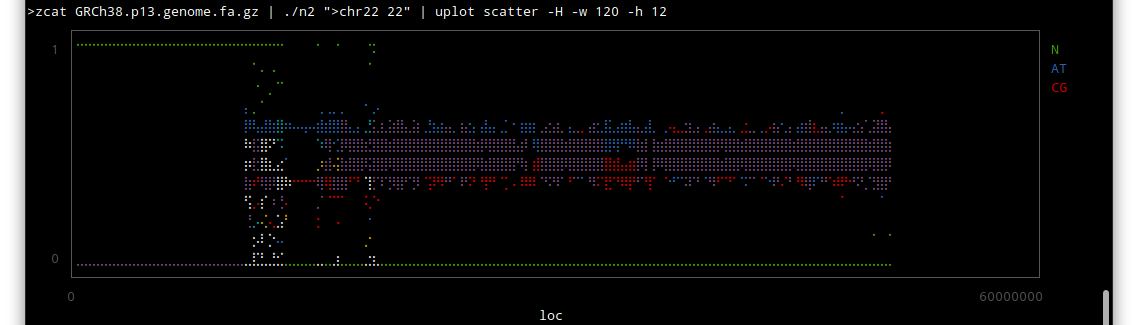
chr 22
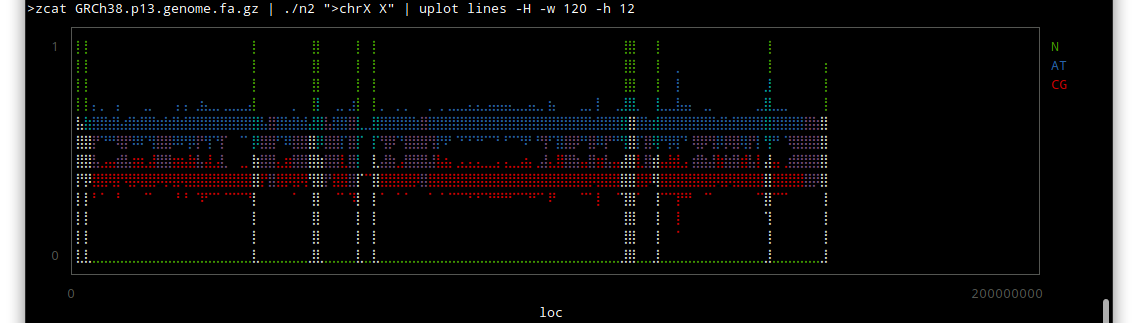
chr X
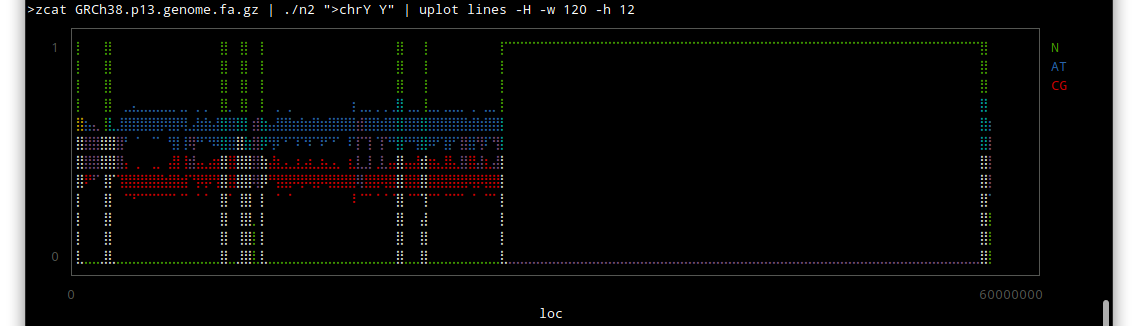
chr Y
Wikipediaによると、
主にヘテロクロマチン領域が未解読であり、その領域は活性がないそうです。転写とかほとんどされないという意味だと思います。
つまり生物学的にはあまり意味がないということなのかも知れません。
しかしこうやってみると、配列のわかっていない部分はずいぶんあるんだな〜という感じですね。
詳しい方とかいらっしゃいましたらご自由に突っ込んでください。
この記事は以上です。