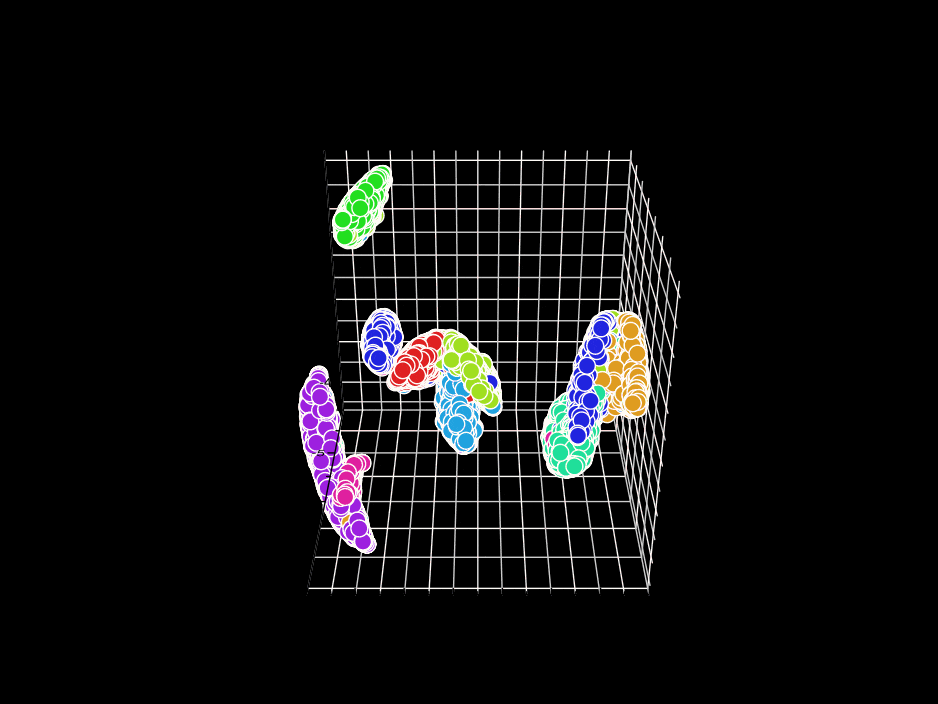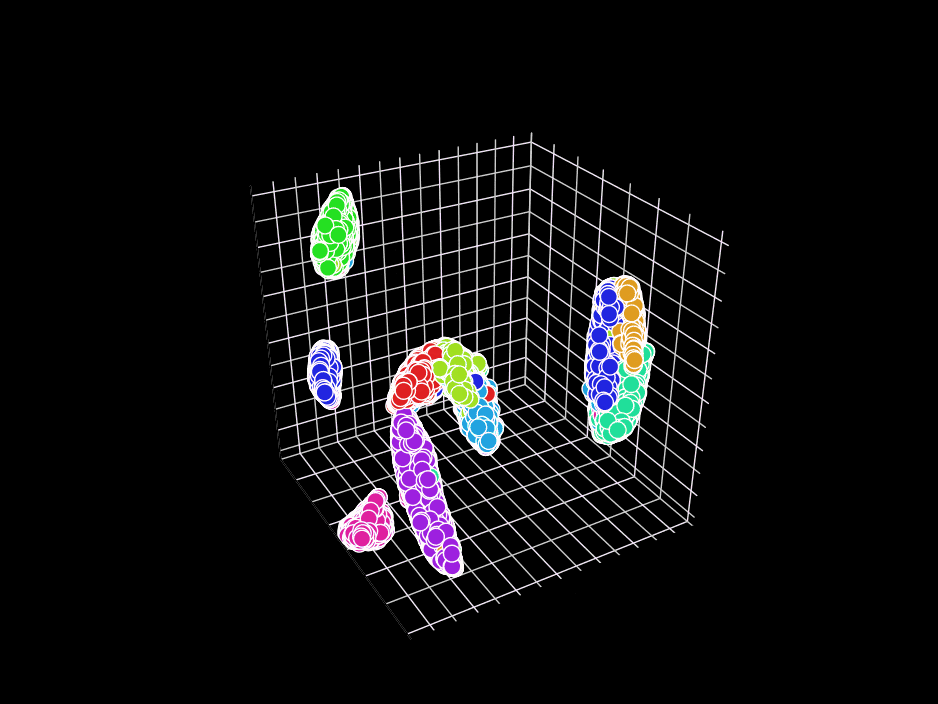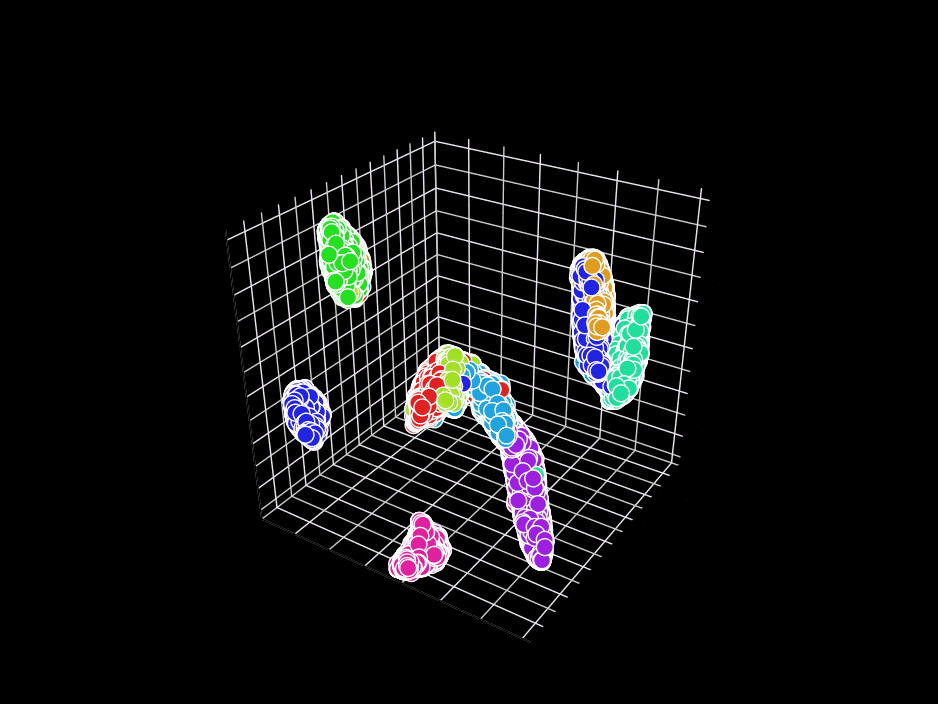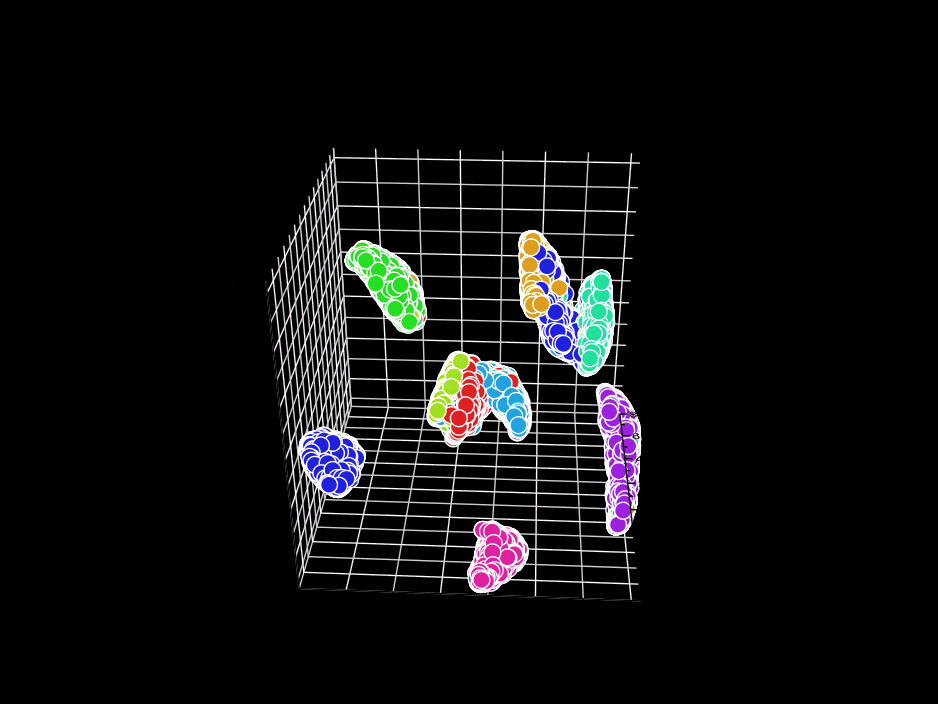はじめに
Uniform manifold approximation and projection (UMAP) はt-SNEとならんでよく使われる次元圧縮による可視化手法です。
Ruby言語で、機械学習を実行する場合、Rumaleというgemを使うケースが多いと思います。Rumaleにはt-SNEが搭載されていますが、UMAPは搭載されていません。
今回、C++のライブラリであるUmapppのRubyバインディングを作成したので忘れないうちに記録します。
Rubyのライブラリがないときはバインディングを作ろう
Ruby言語は、データ解析の分野では比較的マイナーな言語なので、やりたいことを実装したライブラリーが存在しないことがよくあります。そんな時はC言語やRust言語などのライブラリを探してRubyバインディングを作る方法があります。 GitHubの検索では、言語を指定してコードを検索できます。 これを使用して、C言語のライブラリーを見つけます。 GitHubではプロジェクトにタグを付けることができるので、対象のタグを確認することも効果的です。 ただ、UMAPは実装が難しいようで、UMAPを実装しているC言語ライブラリは見当たりませんでした。 代わりに、C++でUMAPを実装するライブラリを見つけました。それがUmapppです。
Umappp - UMAPのC++実装
Umapppは、Aaron Lun さんによって実装されたC++のライブラリです。Rのuwotを参考に開発されているようです。計測はしていませんが、C++で実装されOpenMPも利用しているため高いパフォーマンスが期待できます。
Ruby言語からC++の関数を呼び出す
R言語ではC++を使った拡張ライブラリは一般的ですが、Ruby言語ではC++による拡張ライブラリはそれほど広く利用されていません。最近はRustが人気で、Rubyの拡張機能をRustで作っている人をたまに見かけますが、新規のC++の拡張ライブラリはあまり見かけません。
しかし、RubyでC++のバインディングを作るのは思ったより簡単です。私はC++の経験がありませんが、RubyからC++を呼び出すことができました。
Rubyの拡張をC++で書く方法は2つあります。一つはRice、もう一つはextppです。今回はNumo::NArray とC++を連携するnumo.hppを使いたかったのでRiceを使用しました。
実行時にC++をコンパイルできるように、UmapppとUmapppの依存するすべてのC++のファイルをVendorディレクトリに入れてGemとして配布する方針にしました。extconf.rb の書き方がよくわからなかったので、他のプロジェクトを参考に調節しました。
Rice - C++拡張のためのRubyインターフェース
Riceは、Paul Brannanさん、Charlie Savageさん、 Jason Roelofsさんらによって開発された、RubyでC++拡張を書くためのライブラリです。Riceを使うことで、下記のようにC++のコードからRubyのモジュールやメソッドを定義することができます。
#include <rice/rice.hpp>
#include <rice/stl.hpp>
using namespace Rice;
Hash umappp_default_parameters(Object self)
{
Hash d;
//<中略>
return d;
}
//<中略>
extern "C" void Init_umappp()
{
Module rb_mUmappp =
define_module("Umappp")
.define_singleton_method("umappp_run", &umappp_run)
.define_singleton_method("umappp_default_parameters", &umappp_default_parameters);
}
RubyでUMAPを実行する
有名な irisデータセットと、MNISTデータベースに対してUMAPを実行します。
- ここでは自作の可視化ライブラリGR.rbを使って可視化しています。
- データの取得には red-datasets とその派生ライブラリの red-datasets-numo-narray を使いました。
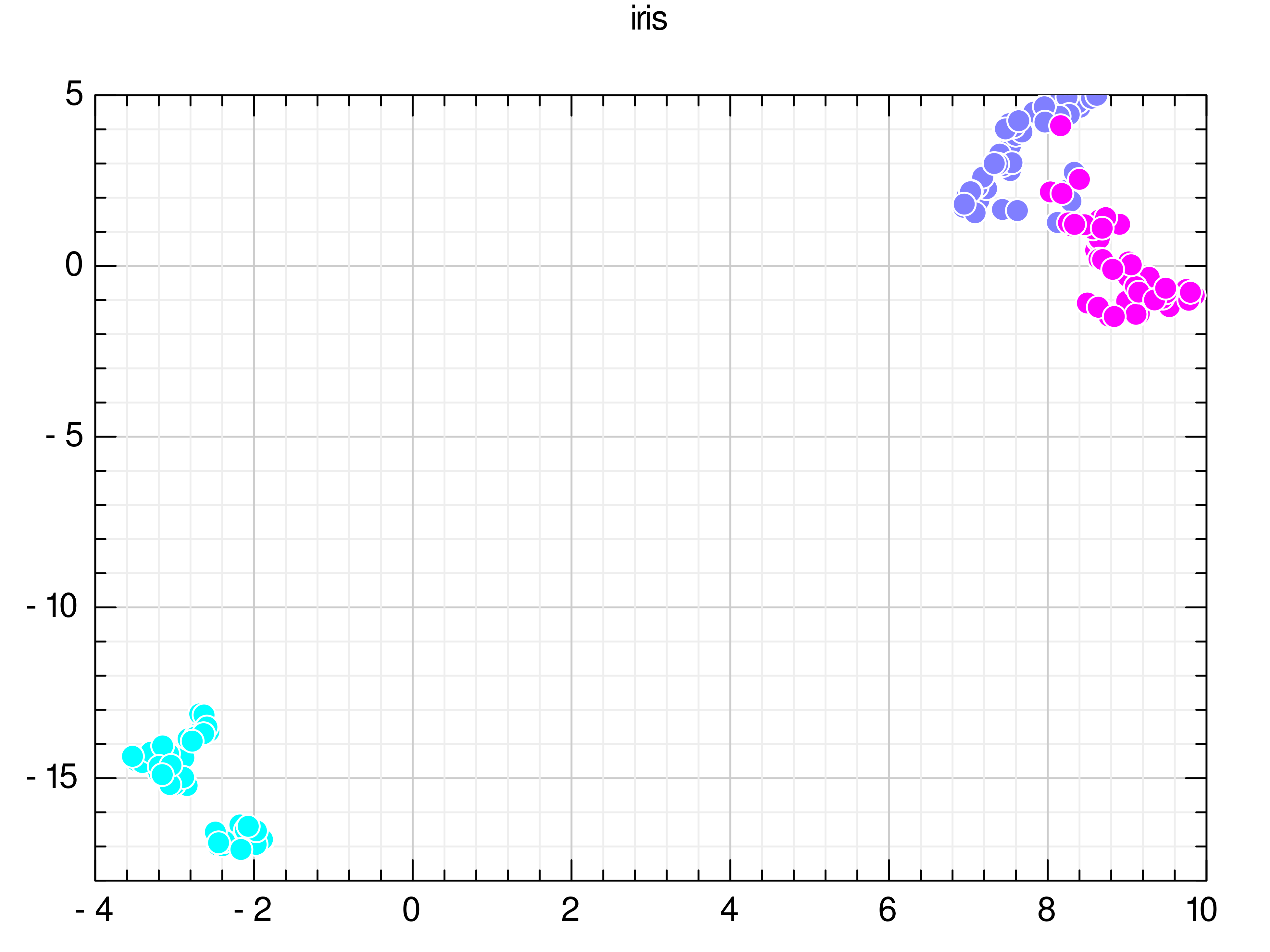
Irisデータセット
3種類のアヤメの花の、がく片(sepal)の長さ、幅、および花弁(petal)の長さ、幅を測定したデータセットです。

画像出典:https://machinelearninghd.com/iris-dataset-uci-machine-learning-repository-project/
Rubyのコード:
require "umappp"
require "datasets-numo-narray"
require "gr/plot"
iris = Datasets::LIBSVM.new("iris").to_narray
d = iris[true, 1..-1]
l = iris[true, 0]
r = Umappp.run(d)
x = r[true, 0]
y = r[true, 1]
s = [2000] * l.size
GR.scatter(
x, y, s, l,
title: "iris",
colormap: 16,
colorbar: true
)
gets
明確にクラスタが分かれました。ここでは、ラベル0(setosa)が水色、ラベル1(versicolor)が藤色、ラベル2(virginica)がマゼンタです。setosaのグループは他のグループと明瞭に分かれていますが、versicolorとvirginicaは一部重なっているのがわかります。
(余談)テーブルデータのGUI表示
Rubyでは、大きなサイズのテーブルデータをCUIで確認しにくい場合があります。そんな時にRedAmberというデータフレームと、そのGUI表示用の自作ツールred-amber-viewを使うと、テーブルデータをGUIで表示することもできます。
# irisデータセットをテーブルとして表示する
iris = Datasets::LIBSVM.new("iris")
dataframe = RedAmber::DataFrame.new(iris).view
# 結果をテーブルとして表示する
RedAmber::DataFrame.new(x: x.to_a, y: y.to_a, l: l.to_a).view
※これは作りかけなので、こんなこともできますよ、というぐらいの話です。
MNISTデータベース
次は手書き数字の画像データセットであるMNISTの次元削減をしてみます。
Rubyのコード:
require "umappp"
require "datasets"
require "gr/plot"
require "etc"
mnist = Datasets::MNIST.new
pixels = []
labels = []
mnist.each_with_index do |r, _i|
pixels << r.pixels
labels << r.label
end
puts "start umap"
nproc = Etc.nprocessors
n = nproc > 4 ? nproc - 1 : nproc
d = Umappp.run(pixels, num_threads: n, a: 1.8956, b: 0.8006)
puts "end umap"
x = d[true, 0]
y = d[true, 1]
s = [500] * x.size
GR.scatter(x, y, s, labels, colormap: 0)
gets
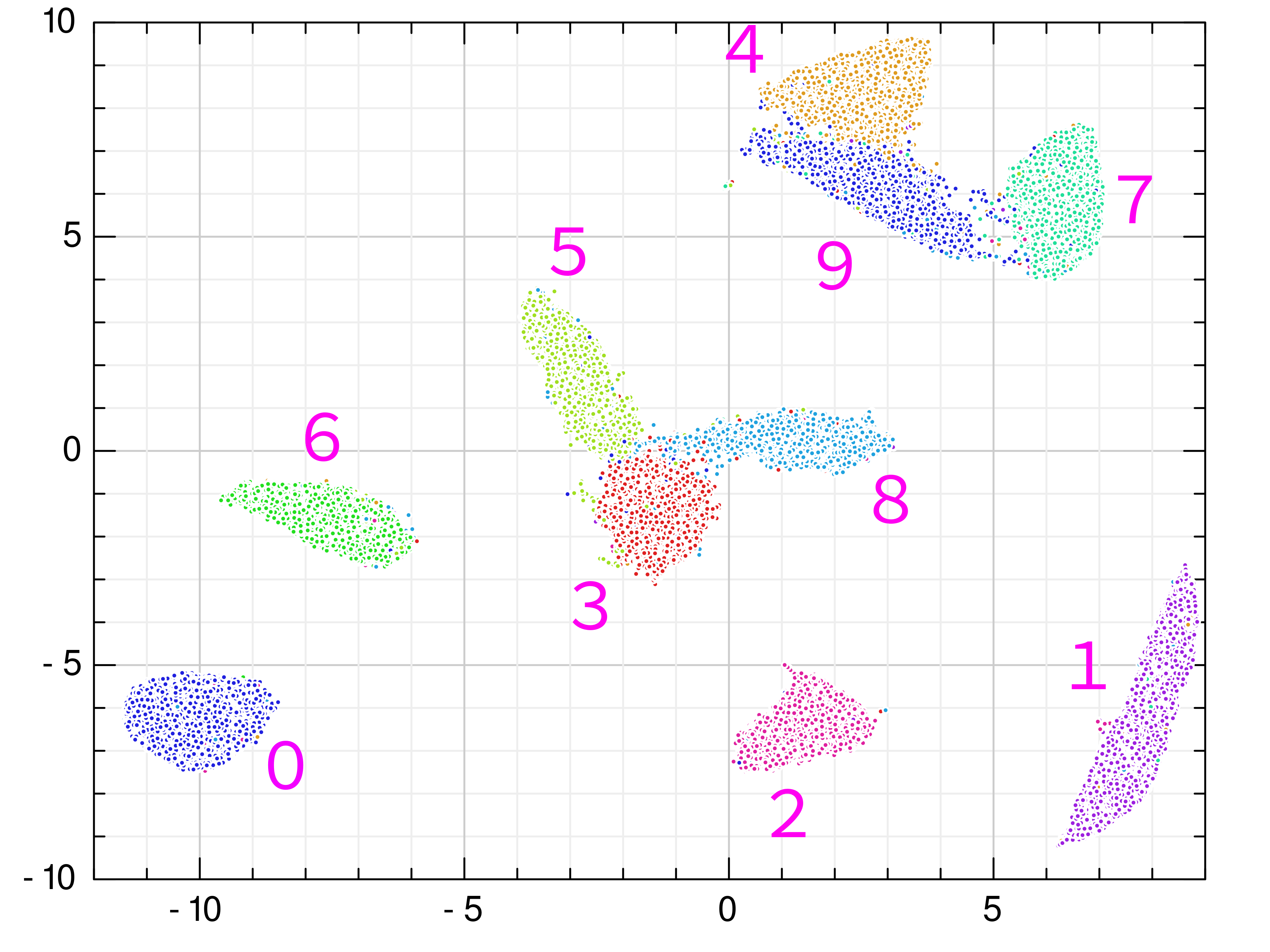
OpenMPをインストールしている環境では htop コマンドなどでCPUの使用率を監視すると、途中から多数のコアを使って計算しているのがわかると思います。非常にきれいにクラスタが分かれました。
GR.rbのカラーマップからラベルを比定すると、以下のようになります。これは、公式によるUMAPの結果とほぼ一致しておりUMAPが正しく実行できていることがわかります。5ー3ー8と、4ー9ー7が類似しているようです。4と9の手書き文字の類似は直感的にもわかりやすく、実際もっとも境界が接しています。私は1を7に似たように書いてしまう癖があるのですが、そういう人はあまりいないようですね。
3D表示に挑戦してみます。ここでは、ndim: 3 を指定します。GR.rb はGIFアニメ出力機能がついているのでそれを使います。
require "umappp"
require "datasets" # red-datasets https://github.com/red-data-tools/red-datasets
require "gr/plot" # GR.rb https://github.com/red-data-tools/GR.rb
require "etc"
mnist = Datasets::MNIST.new
pixels = []
labels = []
mnist.each_with_index do |r, _i|
pixels << r.pixels
labels << r.label
end
puts "start umap"
nproc = Etc.nprocessors
n = nproc > 4 ? nproc - 1 : nproc
d = Umappp.run(pixels, ndim: 3, num_threads: n, a: 1.8956, b: 0.8006)
puts "end umap"
x = d[true, 0]
y = d[true, 1]
z = d[true, 2]
GR.beginprint("mnist.gif")
30.times do |i|
GR.scatter3(x, y, z, labels, title: "mnist", colormap: 0, backgroundcolor: 1, rotation: i * 3)
end
GR.endprint
残念ながら、GR.rbでは、polymarker3d を順番に呼び出しているだけなので、本来は背後に来るべき点が手前に来たり、手前に来るべき点が背後にきたりしています。が、雰囲気は伝わると思います。2Dのときと位置関係の整合性が保たれているのが面白いですね。
反省点としては、GR.rbを使った可視化では、どの色の集合がどの数字に対応するのかわからない(colorbarのようなものが表示できない)ので、その点の改善が必要だなと思いました。
この記事を書いた人は数学が苦手で、UMAPの具体的な仕組みを全く理解していません。 なのでUMAPについてはうまいことは言えませんが、これだけ高速に動作するなら、気になるデータを調べるのにUMAPを使うと便利だなと思いました。 t-SNEよりかなり早く結果が出る印象です。また、UMAPの結果は思ったよりも再現性があるんだなと感じました。
おわりに
RubyからC++を呼び出すことにより、RubyからUMAPを使うことができるようにしました。私の知る限りでは、バインディングも含めてRubyでUMAPを実行できるライブラリはこれがはじめてです。こうしてまた一つ、Rubyでできることが増えました。
【PR】
Rubyでデータ解析をツールを作成したい方は red-data-tools というコミュニティがあります。基本的には個人で開発しているのですが、実装上で困っていることや、実現したいけどやり方がわからないアイディアがあったら相談に乗れる場合がありますので、ぜひお立ち寄りください。
この記事は以上です。