QIIME 2のデータからPCoAを描画したい.
QIIME 2 のデフォルトでもPCoAプロット書けるけど,emperor だしけっこう平面図にすると見づらい.
できれば,RあたりでおしゃれにPCoAをプロットしたい.
1. データを用意
今回は,QIIME 2 の"Moving Pictures" tutorialのデータを使って描画してみる.
・Metadata: sample_metadata.tsv
・shannon diversity: shannon_vector.qza
・Unweighted UniFrac PCoA: unweighted_unifrac_pcoa_results.qza
以上の3ファイルを使用する.
PCoAには通常,UniFrac 距離のデータが必要だが,ここではカテゴリ分け, shannon 多様性指数によるプロットサイズの変更も同時にしていきたい.
メタデータはMoving Pictures sample-metadata (QIIME 2 2018.4) - sample-metadata.tsvというファイル名になるので,sample-metadata.tsvに変更しておく.
2. PCoAの描画を実行
以下はRで実行していく.R のバージョンは以下の通り.
$ R --version
R version 3.5.1 (2018-07-02) -- "Feather Spray"
Copyright (C) 2018 The R Foundation for Statistical Computing
Platform: x86_64-apple-darwin15.6.0 (64-bit)
R is free software and comes with ABSOLUTELY NO WARRANTY.
You are welcome to redistribute it under the terms of the
GNU General Public License versions 2 or 3.
For more information about these matters see
http://www.gnu.org/licenses/.
Rは以下から.
# ライブラリの読み込み
library(tidyverse)
devtools::install_github("jbisanz/qiime2R")
library(qiime2R)
# 各種データの読み込み
metadata<-read_tsv("sample-metadata.tsv")
shannon<-read_qza("shannon_vector.qza")
pco<-read_qza("unweighted_unifrac_pcoa_results.qza")
# PCoAの描画
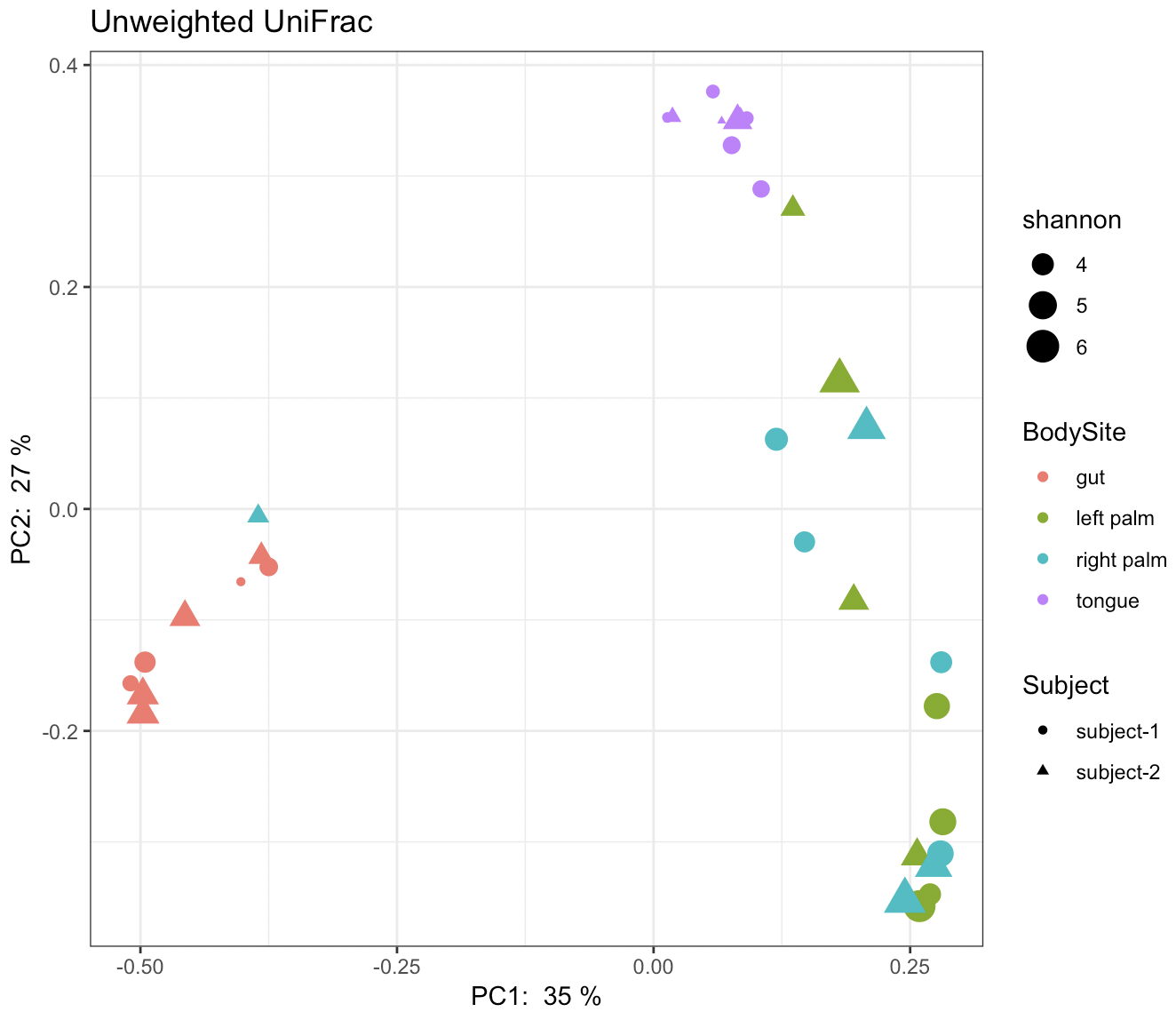
pco$data$Vectors %>%
rename("#SampleID"=SampleID) %>% #rename to match the metadata table
left_join(metadata) %>%
left_join(shannon$data %>% rownames_to_column("#SampleID")) %>%
ggplot(aes(x=PC1, y=PC2, shape=Subject, color=BodySite, size=shannon)) +
geom_point() +
xlab(paste("PC1: ", round(100*pco$data$ProportionExplained[1]), "%")) +
ylab(paste("PC2: ", round(100*pco$data$ProportionExplained[2]), "%")) +
theme_bw() +
ggtitle("Unweighted UniFrac")