微生物の表現型を予測できるTraitar を使ってみよう
少しだけ自己紹介
現在,慶應義塾大学の環境情報学部に在学している学部3年生です.
現在は次世代シーケンサでMicrobiome解析とか,公共データベース使ってゲノム比較解析などをメインで行っています.
AT Advent Calendar 2017 用のネタです.ぜひAT勢のみなさん,取り組んでみてください!!!
Traitar とは
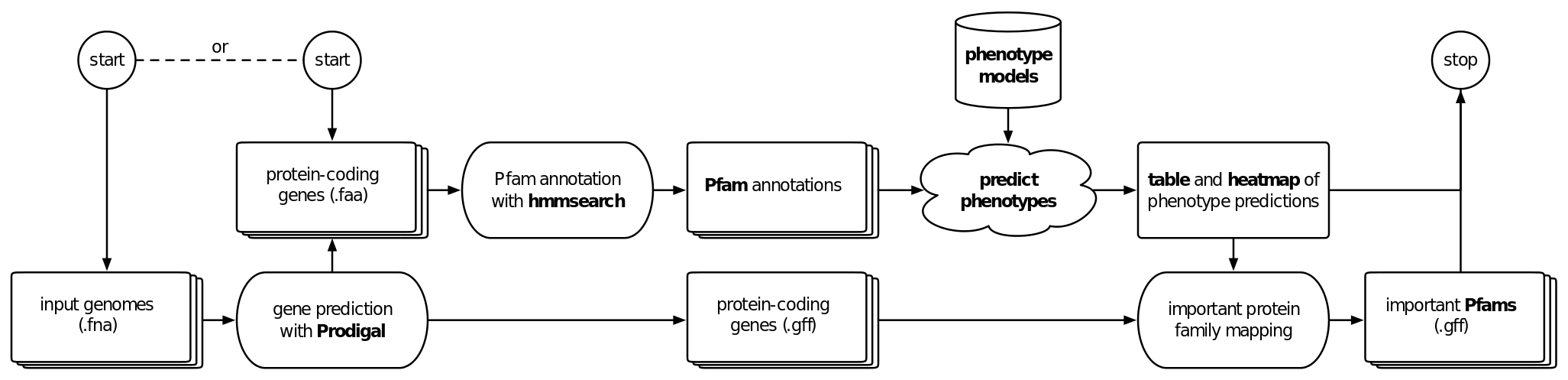
Traitar(Aaron Weimann et al,. 2016) は,ヌクレオチドまたはタンパク質配列から微生物の表現型(67種類)を予測してくれる便利なツール.
Traitarの解析に必要なゲノムデータ
今回は,ヒト常在菌である
Propionibacterium acnes SK137 株
Staphylococcus aureus RF122 株
Staphylococcus epidermidis ATCC 12228 株
最近また話題になっている O157 の仲間である病原菌
Escherichia coli O157:H7 str. Sakai 株
を用いて表現型を予測してみます。
1. 必要なゲノムデータを公共データベースからダウンロードする
今回は,NCBIという大手のゲノムデータベースから4株のゲノムをダウンロードしていく.
# HomeBrew がインストールされていない場合
/usr/bin/ruby -e "$(curl -fsSL https://raw.githubusercontent.com/Homebrew/install/master/install)"
# wget がインストールされていない場合
brew install wget
# ゲノムデータのダウンロード
# Propionibacterium acnes SK137
wget ftp://ftp.ncbi.nlm.nih.gov/genomes/all/GCF/000/025/765/GCF_000025765.1_ASM2576v1/GCF_000025765.1_ASM2576v1_genomic.fna.gz
# Staphylococcus aureus RF122
wget ftp://ftp.ncbi.nlm.nih.gov/genomes/all/GCF/000/009/005/GCF_000009005.1_ASM900v1/GCF_000009005.1_ASM900v1_genomic.fna.gz
# Staphylococcus epidermidis ATCC 12228
wget ftp://ftp.ncbi.nlm.nih.gov/genomes/all/GCF/000/007/645/GCF_000007645.1_ASM764v1/GCF_000007645.1_ASM764v1_genomic.fna.gz
# Escherichia coli O157:H7 str. Sakai
wget ftp://ftp.ncbi.nlm.nih.gov/genomes/all/GCF/000/008/865/GCF_000008865.1_ASM886v1/GCF_000008865.1_ASM886v1_genomic.fna.gz
# ファイルの解凍
gunzip *.gz
# fnaディレクトリの作成
mkdir fna
# fna ファイルの移動
mv *.fna fna/
2.Traitar とその依存ツールのインストールする
Traitar はPython のモジュールであり,pip インストールでインストールできる.
また依存するツールは以下の通り
・python-scipy
・python-matplotlib
・python-pip
・python-pandas
・paralell
・prodigal
・hmmer
# Python のバージョン
python -V
# >> Python 2.7.13
# pip のインストール
brew install pip
# scipy, matplotlib, pandas のインストール
pip install scipy matplotlib pandas
# parallel, prodigal, hmmer のインストール
brew tap homebrew/science
brew install parallel prodigal hmmer
# Traitar のインストール
pip install traitar
3. Pfam データベースをダウンロードする
Traitar はPfam データベースを用いてアノテーションを行う.
そのため,Pfam27.0のデータベースをダウンロードする必要がある.
# Pfam データベースは以下からダウンロード可能.
wget ftp://ftp.ebi.ac.uk/pub/databases/Pfam/releases/Pfam27.0/Pfam-A.hmm.gz
# ディレクトリの作成
mkdir Pfam27.0
# Pfam モデルのファイルの移動
mv Pfam-A.hmm.gz Pfam27.0
# Pfam モデルのファイルの解凍
gunzip Pfam27.0/Pfam-A.hmm.gz
# traitar にパスを読み込ませる
traitar pfam --local $PWD/Pfam27.0
4. Traitar を実行する
環境構築が済んだらTraitar を実行.
samples.txt に記載されたゲノムデータの塩基配列(${in_dir}ディレクトリ内のFASTA形式ファイル)に対して,Prodigal でORFを予測し,Pfam データベースへのアノテーションをhmmer で行い,表現型(67種類)を予測する.
# samples.txt を作成する
vi samples.txt
# samples.txtの中身 (space はtabに変換, categoryの1はグラム陽性菌,2はグラム陰性菌)
sample_file_name sample_name category
GCF_000025765.1_ASM2576v1_genomic.fna Propionibacterium_acnes_SK137 1
GCF_000009005.1_ASM900v1_genomic.fna Staphylococcus_aureus_RF122 1
GCF_000007645.1_ASM764v1_genomic.fna Staphylococcus_epidermidis_ATCC12228 1
GCF_000008865.1_ASM886v1_genomic.fna Escherichia_coli_O157_H7_str.Sakai 2
ではようやくTraitarの実行
# Traitar の実行
in_dir=fna
out_dir=results
traitar phenotype ${in_dir} samples.txt from_nucleotides ${out_dir} -c $(getconf _NPROCESSORS_ONLN)
5. 実行結果を確認する
出力結果は,${out_dir} に格納される.
# Pfam のアノテーション
${out_dir}/annotation
# 遺伝子の予測
${out_dir}/gene_prediction
# 表現型の予測
${out_dir}/phenotype_prediction
・ Pfam のアノテーション
# 表現型とサンプルのクラスタリング樹形図を示すヒートマップ
${out_dir}/phenotype_prediction/heatmap_phypat.pdf
${out_dir}/phenotype_prediction/heatmap_phypat+PGL.pdf
${out_dir}/phenotype_prediction/heatmap_combined.pdf
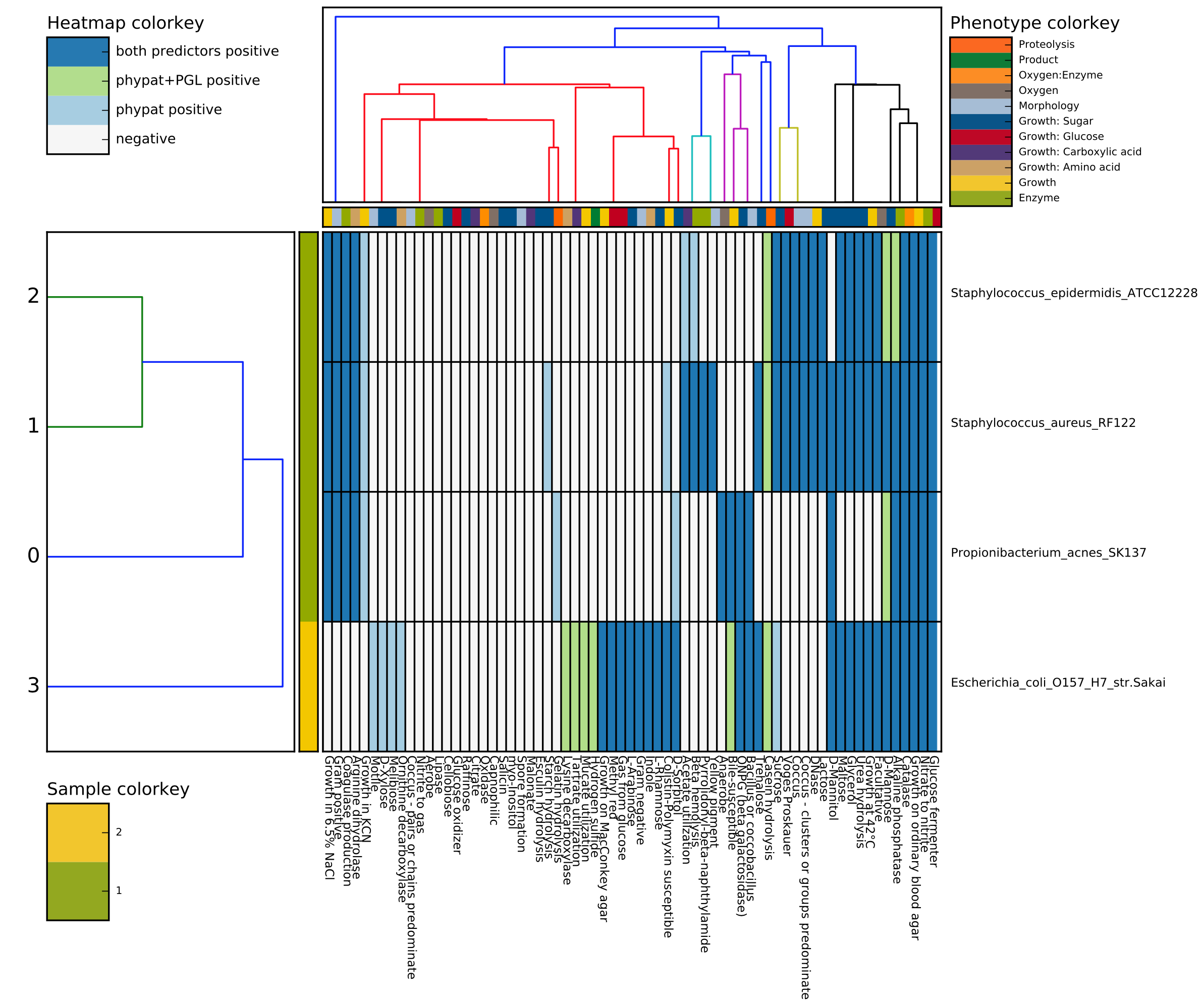
・ 出力結果のHeatMap(heatmap_combined.pdf)
・Heatmap cokorkey
白,水色,緑色,青色の4色があり,各表現型がなんのアルゴリズムで予測されたかを示す.
白:両アルゴリズムで予測されなかった
水色:phypat で予測された
緑色:phypat, PGL で予測された
青色:両アルゴリズムで予測された
・Sample colorkey
Samples.txt の3列目を反映しており,今回は
1: グラム陽性菌
2: グラム陰性菌
とした.
・ 表現型予測テキストファイル
# 表現型予測のテキストファイル
${out_dir}/phenotype_prediction/predictions_majority-vote_combined.txt
${out_dir}/phenotype_prediction/predictions_flat_majority-votes_combined.txt
# 各数値の説明
0 = negative prediction
1 = prediction made only by the pure phyletic classifier
2 = prediction made by the phylogeny-aware classifier
3 = prediction supported by both algorithms
# 両アルゴリズム(phypat, phypat+PGL) の出力結果
${out_dir}/phenotype_prediction/phypat/
${out_dir}/phenotype_prediction/phypat+PGL/
エラーの対処
Traitarは2016年から更新が止まっているツールなので、Pythonも含めた依存ツールのバージョンに注意が必要。
2020年12月にcondaを用いてpython2.7の環境を構築し実行を試みたところ、以下のpandasのエラーがでた。
AttributeError: 'DataFrame' object has no attribute 'sort'
どうやら、pandasがsort()をバージョン0.20にて削除したらしい。
'DataFrame' object has no attribute 'sort'
sort() was deprecated (but still available) in Pandas with release 0.17 (2015-10-09) with the introduction of sort_values() and sort_index(). It was removed from Pandas with release 0.20 (2017-05-05).
そのため、pandasのバージョンをダウングレードしたら実行できた。
pip install pandas==0.17
Traitar論文の引用
Traitar の論文引用は以下から.
From Genomes to Phenotypes: Traitar, the Microbial Trait Analyzer
Weimann A, Mooren K, Frank J, Pope PB, Bremges A, McHardy AC. From Genomes to
Phenotypes: Traitar, the Microbial Trait Analyzer. mSystems. 2016 Dec 27;1(6).
pii: e00101-16. doi: 10.1128/mSystems.00101-16. eCollection 2016 Nov-Dec.
参考文献
・Traitar – the microbial trait analyzer
https://github.com/hzi-bifo/traitar
・Traitar Tutorial Project
https://github.com/haruosuz/mgsa/tree/master/traitar