Qiita はオミクスデータを解析・管理できるプラットフォーム
Qiita (canonically pronounced cheetah) is a software package intended for analysis and administration of multi-omics datasets.
Qiita はオープンソースのマイクロバイオームを主としたオミクスデータを解析し管理できるプラットフォームです.
2018年にQIIME を開発しているRob Knightらのチームによってnature methods誌にて論文が発表されました.
ちなみに読み方は 「チーター (cheetah)」 らしいです.
Qiitaはデータベースや計算資源を提供してくれるので,マシンパワーなどハードウェアの制限,コマンドラインの知識などソフトウェアの制限による負担などを軽減できます.
また,マイクロバイオーム研究では様々な実験手法や解析パイプラインが用いられるため,再現性の担保が難しいとされますが,Qiitaでは複数研究のデータを統合し再解析することによって,再現性のある一貫した特徴を導き出すこともできます.
Qiitaは,ユーザーに以下の無料のオープンプラットフォームを提供します.
- 実験形式で既存のデータセットを簡単に共有して再利用.
- 公開データや未公開データを使用して,複数の研究やデータセットで解析を実行.
- 簡単なインターフェースでEBIへのデータデポジットが自動.
利用登録する
まずはアカウントを作成します.
以下に必要な情報を入力し,「Create User」をクリックします.

メールが来るので,メール内のリンクに飛びアカウントをアクティベートします.
サンプルデータを手に入れる
今回は,CMI Qiita/GNPS workshop のデータを用いて進めていきます.
Dropbox リンクからファイルごとダウンロードしてください.
Studyを作成する
ホーム画面の「Study」から「Create Study」を選択します.
「Principal Investigator」は既存のPIリストから選択しますが,リストにない場合がほとんどだと思うので,「Add a person」で追加します.

Studyを作成するのに必要な情報は以下です.
-
Study Title (required): 実験名
-
Study Alias (required): 実験の別名
-
Study Abstract (required): 実験の要旨
-
Study Description (required): 実験の短い説明
-
Principal Investigator (required): ラボ主宰者名
サンプル情報を追加する
サンプル情報は,16Sやメタゲノムなど生物学的サンプルに関連するメタデータのデータセットであり,マイクロバイオーム研究でこのメタデータはとても重要です.
一貫性があり,正確かつ詳しく記載されていることが必須であり,マイクロバイオーム解析の結果を大きく左右します.
今回はサンプルデータを用いますが,自分で用意したデータを用いる場合には十分注意して作成にあたってください.
「View Studies」から先程作成したStudyを選択してください.
この画面にはStudy の管理を行うことができます.
「Upload Files」をクリックし,先程ダウンロードしたディレクトリ内のsample_information.txtをアップロードします.
終わったら前の画面に戻ります.
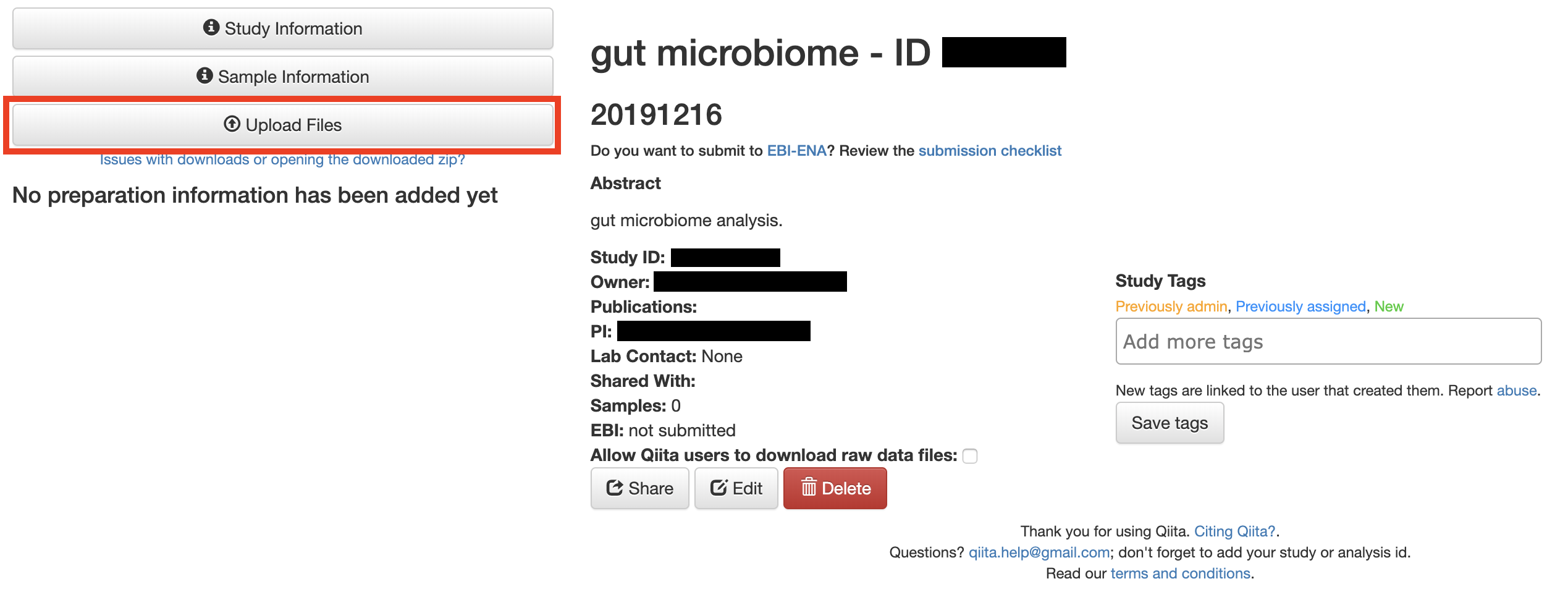
先程の「Upload Files」の一つ上にある「Sample Information」をクリックします.
そこで,「Select sample information file:」で今アップロードしたsample_information.txtを選択します.
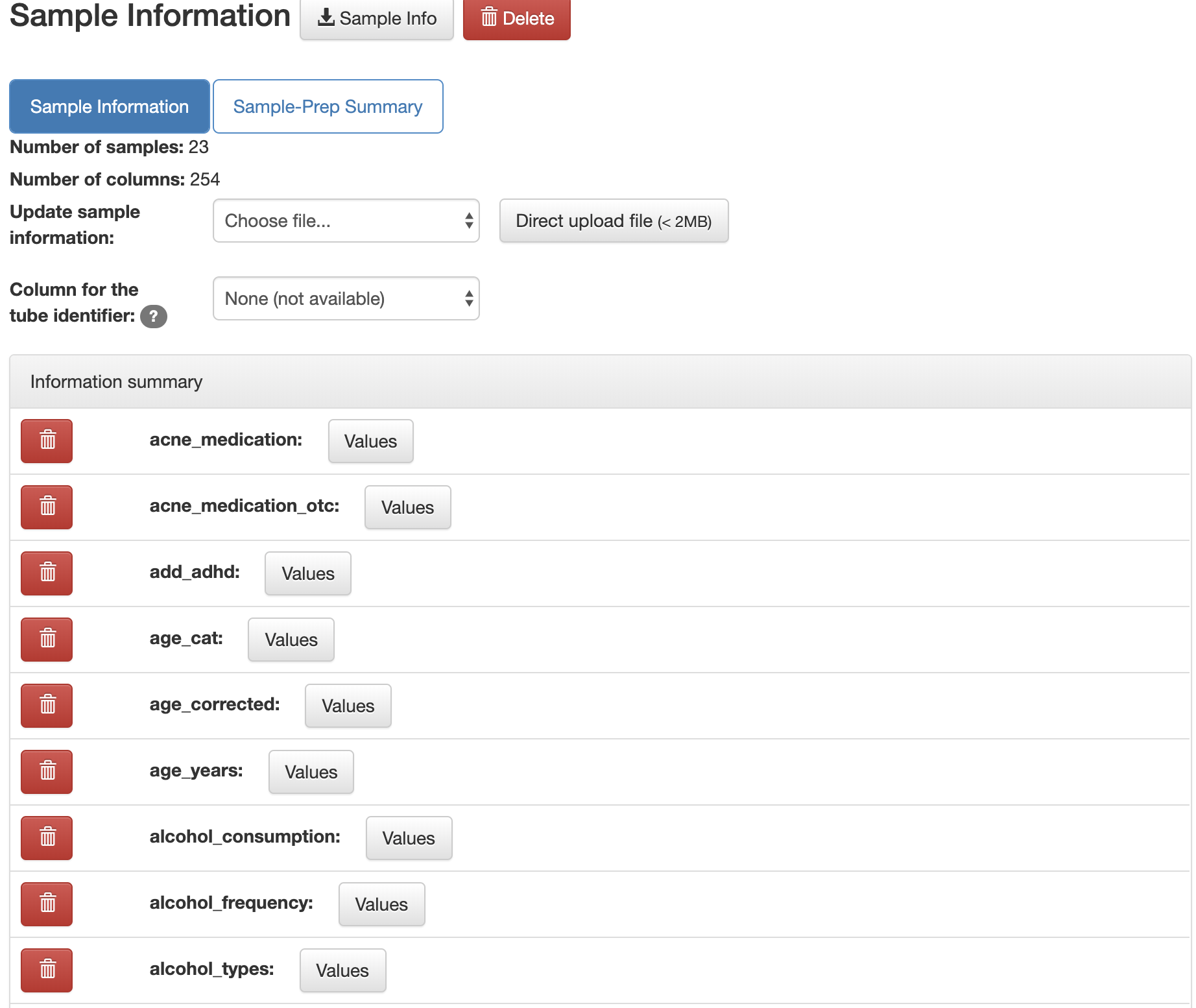
うまくいくと「Sample Information」をクリックするとアップロードしたメタデータの項目が確認できます.
次回は16Sアンプリコン配列データを解析していきます.