モチベーション
分布を調べることはデータ分析の基本的な手順のひとつ。しかし1次データが常に手に入るとは限らない。分位数データしか手に入らないことも多いはず。分位数のデータから分布を予測する方法をメモ代わりに記しておきたい。
パッケージインストールとデータ読み込み
今回はOptim.jlを使ってみる。
using Distributions,StatsPlots,Statistics,DataFrames,CSV,NamedArrays
using Turing,Optim
次にデータを用意。
realdist=Normal(2.3,1.3)
ss = rand(realdist,200);
qs =[0.025,0.10,0.35,0.55,0.75,0.95]
xs = quantile(ss,qs)
知っているデータは下記のQunatileのみと仮定する。
1×6 Named LinearAlgebra.Adjoint{Float64, Vector{Float64}}
A ╲ B │ 0.025 0.1 0.35 0.55 0.75 0.95
─────────┼─────────────────────────────────────────────────────────────────
Quantile │ -0.371107 0.416936 1.85021 2.64672 3.30508 4.56924
モデルを作成
Turingで使われるDynamicPPLでモデルを作成する。
@model function demo(xs,qs)
m ~ Normal(0,3)
s ~ InverseGamma(2,3)
σ ~ Exponential(1) #収束しやすくなる
dist = Normal(m,s)
xs .~ arraydist([Normal( quantile(dist,q), σ) for q in qs])
end
作成したモデルにデータを渡してMAP()を使って推定を行う。
model=demo(xs,qs)
optmap=optimize(model,MAP())
最適化されたパラメータを出力してもらえる。Turing+Optimの最適化はデフォルトではLBFGSが使われる。分布によっては微分ができない場合があるので、その場合はNelderMeadのようなGradient Freeな最適化関数を試すとよい。
もちろん、MLEでの推定もできる。
Julia> optmle=optimize(model,MLE())
ModeResult with maximized lp of 6.31
3-element Named Vector{Float64}
A │
───┼──────────
:m │ 2.34521
:s │ 1.39969
:σ │ 0.0845965
パラメータの確信区間を計算したい場合もあるだろう。Optimの出力をTuringへ渡して確信区間をサンプリングすることができる。
init_parameters=にoptimizeからの出力を渡す。出力はNamedArrayなのでデータarrayだけ渡すようにすること。
Julia> c1 = sample(demo(xs,qs),NUTS(),3000,init_parameters=optmle.values.array)
Summary Statistics
parameters mean std naive_se mcse ess rhat ⋯
Symbol Float64 Float64 Float64 Float64 Float64 Float64 ⋯
m 2.3462 0.0726 0.0013 0.0018 1713.4919 1.0021 ⋯
s 1.3979 0.0631 0.0012 0.0013 1308.9196 1.0004 ⋯
σ 0.1537 0.0962 0.0018 0.0038 650.6264 1.0003 ⋯
1 column omitted
Quantiles
parameters 2.5% 25.0% 50.0% 75.0% 97.5%
Symbol Float64 Float64 Float64 Float64 Float64
m 2.2066 2.3082 2.3468 2.3839 2.4924
s 1.2794 1.3666 1.3972 1.4280 1.5078
σ 0.0656 0.1006 0.1291 0.1746 0.3912
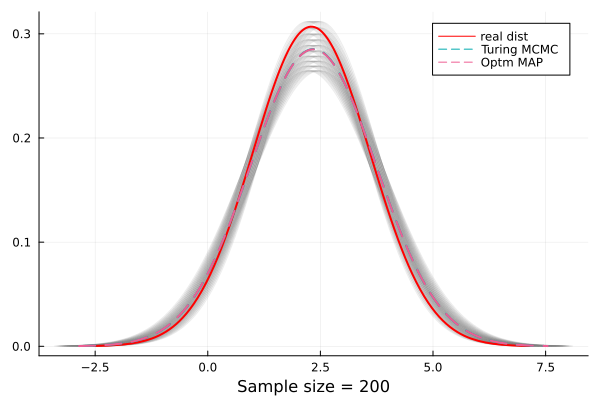
ズレは真の分布からサンプル生成によるばらつきによるもので、パラメータを正確に見つけていることがわかる。
##Gamma分布のパラメータ
Gamma分布でも可能か確かめる。前記同様データを用意する。
realgdist=Gamma(220,1.3)
ss = rand(realgdist,2000);
qs =[0.025,0.10,0.35,0.55,0.75,0.95]
xs = quantile(ss,qs)
NamedArray(xs',(["Quantile"],qs))
1×6 Named LinearAlgebra.Adjoint{Float64, Vector{Float64}}
A ╲ B │ 0.025 0.1 0.35 0.55 0.75 0.95
─────────┼─────────────────────────────────────────────────────
Quantile │ 247.178 261.086 278.057 288.445 299.087 318.269
モデルをGamma分布にあわせて修正する
@model function g_demo(xs,qs)
r ~ InverseGamma(2,3)
s ~ InverseGamma(2,3)
σ ~ Exponential(1) #収束しやすくなる
dist = Gamma(r,s)
xs .~ arraydist( [ Normal(quantile(dist,q),σ) for q in qs] )
end
Gamma分布はデフォルトのLBFGSでは収束失敗するのでNelderMeadを指定する。
Julia> optmle=optimize(model,MLE(),NelderMead())
ModeResult with maximized lp of -5.96
3-element Named Vector{Float64}
A │
───┼─────────
:r │ 207.94
:s │ 1.37399
:σ │ 0.653562
Turingでサンプリングをしようとすると問題発生する。Juliaのgammacdfが自動微分をサポートしないので、エラーになってしまう。むりやりForwardDiffを通るようにコードを書き足すことでサンプリングできるようになるけど、自己責任でお願いしたい。
using ForwardDiff,StatsFuns,SpecialFunctions
function SpecialFunctions.__gamma_inc_inv(a::ForwardDiff.Dual{T}, minpq::ForwardDiff.Dual{T}, pcase::Bool) where {T}
v_a= ForwardDiff.value(a)
v_pq = ForwardDiff.value(minpq)
SpecialFunctions.__gamma_inc_inv(v_a,v_pq,pcase)
end
c2=sample(model,NUTS(),3000,init_parameters=optmle.values.array)
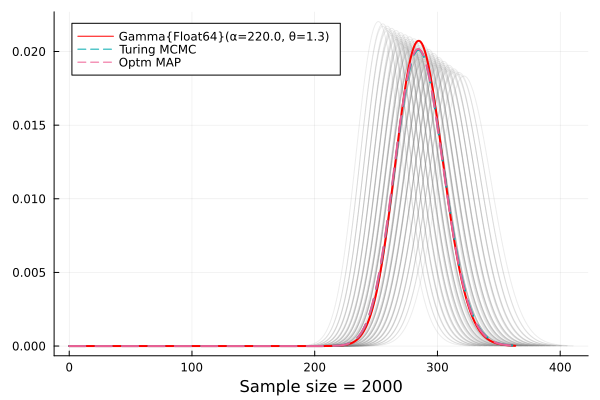
StasPlotsでビジュアル化する。
r = get(c2,[:r,:s])[:r] |> mean
s = get(c2,[:r,:s])[:s] |> mean
rs = quantile(c2)|> DataFrame |> t-> select(t,"2.5%","97.5%")
plot([Gamma(a,b) for a = range(rs[1,1:2]...,length=10) for b = range(rs[2,1:2]...,length=10)], color=:gray, alpha=0.15,label="")
plot!(realgdist,lw=2,label=realgdist,color=:red,xlabel="Sample size = $(length(ss))",legend=:topleft)
plot!(Gamma(r,s),lw=2,label="Turing MCMC",ls=:dash)
plot!(Gamma(optmle.values[:r],optmle.values[:s]),lw=2,ls=:dash,label="Optm MAP")
サンプルが2000もあればほぼ完ぺきに推定できる。まあ、現実のデータはこんなに当てはまりがよくないはずだし、逆にここまで当てはまったらデータを疑うべきだと思う。あとはよろしく!