はじめに
掲題の件、SDFやSMILESで与えた立体異性体をきちんと区別しているのか、ふと疑問に思ったため実験した。
環境
- Python 3.6
- RDKit 2018/9/2
- Jupyter Notebook
やったこと
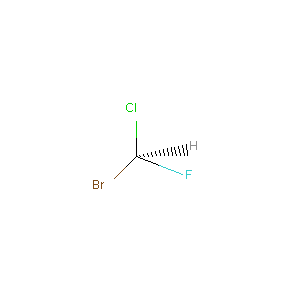
まず、あらかじめSDFで立体異性体の関係にある2つの化合物を作成した。
3次元座標ではなく、結合の種別として、手前側に伸びるのか、後ろ側に伸びるのかを指定し作成した。
1つ目
mol1 = Chem.SDMolSupplier("data/kiral.mol", removeHs=False)[0]
Draw.MolToImage(mol1)
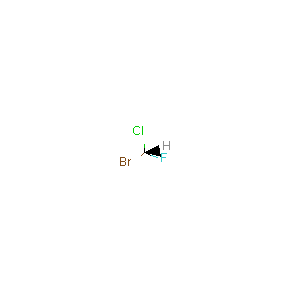
2つ目
mol2 = Chem.SDMolSupplier("data/kira2.mol", removeHs=False)[0]
Draw.MolToImage(mol2)
処理の都合上、画像の大きさが一致していないが、確かに2つをきちんと区別して描画している。
このMOLオブジェクトをSMILESに変換してみよう。
Chem.MolToSmiles(mol1, isomericSmiles=True)
'[H][C@@](F)(Cl)Br'
Chem.MolToSmiles(mol2, isomericSmiles=True)
'[H][C@](F)(Cl)Br'
SMILES上でもきちんと区別されている。
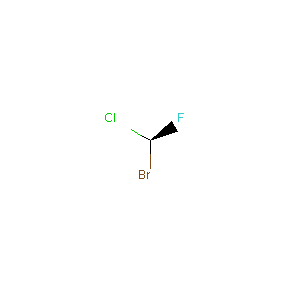
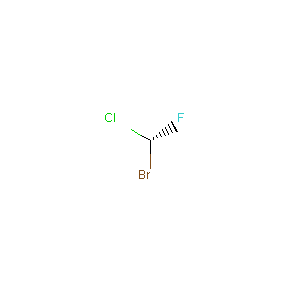
今度は逆に、上の2つのSMILESが区別されてMOLオブジェクトとして認識されるか確認しよう。
mol1FromSmiles = Chem.MolFromSmiles("[H][C@@](F)(Cl)Br")
mol2FromSmiles = Chem.MolFromSmiles("[H][C@](F)(Cl)Br")
上でSMILESから、MOLオブジェクトを生成し、以下の通り表示。
Draw.MolToImage(mol1FromSmiles)
Draw.MolToImage(mol2FromSmiles)
きちんと区別されて認識されているようだ。
ここには記載していないが、3次元化についても区別されることを確認。
当たり前の機能ではあるが、ちょっとスッキリした。