はじめに
化合物の物性・活性を予測するモデルを機械学習で作ったのはいいが、いざモデルの分析しようとしたときに、**説明変数に含まれるこの記述子って何だっけ?**となることがあると思う。記述子の説明や、論文を調べてもピンとこないことも多い。今回はそんな状況で記述子について手がかりを知る方法を共有したい。
方法
本記事で提唱する方法は次の2つである。
- 似たような計算結果となる記述子を調べてみる。似ているかどうかは、計算結果同士の相関をとることで判断する。
- 記述子の計算結果が大きい値をとる化学構造、小さい値をとる化学構造を調べてみる
今回の得体の知れない記述子
今回調べる記述子は独断と偏見で、mordred の GATS8are という記述子に決定~。理由としては、値が計算できないケースがあるので例として適切 および、説明を読んでも本当に何なのか全く想像がつかない という理由による。
mordredのホームページ 説明を読むと、geary coefficient of lag 8 weighted by allred-rocow EN という説明があり、全く想像がつかない。
環境
本手順を動かすには以下モジュールのインストールが必要になる。
- python
- jupyter lab
- rdkit
- mordred
早速やってみよう
まずモジュールをインポートしよう
import numpy as np
import pandas as pd
from rdkit import rdBase, Chem
from rdkit.Chem import AllChem, Descriptors
from rdkit.ML.Descriptors import MoleculeDescriptors
from mordred import Calculator, descriptors
ファイルを読み込もう。1列目に化合物名が、"smi"という列にSMILESが格納されている前提としている。
# ファイルの読み込み
import codecs
with codecs.open('sample.csv', 'r', 'utf-8', 'ignore') as f:
df = pd.read_csv(f, index_col = 0)
SMILESや名前を取り込むと同時に、SMILESからMOLオブジェクトを生成しておこう。
# SMILESのデータを取り出す
smiles_list = df["smi"].values
# 名前を取り出す
names_list = df.index
# SMILESのリストをMOLに変換
mols = []
for i, smiles in enumerate(smiles_list):
mol = Chem.MolFromSmiles(smiles)
if mol:
mols.append(mol)
else:
print("{0} th mol is not valid".format(i))
RDKitの記述子を計算しよう。
# RDKitの記述子計算
descriptor_names = [descriptor_name[0] for descriptor_name in Descriptors._descList]
descriptor_calculator = MoleculeDescriptors.MolecularDescriptorCalculator(descriptor_names)
rdkit_descriptors_results = [descriptor_calculator.CalcDescriptors(mol) for mol in mols]
df_rdkit = pd.DataFrame(rdkit_descriptors_results, columns = descriptor_names, index=names_list)
Mordredの記述子を計算しよう。
# mordredの記述子計算
mordred_calculator = Calculator(descriptors, ignore_3D=True)
df_mordred = mordred_calculator.pandas(pd.Series(mols, index=names_list))
# 数値型への変換
for column in df_mordred.columns:
if df_mordred[column].dtypes == object:
df_mordred[column] = df_mordred[column].values.astype(np.float32)
RDKitとMordredの計算結果をがっちゃんこしよう。列名が重複するので、適当なプレフィックスを事前につけておこう。
# 各記述子の名前にヘッダをつける
for df_tmp, prefix in zip([df_rdkit, df_mordred], ["RDKIT.", "MORDRED."]):
columns_new = [(prefix + column_tmp) for column_tmp in df_tmp.columns]
df_tmp.columns = columns_new
# 各データフレーム名を統合する
df_train_X = pd.concat([df_rdkit, df_mordred], axis=1)
いよいよ記述子の調査をはじめる。
対象記述子の計算結果にNULLが含まれる場合、他の記述子との相関を計算することができないため、行ごと(化合物ごと)削除しておく。
# 記述子を選定
descriptor = "MORDRED.GATS8are"
# 対象記述子がNULLのレコードを除去
df_train_X_mod = df_train_X[~df_train_X[descriptor].isnull()]
続いて、同じく相関が計算できない理由により、計算結果にNULLを含む列(記述子)を削除する。これによりNULLの値がデータフレーム上から一切なくなり、全ての記述子同士の相関を計算することができる。
# nullが含まれる列を削除
df_train_X_mod = df_train_X_mod[df_train_X_mod.columns[~df_train_X_mod.isnull().any()]]
df_train_X = df_train_X_mod
また、分散が0の列は、意味をなさないので削除しておこう。
# 分散が0のものを除去
from sklearn.feature_selection import VarianceThreshold
tmp_values = df_train_X.values
select = VarianceThreshold()
tmp_values = select.fit_transform(tmp_values)
df_train_X_vt = df_train_X[df_train_X.columns[select.get_support()]]
また、相関が1になる者記述子同士は、ほぼ同じものであるため、以前の記事を参考に片方を削除しておこう。ここでは、df_train_X_ctというデータフレームに削除後の結果が格納されたとして進める。
続いて、調査対象の記述子と他の全ての記述子の相関を計算し、絶対値の大きい順にならべてみよう
df_corr = df_train_X_ct.corr()
# 相関の絶対値で並べかえる
df_corr[descriptor].apply(lambda p: np.abs(p)).sort_values(ascending=False)
すると以下の結果とおり相関の高い順に記述子が表示される。一番上は自分自身であるため除外してよい。
MORDRED.GATS8are 1.000000
MORDRED.GATS8pe 0.979997
MORDRED.GATS8se 0.965887
MORDRED.GATS8dv 0.856247
MORDRED.MATS8are 0.821017
...
MORDRED.AATS4are 0.000613
MORDRED.AATSC5v 0.000479
MORDRED.AATS3se 0.000347
MORDRED.AATS4pe 0.000173
MORDRED.n6AHRing 0.000017
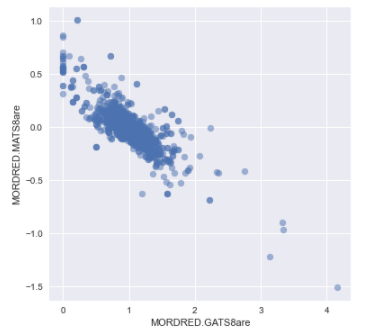
ここでは、試しに5番目の"MORDRED.MATS8are"という記述子との散布図をデータサイエンティストっぽくプロットなんかしてみる。
# プロットしてみる
get_ipython().run_line_magic('matplotlib', 'inline')
import matplotlib.pyplot as plt
plt.style.use("seaborn")
figure = plt.figure(figsize=(6, 6))
ax = figure.add_subplot(1,1,1)
ax.set_xlabel(descriptor)
ax.set_ylabel("MORDRED.MATS8are")
ax.scatter(df_train_X_ct[descriptor], df_train_X_ct["MORDRED.MATS8are"], alpha=0.5)
以上が1番目の方法として示した、似たような記述子を調べてみる、という方法である。
続いて2場面の方法である、記述子の値の大きいもの、小さいものを表示してみよう。
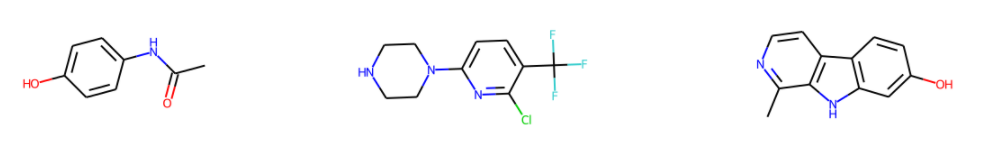
まず、大きい値をとる構造を出してみよう。
# 構造を生成するため、記述子計算結果とSMILESをがっちゃんこする。
df_merge = pd.merge(df_train_X_ct[descriptor], df["smi"], how="left", on="NO")
# 値の高いデータのMOLオブジェクトを生成
df_tmp = df_merge.sort_values(descriptor, ascending=False)
mols_lerge = []
for smiles in df_tmp["smi"]:
#print(smiles)
mol = Chem.MolFromSmiles(smiles)
mols_lerge.append(mol)
# MOLを表示
from rdkit.Chem import Draw
Draw.MolsToGridImage(mols_lerge[:10], molsPerRow=3, subImgSize=(400,400), )
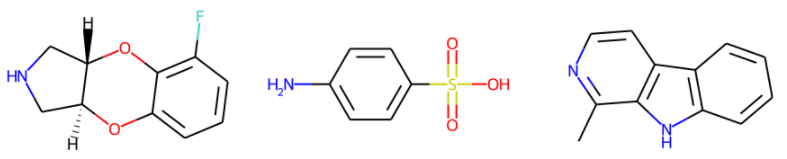
値の小さいものも出してみよう
# 値の小さいデータのMOLオブジェクトを生成
df_tmp = df_merge.sort_values(descriptor, ascending=True)
mols_small = []
for smiles in df_tmp["smi"]:
mol = Chem.MolFromSmiles(smiles)
mols_small.append(mol)
from rdkit.Chem import Draw
Draw.MolsToGridImage(mols_small[:10], molsPerRow=3, subImgSize=(400,400), )
おわりに
正直、今回は例にした記述子が難しすぎて、やってみたけど、ピンとこなかったというオチ。
ただ、うまくいくケースも稀にというか結構あるので、ぜひ使ってほしい**(汗)**