はじめに
Python の matplotlib を使って以下の様々な可視化手法を実行するコマンドを作成した。
- PCA
- tSNE
- MDS
- UMAP
コード
作成したプログラムのコードは以下の通り。
import numpy as np
import umap
import matplotlib.pyplot as plt
from sklearn.manifold import TSNE, MDS
from sklearn.decomposition import PCA
import pandas as pd
import argparse
def main():
parser = argparse.ArgumentParser()
parser.add_argument("-input", type=str, required=True, help="input csv file. first column is ID column")
parser.add_argument("-input2", type=str, help="input other csv file. must be same format '-input'")
parser.add_argument("-target", action='store_true', help="set second column as target column")
parser.add_argument("-target2", action='store_true', help="set second column as target column")
parser.add_argument("-silent", action='store_true', help="not show graph")
parser.add_argument("-save", type=str, help="graph saving folder")
parser.add_argument("-method", type=str, required=True, action="append", choices=["PCA", "tSNE", "MDS", "UMAP"])
args = parser.parse_args()
# データの読み込み
df = pd.read_csv(args.input, index_col=0)
# 説明変数の取得
if args.target:
df_target = df.iloc[:, 0]
df_data = df.iloc[:, 1:]
else:
df_data = df
# input2が指定されている場合
if args.input2:
df2 = pd.read_csv(args.input2, index_col=0)
if args.target2:
df_target2 = df2.iloc[:, 0]
df_data2 = df2.iloc[:, 1:]
else:
df_data2 = df2
list_class = [0] * df_data.shape[0]
list_class2 = [1] * df_data2.shape[0]
df_last = pd.concat([df_data, df_data2])
#df_target_last = pd.concat([df_target, df_target2])
list_class_last = list_class + list_class2
print(df_data.shape)
print(df_data2.shape)
df_data = df_last
df_target = list_class_last
if "PCA" in args.method:
pca = PCA(n_components=2)
pca_result = pca.fit_transform(df_data)
plt.title("PCA")
if args.target:
plt.scatter(pca_result[:, 0], pca_result[:, 1], c=df_target)
else:
plt.scatter(pca_result[:, 0], pca_result[:, 1])
if not args.silent:
plt.show()
if args.save:
plt.savefig(args.save + "/PCA.png")
if "MDS" in args.method:
mds = MDS(n_jobs=4)
mds_result = mds.fit_transform(df_data)
plt.title("MDS")
if args.target:
plt.scatter(mds_result[:, 0], mds_result[:, 1], c=df_target)
else:
plt.scatter(mds_result[:, 0], mds_result[:, 1])
if not args.silent:
plt.show()
if args.save:
plt.savefig(args.save + "/MDS.png")
if "tSNE" in args.method:
tsne_model = TSNE(n_components=2)
tsne = tsne_model.fit_transform(df_data)
plt.title("tSNE")
if args.target:
plt.scatter(tsne[:, 0], tsne[:, 1], c=df_target)
else:
plt.scatter(tsne[:, 0], tsne[:, 1])
if not args.silent:
plt.show()
if args.save:
plt.savefig(args.save + "/tSNE.png")
if "UMAP" in args.method:
embedding = umap.UMAP().fit_transform(df_data)
plt.title("UMAP")
if args.target:
plt.scatter(embedding[:, 0], embedding[:, 1], c=df_target)
else:
plt.scatter(embedding[:, 0], embedding[:, 1])
if not args.silent:
plt.show()
if args.save:
plt.savefig(args.save + "/UMAP.png")
if __name__ == "__main__":
main()
使い方
コマンドラインの説明を以下に示す。argparseの出力をそのままはりつけ~。argparseは本当に便利だ。
usage: ViewMat.py [-h] -input INPUT [-input2 INPUT2] [-target] [-target2]
[-silent] [-save SAVE] -method {PCA,tSNE,MDS,UMAP}
オプションの説明
- input 入力ファイルを指定。csv形式を前提。1列目はIDと仮定し、解析には含まない。
- target 指定した場合、2列目を目的変数とみなす。この場合、この列は解析に含まず色表示に利用。
- silent 画面にグラフを表示しない。長時間かかる処理でかつ保存だけしたい場合に便利。
- save 指定したフォルダに画像を保存する
- method 処理したい手法を指定。複数指定したい場合は、-target PCA -target tSNE等と繰り返し指定する。
- input2 入力ファイルその2を指定(オプション)。これを指定すると、inputで入力したファイルのデータと色を分けて表示する。当然inputとinput2の列は同じものでなければならない。
- target2 input2を指定した場合に、目的変数列(2列目)を解析に含まない場合は指定する必要がある。
入力ファイルの説明
- csv形式
- 1行目はヘッダ
- 2行名以降はデータ(当然数値です)
- 1列目はID。(今回は処理に全く絡まない)
- targetオプションを指定した場合, 2列目は目的変数。3列目以降が可視化手法に与えられるデータ。
- targetオプションを指定しない場合、2列目以降が可視化手法に与えられるデータ。
実行例
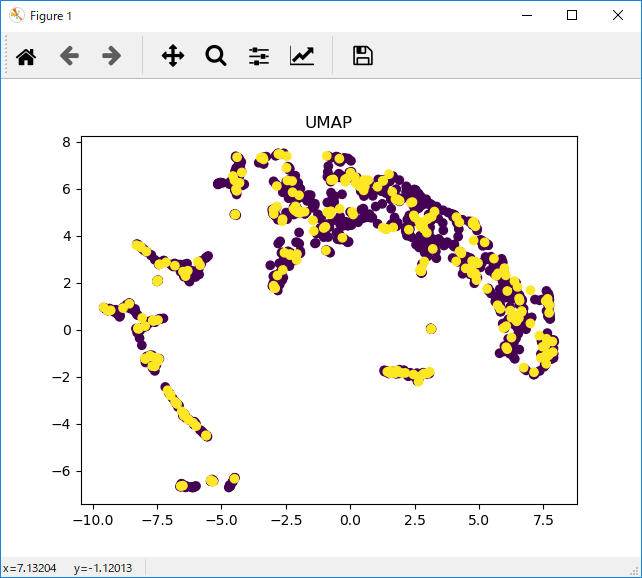
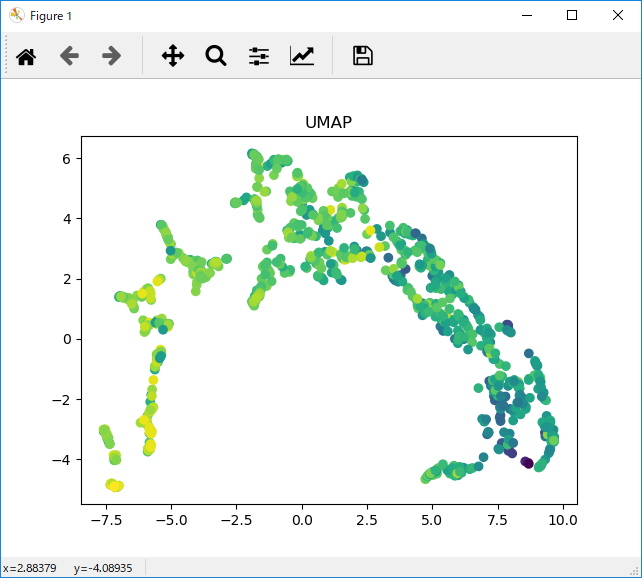
inputとして92列をもつ学習データ数1025件でtargetを指定した場合、こんな感じ。学習データとtarget値にかなり相関がある様子が伝わってくる。
さらに同じ列数をもつ257件のテストデータをinput2を指定した場合、こんな感じ。多分黄色がinput2の方。凡例だせよって感じ。
学習データとテストデータのばらつきがそんなに変わらない様子が伝わってくる。
所感
- Jupyter Notebook だとその場限りのコードになってしまいがちなので、コマンドラインで使えるようにしたが、特につまるところはなかった。
- 10000件以上のデータでUMAPを試してみたが、数分で終了して噂通り、速いなぁーと実感。
- 次はbokehをつかってグラフの点にフォーカスしたらIDを表示したり、色表示するtargetを動的に変更したりできるインタラクティブなグラフ作成に取り組みたい。