はじめに
最近、Google Colaboratoryを使い始めた。ローカルにJupyter環境を設定し起動するのが面倒なので、ちょっとした確認などは、Google Colaboratoryを使うようにしている。
基本的なライブラリは、Google Colaboratoryに最初からインストールされているのだが、ケモインフォマティクス用のライブラリ(RDKitやmordredなど)はインストールされていない。しかもこれらは、基本condaによるインストールとなっており、Google Colaboratoryではcondaがデフォルトでは使えない。
そこで今回、Google Colaboratoryでcondaを使えるようにし、RDkitやmordredをインストールして動かしてみるところまでやったのでメモっておく。
condaのインストールは、Conda + Google Colab を参考にした。
また、本記事と同じような趣旨の記事が既に「化学の新しいカタチ」さんの
Google ColabでRDKit:ケモインフォマティクス用のpython環境を手軽に構築に記載されている。
Condaのインストール
Pythonパスの確認
!which python
するとPythonのパスが返ってくる。ここでは/usr/local/bin/pythonが返ってくる
Pythonバージョンの確認
!python --version
するとPythonのバージョンが返ってくる。ここでは、Python 3.6.9が返ってくる
PYTHONPATH環境変数の確認と解除
!echo $PYTHONPATH
すると PYTHONPATH環境変数が返ってくる。ここでは、/env/pythonが返ってくる。
minicondaをインストールする場合、PYTHONPATHに含まれるディレクトリを介してインストールおよびアクセスできるパッケージがある場合に、問題が発生する可能性があるらしいので、以下コマンドによりPYTHONPATH変数の設定を解除する。
%env PYTHONPATH=
minicondのインストール
%%bash
MINICONDA_INSTALLER_SCRIPT=Miniconda3-4.5.4-Linux-x86_64.sh
MINICONDA_PREFIX=/usr/local
wget https://repo.continuum.io/miniconda/$MINICONDA_INSTALLER_SCRIPT
chmod +x $MINICONDA_INSTALLER_SCRIPT
./$MINICONDA_INSTALLER_SCRIPT -b -f -p $MINICONDA_PREFIX
インストール後の確認
!which conda
うまくいっていれば、/usr/local/bin/condaと返ってくるはずだ。
Pythonのバージョン確認
!python --version
するとpython 3.6.5 :: Anaconda, Inc.のように、minicondaによって異なるバージョンのpythonがインストールされていることが分かる。
Pythonのアップグレード
以下のコマンドでpythonやライブラリをアップグレードしよう
%%bash
conda install --channel defaults conda python=3.6 --yes
conda update --channel defaults --all --yes
Pythonのバージョン確認
!python --version
するとPython 3.6.12 :: Anaconda, Inc.のようにpythonがアップグレードされたことが分かる。
sys.pathへのパスの追加
Minicondaをインストールしたので、インポートするモジュールを探すときにPythonが検索するディレクトリのリストに、condaのパッケージパスを追加する必要がある。以下の通り実行する。
import sys
_ = (sys.path
.append("/usr/local/lib/python3.6/site-packages"))
以上でcondaのインストールは完了である。
RDkitのインストール
condaをインストールすることができたので、以下のcondaコマンドでRDKitをインストールしよう。
!conda install -c rdkit rdkit --yes
うまくインストールされたかバージョンを確認する。
import rdkit
print(rdkit.__version__)
すると、2020.09.1のようにRDKitのバージョンが表示されるはずだ。
SMILESからmolオブジェクトを作ってMDL形式で表示してみよう。
from rdkit import Chem
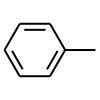
mol = Chem.MolFromSmiles('Cc1ccccc1')
Chem.MolToMolBlock(mol)
続いてmolオブジェクトを画面上に表示してみよう。
from rdkit.Chem import Draw
Draw.MolToImage(mol, size=(100,100))
mordredのインストール
続いて記述子計算ソフトウェアであるmordredをインストールしてみよう。
!conda install -c rdkit -c mordred-descriptor mordred --yes
そして、早速記述子計算をしてみよう。
from mordred import Calculator, descriptors
import pandas as pd
calc = Calculator(descriptors, ignore_3D=True)
df_descriptors_mordred = calc.pandas([mol])
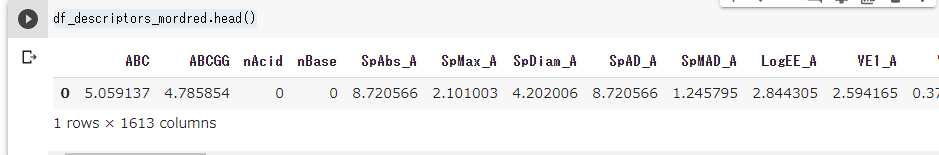
計算がおわったらpandasのデータフレームに結果が得られるので表示してみよう。
df_descriptors_mordred.head()
するとこんな感じで計算結果が得られるはずだ。
さて、ここまでできればあとは通常の機械学習と同じなので、matplotlibや、scikit-learn、PyTorch等を使って色々な解析ができるはずだ。
おわりに
condaが使えるようになったことで、他にも色々なライブラリが利用でき、実質ローカルで環境構築するのとかわらない環境を手に入れたといえるだろう。勿論Google Colaboratoryは一定時間立つとリセットされるようなので毎回この手順を踏まなければならないが、保存したNotebookに従って実行するだけなのでそれほど手間ではないだろう。