はじめに
前回記事 AWSのライフサイエンス分野向けグラフ深層学習ライブラリ DGL-LifeSci を触ってみたで作成した予測モデルを用いて、KNIME で化合物の物性を予測できるようにしてみたメモ
前提
本記事では、KNIMEについては説明を省略する。
環境
前回と同様以下の環境を用意する。ただしKNIMEは別途インストールする。
- Windows 10 (RAM16G、GPU:GetForce GTX 1060 6GB)
- Miniconda 3
- python 3.7
- pytorch 1.10.2
- dgl dgl-cuda11.0
- RDKit 2018.09.3
- KNIME 4.5.2
やり方
以下の方法で予測モデルを実行する。
- 予測したい化合物のファイルはSDFに格納されている前提とする。
- SDFファイルの読み込みに、KNIME の SDF Reader ノードを利用する。
- 予測モデルを実行するために、KNIME のExternal Tool(Labls) という外部コマンドを実行するためのノードを利用する。
- DGL-LifeSciの予測コマンドは、
C:\dgl-lifesci\examples\property_prediction\csv_data_configurationにあり、予測モデルはその下のregression_result\1にあるとする - External Tool(Labls)ノードと予測モデルの実行をつなぐプログラム(バッチファイル、pythonスクリプト)は、
C:\dgl-lifesci\examples\property_prediction\csv_data_configuration\my_programフォルダに作成する。
早速やっていこう
External Tool(Labls)ノードが呼び出す外部ツールとしてのバッチファイルの作成
まず、KNIMEのExternal Tool(Labls)ノードから呼び出されるバッチファイルを作成しよう。
call conda activate dgllife
cd C:\dgl-lifesci\examples\property_prediction\csv_data_configuration
python regression_inference.py -f %1 -sc SMILES -t lipo -tp .\regression_results\1 -ip my_program
python .\my_program\merge.py -i %1 -o %2
ざっくりいうと、仮想環境のアクティベート、ディレクトリの移動、DGL-LifeSciの予測コマンドの実行、次で説明するマージ用Pythonスクリプトの実行を行っている。%1, %2は、External Tool(Labls)ノードから呼び出される際に指定される引数(%1が第一引数、%2が第二引数)であり、それぞれExternal Tool(Labls)ノードが外部ツールに与える入力ファイル、External Tool(Labls)ノードが外部ツールから結果を受け取る出力ファイルを意味している。
マージ用Pythonスクリプトの作成
次に、External Tool(Labls)ノードから受け取った入力ファイルと、DGL-Life/Sciの予測結果をマージし、External Tool(Labl)ノードが結果を受け取るための出力ファイルを生成するスクリプトを作成する。
import pandas as pd
from argparse import ArgumentParser
from rdkit.Chem import AllChem, Descriptors, MolFromSmiles, SDMolSupplier, MolToSmiles
def main():
parser = ArgumentParser()
parser.add_argument('-i', type=str, required=True)
parser.add_argument('-o', type=str, required=True)
args = parser.parse_args()
# KNIMEの「ExternalTool(Labs)」の出力ファイルを読み込み
df_input = pd.read_csv(args.i, index_col=0)
canonical_smiles = []
# KNIMEのSMIELS列は"SMILES"と仮定。DGL-LifeSciの予測結果とマージするためcanonical smilesに変換
for smiles in df_input["SMILES"]:
mol = MolFromSmiles(smiles)
if mol is not None:
canonical_smiles.append(MolToSmiles(mol))
else:
canonical_smiles.append(None)
df_input['canonical_smiles'] = canonical_smiles
# DGL-LifeSciの予測結果を読み込み
df_output = pd.read_csv("./my_program/prediction.csv")
# KNIMEの結果とDGL-LifeSciの予測結果をcanonical smilesでマージ
df_marge = pd.merge(df_input, df_output, how="left", on="canonical_smiles")
# KNIMEの「ExternalTool(Labs)」が入力と出力をマージできるよう列名を"Row1,Row2,..."に振りなおす
index = ["Row{0}".format(i) for i in range(len(df_input))]
df_marge.index = index
df_marge.index.name ="Row ID"
# 予測結果列のみに絞り込み
df_marge = df_marge[["lipo"]]
# 結果を出力
df_marge.to_csv(args.o)
if __name__ == "__main__":
main()
ざっくりいうと、(バッチファイルの実行フォルダから相対的にみて) ./my_program/prediction.csv にDGL-LifeSciの予測結果が出力されるため、それを読みだしてExternal Tool(Labls)ノードが結果を受け取れる形式に変換している。具体的には External Tool(Labls)ノードの、外部ツールに与える入力ファイルの行ID(この場合、SDF Readerの出力であるRow 1, Row2, ...) と、行IDが一致している行を出力ファイルから読み込み出力する という仕様に沿う形でファイルを出力している。DGL-LifeSciの予測結果に出力されるSMILESが、入力ファイルのSMILESではなくcanonical smilesであるということもあいまって、かなり面倒な処理をしているがあとはソースを見てほしい。
KNIMEを起動
さて準備がととなったので、KNIMEを起動し、ワークフローを作成しよう。
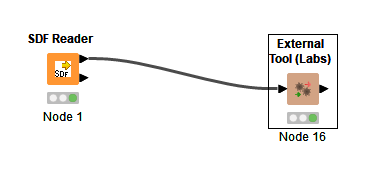
ノードを配置
続いてSDF ReaderノードとExternal Tool(Labs)ノードを以下のように配置しよう。どのノードか分からない場合は、Node Repositoryから検索すればよいだろう。
SDF Reader ノードの設定
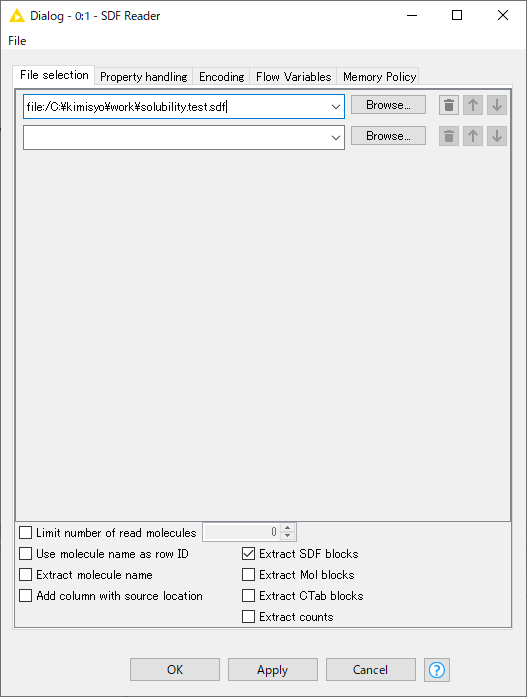
SDF Readerノードを選択し、右クリック→「Configure」をクリックし、予測したいSDFファイルを選択しよう。
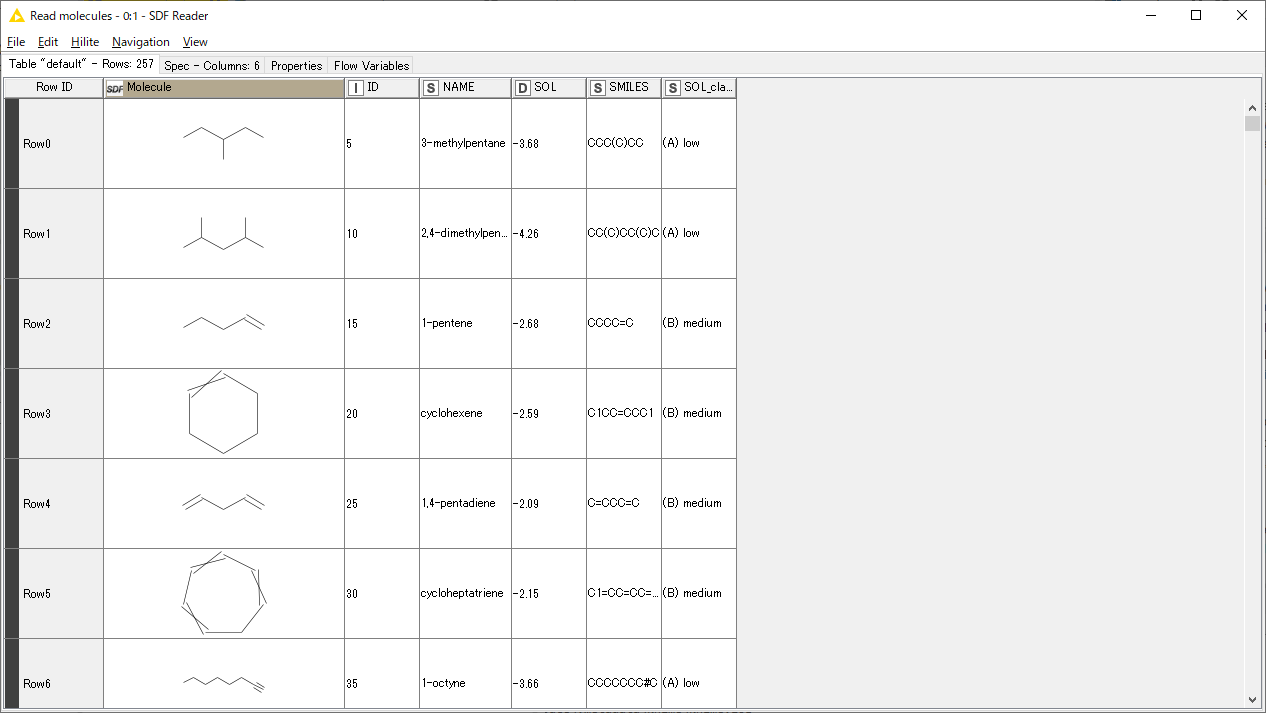
ちなみに右クリック→「Read Moecule」をクリックすると、こんな形で次のノードに引き渡されることが分かる。
External Tool(Labls)ノードの設定
次にExternal Tool(Labls)ノードを選択し、右クリック→「Configure」をクリックしよう。
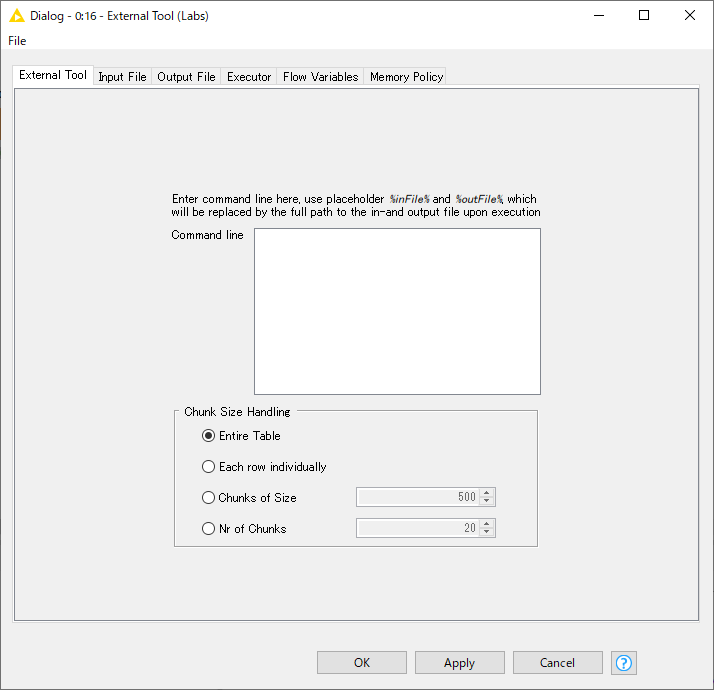
External Toolタブの設定
External Toolタブを開き、Command Line に先ほど作ったバッチファイルのパス、引数を以下の通り設定しよう。%inFile%, %outFile%はこのあと、input Fileタブ, Output Fileタブで設定する入力ファイル、出力ファイルを指定するための変数である。
C:\dgl-lifesci\examples\property_prediction\csv_data_configuration\my_program\Predict.bat %inFile% %outFile%
なお、パスのデリミタとして '\' が認知されない場合は、"\\" とエスケープする必要がある
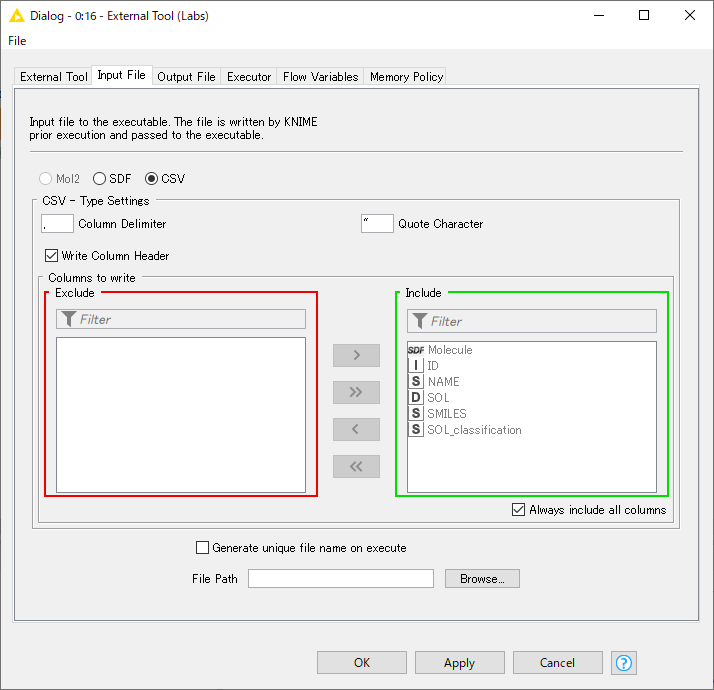
Input Fileタブの設定
Input Fileタブを開き、File Path に以下の通り入力ファイルパスを指定しよう。このファイルはKNIMEが自動で作成し、バッチファイルの第一引数に与える。
C:\dgl-lifesci\examples\property_prediction\csv_data_configuration\my_program\input.csv
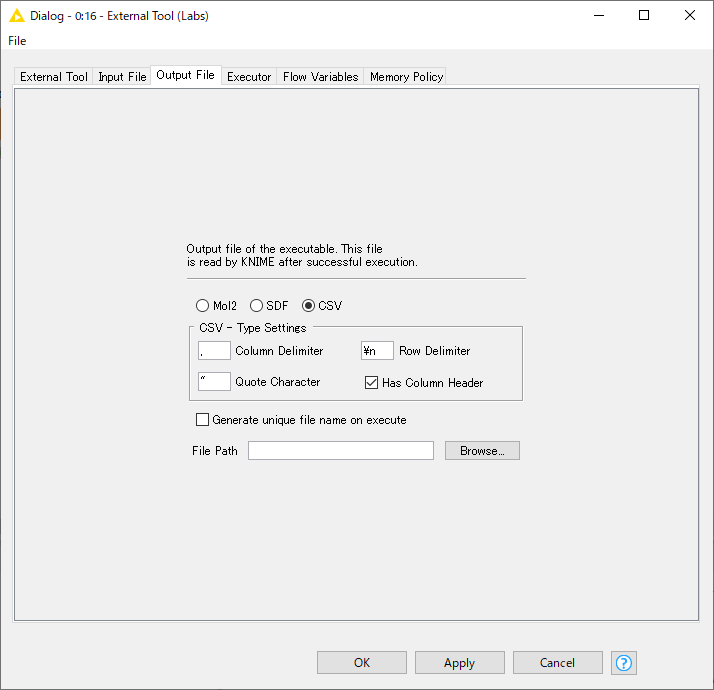
Output Fileタブの設定
Output Fileタブを開き、File Path に以下の通り出力ファイルパスを指定しよう。このパスはバッチファイルの第二引数に与えられ、バッチファイル(の中のmerge.py)が最終的に出力するファイルとなる。
C:\dgl-lifesci\examples\property_prediction\csv_data_configuration\my_program\output.csv
予測実行
さて準備は整った。External Tool(Labls)ノードを選択し、右クリック→「Execute」をクリックしよう。
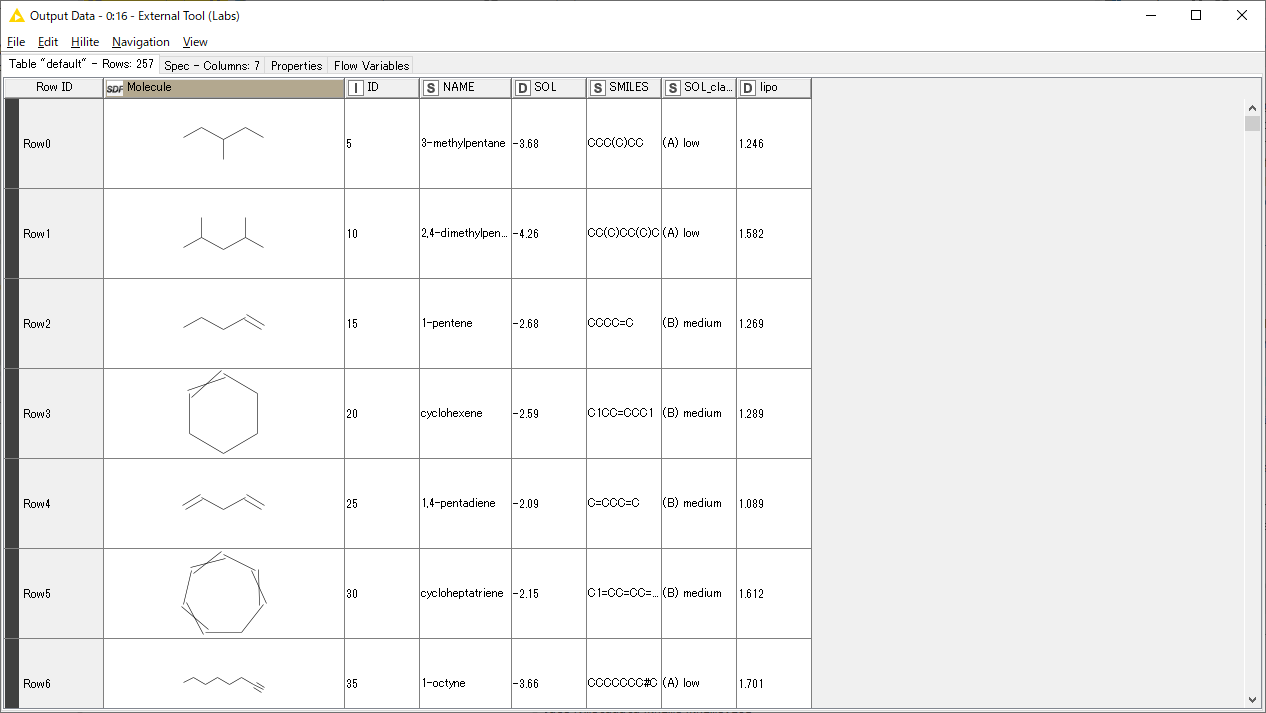
実行が終わったらExternal Tool(Labls)ノードを右クリックし、「Output Data」をクリックしよう。
うまくいっていれば、一番右に列が追加され、そこに予測結果が出力されているはずだ。
パスのミスなどで中々うまくいかなかったため、動いた時は思わずガッツポーズが出た。
おわりに
今回は DGL-LifeSciで作成した予測モデルを例に紹介したが、scikit-learnで作成した予測モデルなど、コマンドラインで実行できる予測モデルであれば、今回の merge.py のように小細工をすれば、全て同じように予測できるはずだ。快適な環境になるよう色々と試してほしい。