はじめに
マイクロバイオーム解析によく使われる QIIME2 は素晴らしいプラットフォームであるが、標準装備以外の解析や可視化を行いたい場合、簡単ではない。
本記事では、今回 Taxonomiy analysis における Taxonomic Bar Plots の表示に関して、Jupyter で再現し見やすく表示してみたので共有したい。
前提環境
- Windows10
- WSL2 (Ubuntu 20.04.5 LTS)
- miniconda 3
困ったこと
Taxonomic Bar Plotsとは
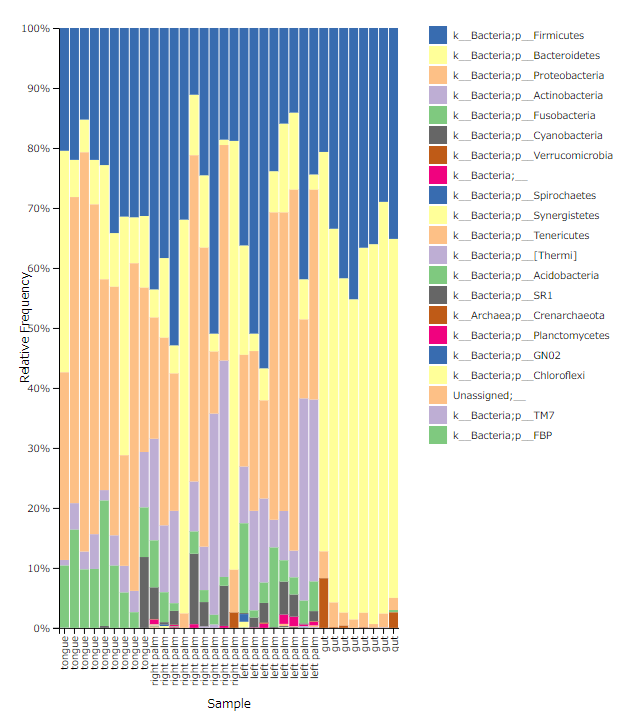
Taxonomy analysis における Taxonomic Bar Plots とはこのリンクにあるように、各サンプルの細菌叢における菌種の割合をグラフィカルに表示してくれるものである。
困ったこと
上のサンプルのリンクで、例えば Taxonomy Level に「Level2」, Sort Samples By に「k__Bacteria;p__Firmicutes」, 「Descending」を指定すると、以下のように「k__Bacteria;p__Firmicutes」という細菌種別の割合の多い順にサンプル順を並べ替えることができる。
素晴らしい。続いて、メタデータにより細菌叢の違いがあるかどうか知りたいため、Sort Samples Byに例えばsample-metadataの「body-site」を指定してみる。
すると、確かにX軸のSampleは「tongu」, 「right palm」, 「left palm」, 「gut」のカテゴリ別にソートされてはいるものの、カテゴリ内の順番がばらばらになっておりカテゴリ毎の菌種の分布が良く分からない。これは 指定できるソートが1つのみ のためだろう。さらにいうと、サンプルIDが表示されないため、各バーがどのサンプルか分からない。右上のRelabel X?からチェックを外すと、サンプルIDは表示されるが、今度はカテゴリが表示されない。
本記事では、これらを解消すべく、Jupyter Lab を使って QIIME2 の Taxonomic Bar Plots を 完全に再現 し、なおかつサンプルIDを カテゴリ順、菌種順に並べ替えて表示させる手順 について説明する。
事前準備
前提環境に記載してる環境は用意されており、conda のPython仮想環境は作成済の前提で記載する。
Pythonモジュールのインストール
必要モジュールで必要なものをインストールしよう
$conda activate <仮想環境名>
$conda install -c conda-forge jupyterlab
$conda install pandas -c conda-forge
$conda install matplotlib -c conda-forge
Jupyter Labを起動
Jupyter Lab を起動しよう
$jupyter lab --ip 0.0.0.0
うまくいけば以下のようなメッセージが出るため、URLをクリックするとJupytor Labが表示される(下図)。
Jupyter Server 1.23.3 is running at:
http://MyComputer:8888/lab?token=607d65583fb4cb4f5a4bb17c71e816605820f41c9d0ace04
or http://127.0.0.1:8888/lab?token=607d65583fb4cb4f5a4bb17c71e816605820f41c9d0ace04
Jupyterlab上でNoteBookを作成
続いてメニューの「File」⇒「New」⇒「NoteBook」を選択すると以下のダイアログが表示されるため「Select」をクリックする。

すると以下のように「Untitled.ipynb」という名前のNoteBookが生成される。
名前などは変更できるため、やり方をググって修正してみよう。
データの準備とアップロード
QIIME のメタデータファイルを JupyorLab にアップロードしておく。
また、再現したいデータを Qiime2 View に表示し、Download の下のCSVボタン(下図参照)よりcsvファイルをダウンロードし、これも Jupyter Lab にアップロードしておく。

以下それぞれ、sample-metadata.tsv、level-2.csvの名前でアップロードしたとして説明する。
アップロードは、下の赤枠のところにファイルをドラッグ&ドロップすることで簡単に行うことできる。

Jupyter Lab で Taxonomic Bar Plots を再現する手順
Jupyter Lab ではセルにプログラムを入力・実行することで、実行結果を逐次確認しながら対話的に分析を進めることができる。詳しい使い方は、ググるとここのようなページが見つかるので参考にしてみよう。
以下、各プログラム片を順にセルに入力・実行することを繰り返していくことで最後に Taxonomic Bar Plots が表示される。
モジュールの読み込み
import pandas as pd
import matplotlib.pyplot as plt
import matplotlib.cm as cm
import numpy as np
from functools import cmp_to_key
カテゴリの設定
サンプルIDのソートに使うカテゴリIDを指定する。
メタデータファイルに定義されているものを指定する。
# カテゴリの設定
category = "body-site"
メタデータの読み込み
メタデータファイル名が異なる場合は修正する。
# メタデータの読み込み
df_metadata = pd.read_table("./sample-metadata.tsv", index_col=0, skiprows=[1])
# 大元のサンプルID一覧を保持
sample_ids = df_metadata.index.values
# サンプルIDを、サンプルID-カテゴリに変換
sample_ids2 = [label + "-" + er for (label, er) in zip(df_metadata.index, df_metadata[category].values)]
QIIME2 view からダウンロードしたCSVファイルの読み込み
CSVのファイル名が異なる場合は修正する。
# サンプルID-Levelのファイルの読み込み
df_bar_org = pd.read_csv("./level-2.csv", index_col = 0)
# サンプルIDの並びがメタデータファイルの順番と一致していない場合があるためそろえる
df_bar_org = df_bar_org.reindex(index=sample_ids)
以下、-8 の "8" はメタデータファイルにおけるメタデータの数を示している。数が異なる場合は調整する。
#不要なカラムを削除
df_bar_tmp= df_bar_org.iloc[:, :-8]
ソート
以下、存在量の最も大きな菌種を選定し、行と列を並べ替える
#存在量の大きな菌種レベル順に並べ替え
df_sum_by_class = df_bar_tmp.sum(axis=0)
df_sum_by_class = df_sum_by_class.sort_values(ascending=False)
sum_by_class = df_sum_by_class.index.values
sort_class = sum_by_class[0]
# サンプルID毎のLevelの存在量を、サンプル内の割合(%)に変換
df_sum_by_sample = df_bar_tmp.sum(axis=1)
for column in df_bar_tmp.columns:
df_bar_tmp[column] = (df_bar_tmp[column]/df_sum_by_sample)*100
# サンプルID-Levelテーブルの列を指定の菌種順に並び替え
df_bar = df_bar_tmp.copy()
df_bar = df_bar.reindex(columns=df_sum_by_class.index)
# サンプルIDをカテゴリ、菌種(sort_class)の存在量、サンプル順にソートする関数
def index_compare2(data1, data2):
data1s = data1.split("-");
data2s = data2.split("-");
value1 = df_bar_tmp.loc[data1s[0], sort_class]
value2 = df_bar_tmp.loc[data2s[0], sort_class]
# カテゴリ
if data1s[1] < data2s[1]:
return 1
elif data1s[1] > data2s[1]:
return -1
else:
# 菌種(sort_class)の存在量
if value1 < value2:
return 1
elif value1 > value2:
return -1
else:
# Sample ID
if data1s[0] < data2s[0]:
return -1
elif data1s[0] > data2s[0]:
return 1
else:
return 0
df_bar.index = sample_ids2
sample_ids_sort2 = sample_ids2.copy()
sample_ids_sort2.sort(key=cmp_to_key(index_compare2))
df_bar = df_bar.reindex(index=sample_ids_sort2)
いよいよmatplotlibで描画
グラフの色をQIIMEのデフォルト色に設定する。
# 色の設定
colors = [(56, 108, 176), (255, 255, 153), (253, 192, 134), (190, 174, 212), (127, 201, 127), (102, 102, 102), (191, 91, 23), (240, 2, 127) ]
colors_new = []
for color in colors:
colors_new.append(tuple(np.array(color)/255))
いよいよ最後の積み上げグラフの描画になる。(積み上げグラフはこちらを参考にしています)
df_bottom = pd.DataFrame(index=df_bar.index);
for i, column in enumerate(df_bar.columns):
df_bottom[column] = np.ones(len(df_bar)) * 100
for j, column_j in enumerate(df_bar.columns):
if j > i:
break
df_bottom[column] -= df_bar.iloc[:, j]
plt.figure(figsize=(7, 5))
barWidth = 0.99
for i, column in enumerate(df_bar.columns):
print(column)
plt.bar(df_bar.index, df_bar[column], bottom=df_bottom[column], color=colors_new[i % len(colors)], edgecolor='white', width=barWidth, label=column)
# Custom x axis
plt.ylabel("Relative Frequency (%)")
plt.xlabel("Sample")
plt.xticks(rotation=90)
plt.ylim(0,100)
import matplotlib as mpl
mpl.rcParams['axes.xmargin'] = 0
mpl.rcParams['axes.ymargin'] = 0
# Show graphic
plt.legend(loc='upper left', bbox_to_anchor=(1,1), ncol=1)
plt.show()
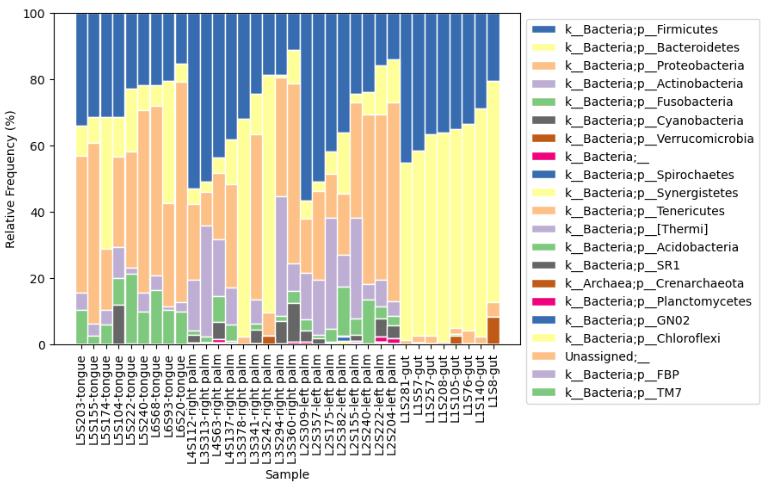
実行結果
実行結果は以下のようになる。「site-body」別に、「k__Bacteria;p__Firmicutes」の存在割合の大分な順に表示されている。また、X軸のラベルがサンプルID-カテゴリの形式になっている。QIIME2 viewに比べると大分見やすくなったのではないだろうか。
終わりに
QIIME2は素晴らしいツールであるが万能ではない。本記事ではQIIME2で出力したデータを元に自前で可視化する例を紹介したので、より深い解析をしたい方の参考になれば幸いである。