Cytoscapeとは?
Cytoscapeとは、欧米の研究機関によって開発されているオープンソースのネットワーク可視化ソフトウェアプラットフォームです。ネットワーク(数学の用語ではグラフ)可視化ソフトウェアとは、要するにデータを分析して、最終的には以下の様な描画を行うものです。
元となる情報は基本的に何でも良いので、以下の様な分野で利用されています:
- 遺伝子ネットワーク分析(主な開発目的です)
- ソーシャルネットワーク分析(日本語でも幾つか著作のあるJames Fowler教授にも協力していただいています)
- コンピュータ・ネットワークの可視化とセキュリティ分析
- オントロジー等、グラフ構造を持つデータの可視化
このソフトウェアはもう10年以上も開発が続いています。私もカリフォルニア大学のチームの一員として、かなり初期から開発に加わっておりますが、私の努力不足もあり、日本語のドキュメントはとても少ないのが現状です。かつて私が書いたいくつかのドキュメントやスライドは既に情報が古くなってしまったものが多いので、この辺りで一度、日本の皆様にもっとお使いいただけるように、情報の探し方だけでもお伝えできればと思いこの記事を書いてみました。
(因みにリンク先の画像は、全て私がCytoscapeのみで作成したものですから、CC BYでご自由に再利用して頂いて構いません。)
何が出来るのか?
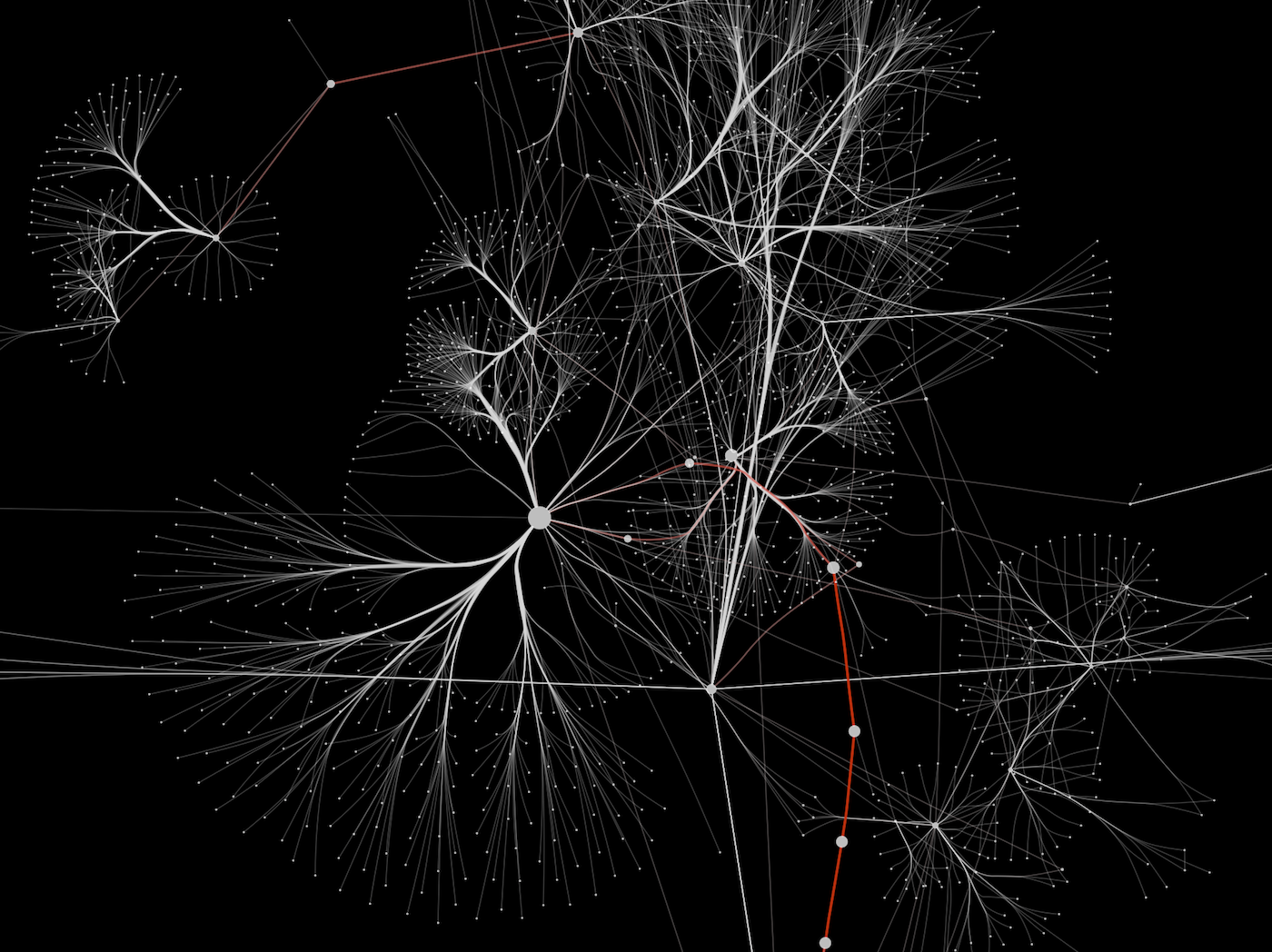
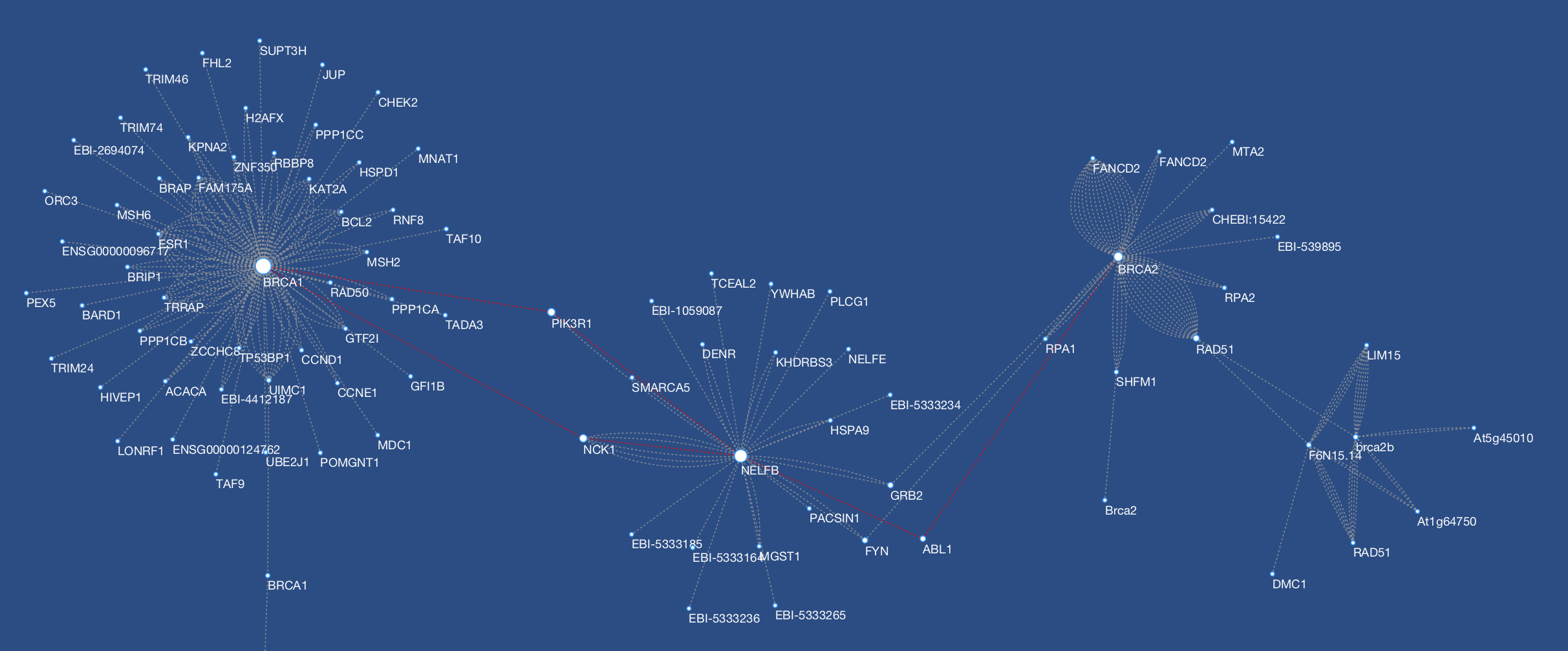
基本的な考え方はとてもシンプルです。それは、__グラフ構造(ネットワーク)とそれに付随する情報(アトリビュート)を読み込み、それを組み合わせて分析、可視化を行う__というものです。簡単な具体例をあげてみます。下の画像があるソーシャルネットワークの一部だとしましょう(実際は生体内のタンパク質ネットワークの一部ですが):
この図では、相対的にネットワークの中で重要な位置を占める人が大きく、ネットワークの中で情報(口コミ)が伝播する時に重要な経路が濃い赤色で表示されています。なぜそれらが重要なのかというのは、様々な決め方がありますが、最も単純な方法としては、ノードやエッジの特徴を表す統計値(degree/centrality/betweenness等といった値)をグラフ構造そのものから計算する方法です。こういった値と視覚的要素をマッピングし、人間にとってExcelの表などよりも理解しやすく可視化するのがCytoscapeの目的です。
最も基本的なワークフローは以下の様なものです:
- ネットワークデータを読み込む
- アトリビュート(データテーブル)を読み込む
- ソーシャルネットワークの例で言えば
- 人の名前、年齢、職業、性別などがノードアトリビュート
- 婚姻関係、友人関係、メールの交換された数などといった関係性に関する情報がエッジアトリビュート
- ソーシャルネットワークの例で言えば
- 統合されたデータを使って、統計的な解析やフィルタリングなどを行い分析する
- 自動レイアウト機能を使って、ノードを見やすく配置する
- ノード、エッジに付随する各種データに基づきマッピングを設定し、描画を行う
- PDFやJavaScriptウィジェットに出力する
つまり、人や物の繋がり、概念の関係性など、実際には目に見えない物をデータ化し、それを目に見える形に落としこむためのソフトウェアと考えていただければ良いと思います。
プロジェクトの現状
Cytoscapeは、私のようなフルタイム開発者も含めた国際的なチームが、主に米国、カナダ、フランス政府機関の資金でNPOを立ち上げ、オープンソース・ソフトウェアとして開発を続けております。基本的にはJavaで開発されたデスクトップアプリケーションで、Linux, Mac, Windowsの3つのプラットフォームをオフィシャルにサポートしています。
データ量の増加とエコシステムの拡大
2000年代前半、まだGoogleがAjaxを「再発見」する前にCytoscapeは生まれました。従って、主なターゲットはデスクトップマシンでした。当時は扱う必要のあったネットワーク(具体的には酵母等のタンパク質間相互作用ネットワーク)も、せいぜいノード数が数千のオーダーで、デスクトップアプリケーションで解析から可視化まで完結できるような世界を目指していました。しかしながら、実験手法の革新により怒涛のように増えていくデータに対し、デスクトップマシン一台で立ち向かうのは基本的に無理な世界がやって来ました。これは何も生命科学に限ったことではなく、ソーシャルネットワークにおいては、ディスクベースのグラフデータベースエンジンを利用しても厳しいような量のデータが出現してきていますし、そもそも解析から可視化までを全て一つのアプリケーションで行うのは難しいのが現実です。そこで必要になるのは:
- 拡張に対応したオープン性
- 外部のソフトウェアとの連携を考慮に入れたデータ入出力フォーマットへの対応
- ブラウザベースでデータを共有したい人々に向けてのウェブへの対応
- コア部分の機能へのスクリプトによる容易なアクセス
と言った機能です。以下ではこれらを一つ一つ見て行きます。
1. オープン性
Cytoscapeは、バージョン1の時からプラグイン(App)のサポートを行い、現在では200を超えるAppが世界各国で開発されています。ほとんどのものがオープンソースで、以下のサイトにて無償で公開されています:
コア部分ではEclipseと同じOSGiという仕組みを採用し、プラグイン同士の干渉を最小化しています。しかし、Appを作成するにはJavaの知識が必要で、ちょっとしたユーティリティーのようなものを軽量言語で書きたいというようなニーズには対応できていません。これが4に繋がります。
2. 外部との連携
Cytoscapeは優れた可視化プラットフォームですが、すでに他のソフトウェアに存在する解析アルゴリズムなどをCytoscape上で実装し直すというのはあまり賢い開発リソースの使い方ではありません。オープンソースの世界には、既にigraph、NetworkXと言った素晴らしいネットワーク解析ソフトウェアが存在するので、それらに用意されたアルゴリズムを利用して解析を行い、可視化はCytoscapeで、というような利用法はよく見られます。そういったニーズに対応するため、Cytoscapeは出来る限り幅広いデータフォーマットに対応しています:
- GML
- XGMML
- GraphML
- SIF
- CSV/TSVなどのテキスト
- Excelのワークブック
- Cyoscape.js(後述)形式のJSON
しかし、当然ながら、「ファイル経由ではなく、直接データをigraphからCytoscapeに送り込みたい」と言うニーズも存在するので、これも4に繋がります。
3. ウェブへの対応
データ可視化はとても巾の広い分野で、ハイエンドワークステーションでGPUを限界まで使いながら3次元画像を表示するようなものもあれば、スマートフォンでちょっとしたヒストグラムを表示するような可視化もあります。しかしひとつ言えるのは、可視化を皆で共有するという一点においては、デスクトップアプリケーションは向かないということです。クリックひとつで複雑な可視化結果をブラウザに表示できるJavaScriptベースのデータ可視化は、近年最も開発者が集まっている分野の一つで、D3.jsなどといったライブラリの名前はお聞きになったことがある方も多いでしょう。我々の関わっている生命科学分野でもブラウザベースのツールへの需要は高く、「Cytoscapeで作成したネットワーク図を何とかインタラクティブな可視化としてウェブで共有できないか?」と言うリクエストはよくいただいています。こういった問題に対応するために、カナダのトロント大学のチームがCytoscape.jsというグラフ可視化ライブラリを開発しました:
- Cytoscape.js - An open-source JavaScript graph theory library for analysis and visualisation
これはブラウザ上でのグラフの描画や解析に特化したライブラリで、モダンブラウザとNode.jsをサポートしています。ここで注意していただきたいのは、__Cytoscape.jsはウェブ版のCytoscapeそのものではない__という点です。あくまでこれは100% JavaScriptで書かれたライブラリで、ウェブアプリケーションそのものではありません。ウェブにおける多用なユースケースに対応するため、まずは安定したハイレベルな(抽象度の高い)APIを持ったライブラリを整備し、それを使った特定の問題に向けての各種アプリケーションを作っていこう、と言う方針で開発されています。しかしながら、Cytoscapeと言う名前を共有しているのにはわけがあります:
コアとなるコンセプトの共有
両者共に、Visual Styleと言うコンセプトを共有し、ユーザーが直接色や形を指定するのではなく、__データが直接visual variablesをコントロールする__と言う設計思想でデザインされています。つまり、ユーザーが指定するのはデータから視覚情報(色や形、大きさなど)へのマッピング関数であり、これは異なるデータに対しても再利用することが出来ます。因みに、著名ライブラリのD3.jsも名前の通り、この思想に基づいてデザインされています(「データがドキュメントの内容をコントロールする」)。
同じチームによる開発とメンテナンス
どちらも同じCytoscape Consortiumと言う非営利団体により開発されており、深いレベルでの両者の連携を目指しています。具体的には、
- Cytoscapeのネットワーク、テーブルのJSONとしての書き出しと読み込み
- デスクトップ版Visual StyleのCSSとしての書き出し
- セッション全体のHTML5ページとしての書き出し(v3.2にて対応予定)
といったものがあります。英文ですが、以下のサイトにこの辺りの連携について書いてあります。
今後は、YeomanのGeneratorを作成し、テンプレートコードから素早くグラフ可視化アプリケーションを作成していただけるように開発者向けの環境も整備していく予定です。
4. 軽量言語からのアクセス
データの増加により、データ解析のためのワークフローは複雑になる一方です。シンプルな解析と可視化を行うだけでしたら全てGUIから行ってもよいでしょうが、以下の様な場合はどうでしょう?
- 100以上のデータファイルを読み込ませ、それぞれのネットワークを可視化したものをPDFに落としたい
- IPython Notebook上でPandasを利用し、巨大なDataFrameをフィルタリングして作成したNetworkXのグラフオブジェクトを直接Cytoscapeに送り込みたい
- 最終的な可視化に至るまでの解析の過程そのものをコードとして記録したい
- データから動的に調節されたVisual Styleを生成し、ネットワーク図のsmall multiplesを作成し、時系列データの変化を見たい
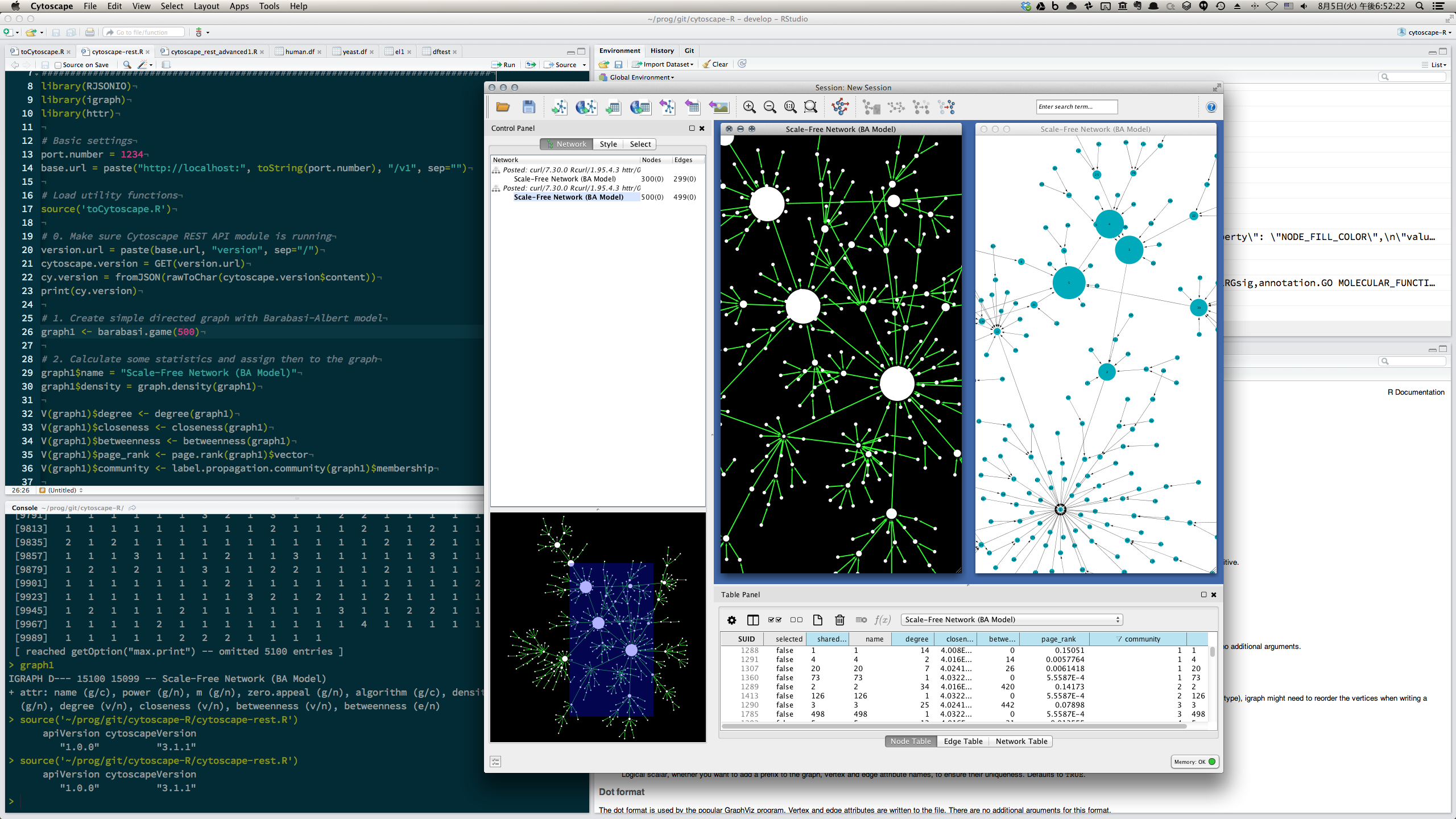
これらを根性でGUI上から行うことも可能ですが、あまりスマートなやり方とはいえません。こういった場合にはデータ解析コミュニティで人気のあるRStudioやIPython Notebookと言った環境から、直接Cytoscapeをコントロールできれば便利です。そこで我々が現在取り組んでいるのがCytoscapeのコアなデータオブジェクト(Network, Table, Visual Style)にCRUD操作を行うことが出来るRESTfulなAPIの整備です:
端的に言えば、これはRやPythonと言った事実上の標準的データ解析言語から、Cytoscapeの各種機能を利用できるようにして、大規模な解析ワークフローの中でもCytoscapeの各種機能をお使いいただけるようにするというのが目的です。こちらはまだ非常に実験的な要素が強く、ドキュメントも整備されていない状態ですが、日常的にRやPythonを使ってデータを解析するスクリプトをガリガリ書いておられる方がいらっしゃいましたら、ぜひご意見を伺いたいです。「R/Pythonからこんなことがしたいけど、出来る?」と言う簡単なユースケースでも参考になりますので、興味のある方はぜひ。
まだ全くドキュメントが無いですが、ぼちぼちサンプルも作り始めているので、興味のある方はどうぞ。ただしまだ実験段階のため、暫くは色々と変更が加わると思います:
R + igraph
IPython Notebook + NetworkX
- http://nbviewer.ipython.org/github/idekerlab/cy-rest-python/blob/develop/basic/CytoscapeREST_Basic1.ipynb
- http://nbviewer.ipython.org/github/idekerlab/cy-rest-python/blob/develop/basic/CytoscapeREST_Basic2.ipynb
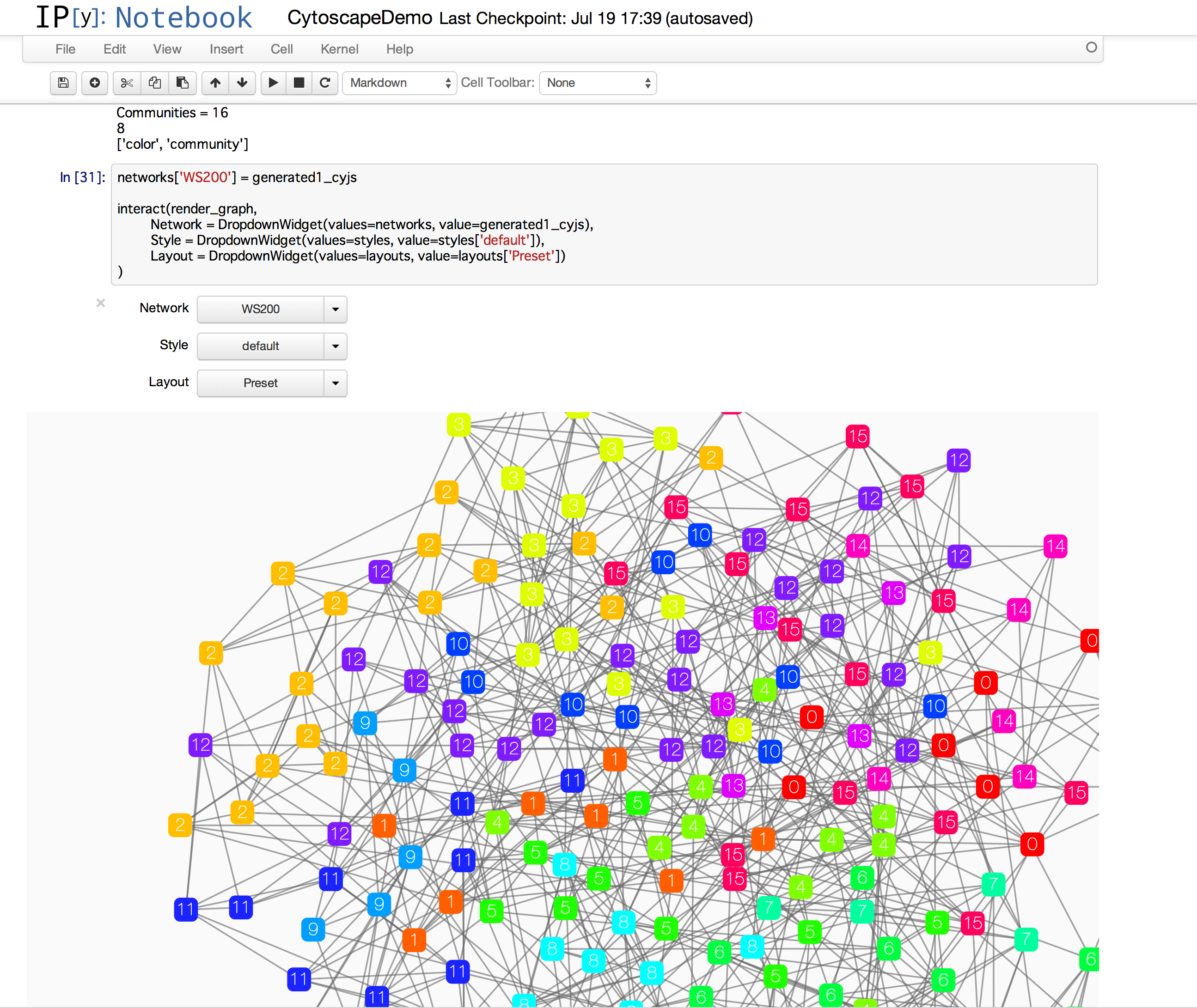
IPython Notebookへの埋め込みウィジェット
また、せっかくIPython Notebookを使って解析ワークフローを書き下せるようになるのだから、ノートブックの中でインタラクティブなネットワークビューアが走ったら便利では?と言う意見は以前から私の職場のユーザーに言われていましたので、そういうニーズにも対応しようとしています。これは、Cytoscape.jsの方を使って、ネットワークビューアをノートブックに埋め込んだ一例です:
現在、Python/R両コミュニティ共に、可視化ライブラリ関連は大幅な移行期にあり、今までのmatplotlib等の定番ライブラリから、もっとダイナミックな表現のできるJSベースの可視化ライブラリに移行しようとしています。そのような中で、Canvasベースのネットワーク描画ライブラリはまだ定番となるものがないようなので、この辺りも整備して、Cytoscape, Cytoscape.js, IPython Notebookなどを統合した、より便利なワークフローを構築できるようにしたいと考えています。
おわりに
駆け足でプロジェクトの現状だけ書きましたが、ここから先実際に使う場合には、どうしても英文を読んで頂く必要があります。その現状を変えるためにも、今後はできるだけ日本語の情報も増やしていきたいと考えております。基本チュートリアルの翻訳なども行いたいと思うので、実行のメドが立ちましたら、またお知らせいたします。
我々は非営利団体なので、いつでもコラボレーションは歓迎いたします。他のメンバーは英語とフランス語しか話せませんが、私が日本語の窓口になれますので、何か質問やリクエストがありましたら、Twitterなどでお知らせ下さい。もちろん日本語で大丈夫です :)
- 8/10/2014(日) : 補足記事を追加しました。
- 8/20/2014(水): 実際のチュートリアルをお探しの方のために、簡単なものを作成しました。Cytoscape最新版に準拠しています。
Keiichiro Ono
Cytoscape Consortium
National Resource for Network Biology