この記事は,Stan Advent Calender 2016の13日目の記事です。
RStudioでStanを実行する
Stanを実行する環境としてはRやPythonなど色々あるかと思いますが,私の周りではもっぱらRStanが多く,またRStudioを利用している人が多い印象です。実際RStudioにはStanを実行するために色々なものが準備されているのですが,まだあまり広まっていないように感じます。
そこで本記事では,知っておくと便利なTipsをいくつかご紹介します。なお,一部は記事公開時点(2016/12/13)ではPreview版RStudioを必要とするものがあります。ご了承ください。
stanファイルの編集
RStudioは実のところ色々なファイル形式に対応しており,.stanにも対応しています。しかし新規ファイル作成ではstan fileという項目はありません。なので以下のような手順を取ります:
- Text fileを新規で作成
- [File] - [SaveAs...]で,
kosaki.stanと拡張子に.stanをつけて保存
これでRStudioはこのファイルをstanファイルとして認識し,そのように扱うようになります。
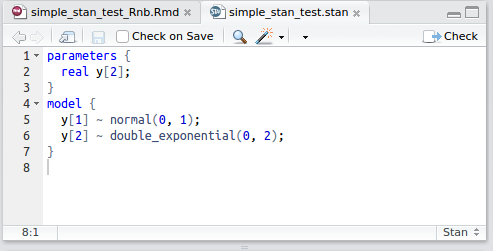
Stanのコードハイライトが有効になる
RStudioで.stanとして認識させると,Stanのコードハイライトが有効になります:
Stanのコードを編集する上で必須と言えるでしょう。
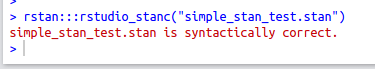
Stanコードの文法チェックができる
.stanを開いているpane(前述の画像参照)では,右上に「Check」というボタンが出てきます。これをクリックすると,Stanコードのシンタックスチェックが行われます。結果はConsoleに表示され,問題ないならば以下のようなメッセージが出ます:
もしシンタックスに問題がある場合,エラーを返すようになります。試しに一部セミコロンを取り去ってCheckすると,こうなります:
普段Rしか使わない人にとってはこの;を入れ忘れるというのはよくあるんじゃないのでしょうか。まずは一度この文法チェックをしてみるのをおすすめします。
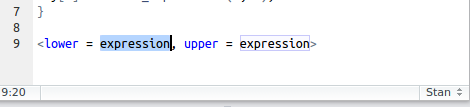
コードスニペットが使える
RStudioにはスニペット機能1があり,いろんな言語に対応しています。実はStanにも対応しており,.stanファイル内ではStan用のスニペットが動くようになります。例えば,<luと入力してShift&Tabキーを入力すると,以下のようになります:
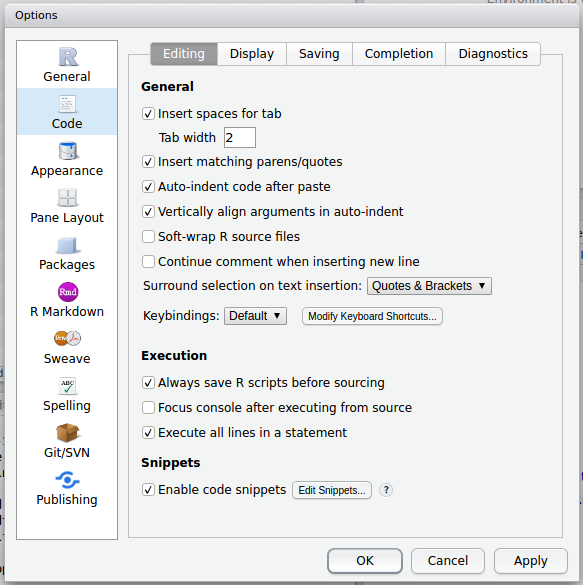
この後は選択部分を編集してTabキー,選択部分を編集してTabキーと進めばあっという間に編集できるかと思います。Stan用のスニペット一覧を確認するには,RStudioのメニューの[Tools] - [Global Options...]をクリックし,ダイアログボックス左側の[Code]をクリックします:
一番下のSnippetsから,**Edit Snippets...**のボタンをクリックしてEdit Snippetsを呼び出して,Stanを選択してください:
RmdファイルにStanコード直書き
Rでドキュメントを生成することを可能にするR Markdownは,他の言語エンジンも利用することができます。これについては本家の以下のサイトが参考になります:
knitr Language Engines - RStudio
対応する言語にはStanも含まれており,上記Webページのこの場所に説明があります。なお,記事作成時点(2016/12/13)において,安定版(v1.0.44)では全体を一度にknitする場合のみ有効です。しかしながら,Preview版(執筆時でv1.0.136)ではstanチャンクの実行にも対応しています。この記事ではPreview版であるv1.0.136を想定して説明します。
基本的な使い方
上記公式Webページにあるように,.stanファイルとして記述する内容をstanチャンクを埋め込めばOKです:
```{stan, output.var="kosaki"}
parameters {
real y[2];
}
model {
y[1] ~ normal(0, 1);
y[2] ~ double_exponential(0, 2);
}
```
このチャンクオプションにあるoutput.var=は必須で,指定した文字列で生成されたstanmodelオブジェクトを格納させます。この場合,kosakiというオブジェクトが生成され,以降のrチャンクでそれを指定してサンプリングなどを実行することになります:
```{r}
library(rstan)
fit <- sampling(kosaki)
print(fit)
```
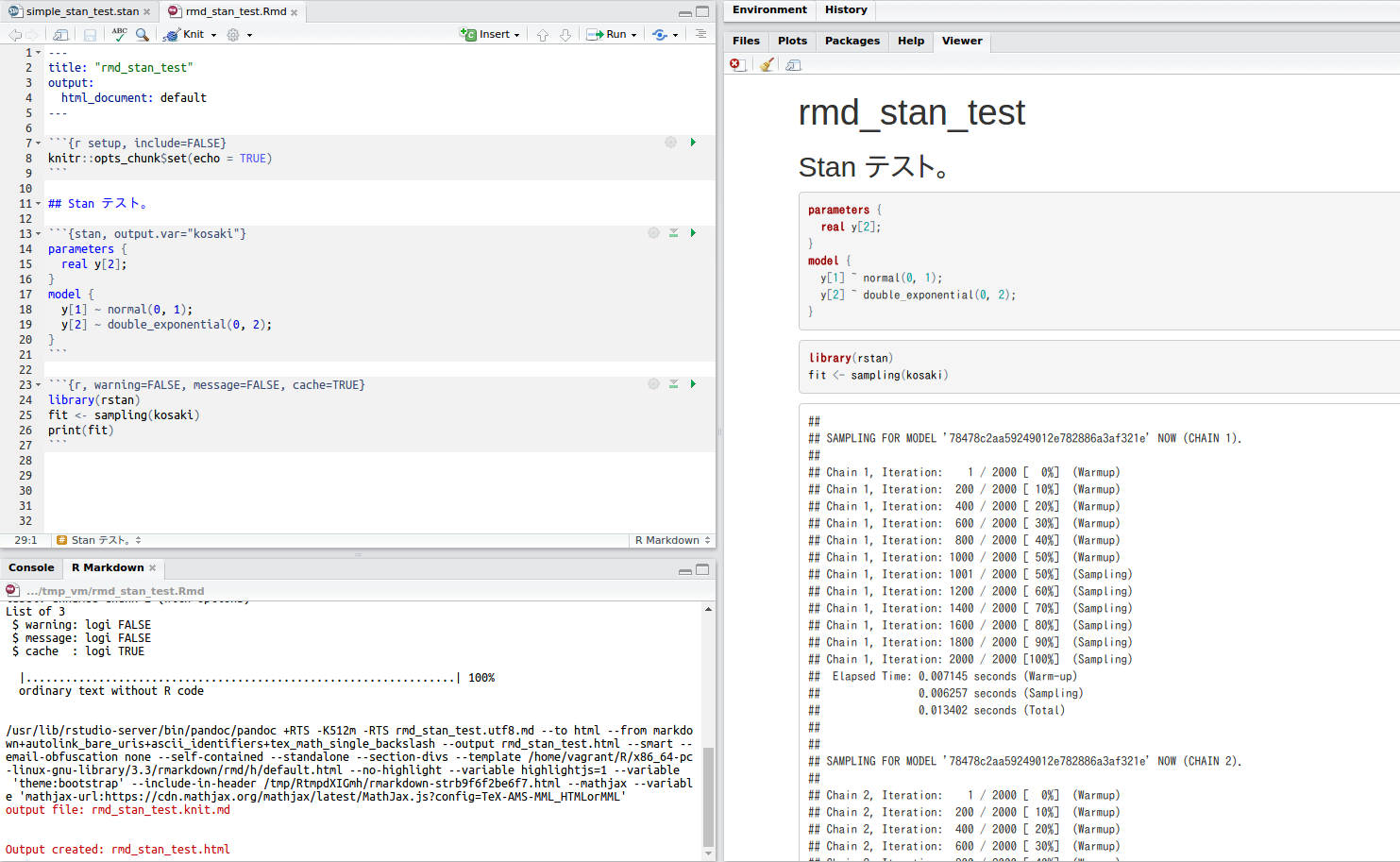
なお,このStanチャンク内ではStanのスニペットが有効です。これらを含むRmdファイルをKnit(あるいはrmarkdown::render)すれば,StanコードおよびRのコードに問題がなければ実行されるでしょう。試しに,以下のようなRmdファイルを準備してみます:
---
title: "rmd_stan_test"
output:
html_document: default
---
```{r setup, include=FALSE}
knitr::opts_chunk$set(echo = TRUE)
```
## Stan テスト。
```{stan, output.var="kosaki"}
parameters {
real y[2];
}
model {
y[1] ~ normal(0, 1);
y[2] ~ double_exponential(0, 2);
}
```
```{r, warning=FALSE, message=FALSE, cache=TRUE}
library(rstan)
fit <- sampling(kosaki)
print(fit)
```
これを適当なファイル名で保存してKnitすると,以下のような感じになります:
注意事項
- Stanチャンクで
cache=TRUEと指定すると,2回目以降でR Markdown側でエラーが発生し,cacheをクリアしないとうまくいかなくなる - 全体をKnitすると毎回stanコードのコンパイルが実行されたりするのでかなり時間がかかる
なので,手探りで色々やっていく場合,このままだと実用的ではないと思います2。Stanによるモデリングは,結構色々と試しながら実行することを考えると,これは致命的です。そこで次の手を使います。
stanチャンク単体で実行
R Markdownはチャンク単体で実行することも可能です。これはrチャンク以外でも有効です。チャンクを単体で実行した場合,それらはGlobal Environmentに入ります。なので気軽に再利用したりできますし,Rのコード評価に集中することができます。
「チャンクごとに実行(Run Current Chunk)」するには,チャンク部分右上にある右向き三角形ボタンをクリックすればOKです:
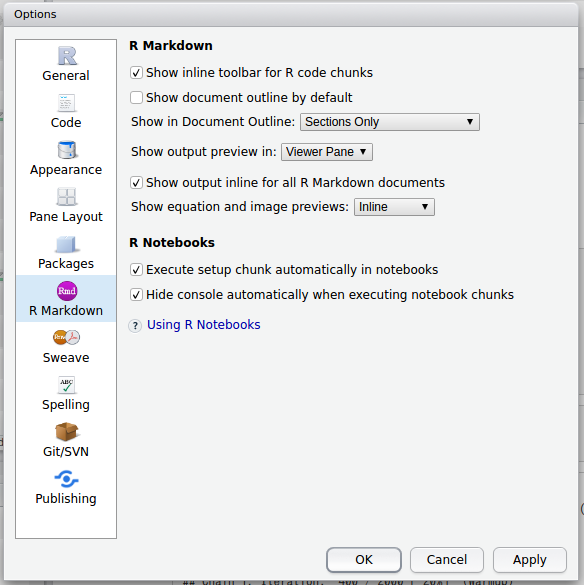
また,その隣にある下向き矢印は,「そのチャンクより前にある全てのチャンクを実行(Run All Chunks Above)」するボタンです。なおRStudioのデフォルト設定では,実行結果をチャンクのすぐ下に表示するようになっています。これを邪魔に感じるようであれば,設定ボタンで変更できます:
ここでChunk Output in Consoleを選択すれば,Consoleにだけ出力されるようになります。R Markdown全体の設定としてそのようにしたい場合は,[Tools] - [Global Options...]で,R Markdownの設定を変更してください:
R NotebookでStanコード直打ち
R NotebookもStan直打ちに対応しています3。基本的な使い方は上述のR Markdownと同様です。ただ,R NotebookでPreview(Knitのところのボタン)を使用すると,Stanチャンク内のコードはドキュメントとしては表示されません4。それ以外,つまりNotebook的な利用については基本問題なく動作しました。
結構荒く説明したので,不明な点がありましたらこの記事のコメントや,執筆者のTwitterアカウント(@kazutan)にお問い合わせください。
Enjoy!
-
RStudioのスニペット機能にはこちらの記事で軽く紹介しています。詳しくはリンク先の記事およびそこで紹介している記事を参照してください。 ↩
-
R Markdownにはキャッシュ機能があるのですが,Stanエンジンの分はどうも毎回コンパイルを実行してしまい,これ自体はキャッシュとして残ってくれないっぽいです。もっと色々調べると解決策があるような気がしますが,そこまでの余裕もないですし,チャンク単体で実行していけば気にならないので放置することにしました…。 ↩
-
これもRStudio v1.0.44では未対応で,Preview版(v1.0.136)で動作確認しました。 ↩
-
これはバグなのか何なのかはちょっとわからないです。とはいえR Notebookの内部を考えるとそれも仕方ないかなぁという気もしますが,未検証なので明言は避けます。 ↩