はじめに
機械学習をやってみて、すぐに困るのは訓練用のデータセットを用意することだと思う。最初にMINISTか何かでテストして、次にせいぜい論理演算を学習させて、さて次にどうしようかと悩むことになる。なんか画像の分類とかやりたいと思っても、落ちてるデータセットでやっても面白くないし、自分で画像を用意する場合はラベルをつけるのが死ぬほど面倒くさい。
というわけで、自分で適当にデータが用意できて、それなりに非自明な分類として、パイこね変換と擬似乱数の分類をやってみる。
ソースはここにおいておく。
https://github.com/kaityo256/chainer_bakermap
Chainerのバージョンは2.0.1。
注意:本記事を書いたのは機械学習のド素人です。
目的
以下のことを目標とする。
- 教師用データを簡単に作れること
- Pythonで学習したデータをC++に読み込ませること
- Pythonでの結果とC++での結果が比較できるようにすること
データセット
データは、一次元数列${v_n}$として与える。ひとつはPython標準のrandom.random()で与え、もうひとつは初期値のみrandom.random()で、それ以降は
v = 3.0 * v - int(3.0*v)
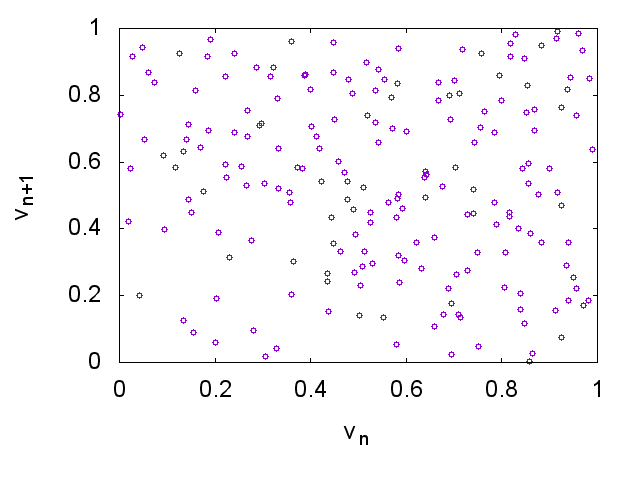
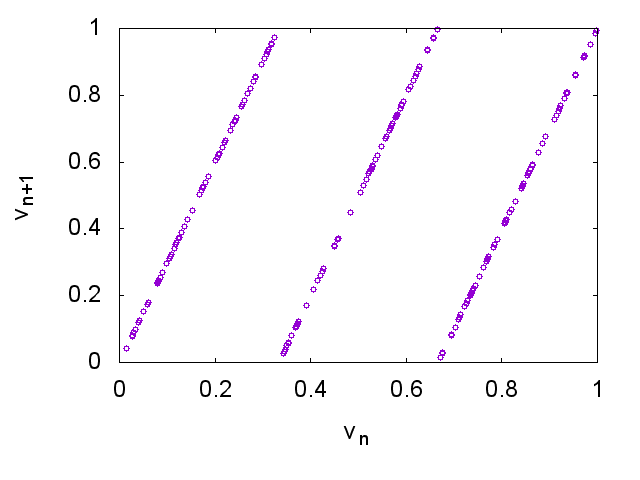
で与える。これはいわゆるパイこね変換(baker's map)で、一見乱数っぽいんだけど、$(v_n, v_{n+1})$をプロットすると違いがわかる。
まず、標準の乱数を使った場合は特に構造が出ない。
対して、パイこね変換の場合にはすぐにそれとわかる。
両者の違いをニューラルネットが学習によって見抜けるようになりますか?という問題。これだと教師データを作るのが容易で、サイズの調整なんかも非常に簡単。標準の乱数も、厳密には周期や構造があるのだけれど、それは200個の範囲ではまったく見えないはずなので、その範囲ではランダムに見えるはず。
設定
とりあえずこんな設定で学習させる。
- 与えるデータ列の長さ:200
- 全データ数: 20000
- そのうち半分が乱数、半分がパイこね変換
- 90%を訓練用、10%をテスト用に使う
- エポック: 100
- バッチサイズ: 100
- 入力が乱数だと1、パイこね変換なら0を出力させる(二値分類)
数字は全部適当に決めた。
データの準備
Chainerの最初の関門(ってほどでもないけど)はデータの準備だと思う。詳細は別記事を参照してほしいが、要するに
- 入力をNumpyの配列(
numpy.float32)の配列で与える(Numpyオブジェクトの配列となる)。 - 出力(ラベル)を、Numpyの整数配列(
numpy.int32)で与える(Numpyオブジェクトひとつを与える)
そして、入力、出力それぞれをx,yとして
dataset = chainer.datasets.TupleDataset(x,y)
とすれば、Chainerに食わせられるデータセットフォーマットになる。
あまり長くないので、データを作るモジュールを掲載しておく。
import random
import numpy as np
import chainer
def make_baker(n):
a = []
x = random.random()
for i in range(n):
x = x * 3.0
x = x - int(x)
a.append(x)
return a
def make_random(n):
a = []
for i in range(n):
a.append(random.random())
return a
def make_data(ndata,units):
data = []
for i in range(ndata):
a = make_baker(units)
data.append([a,0])
for i in range(ndata):
a = make_random(units)
data.append([a,1])
return data
def make_dataset(ndata,units):
data = make_data(ndata,units)
random.shuffle(data)
n = len(data)
xn = len(data[0][0])
x = np.empty((n,xn),dtype=np.float32)
y = np.empty(n,dtype=np.int32)
for i in range(n):
x[i] = np.asarray(data[i][0])
y[i] = data[i][1]
return chainer.datasets.TupleDataset(x,y)
def main():
dataset = make_dataset(2,3)
print(dataset)
if __name__ == '__main__':
random.seed(1)
np.random.seed(1)
main()
中身の理解は難しくないと思う。一度、(入力、出力)の組をリストにしたdataを作り、それをnumpy形式に変換してdatasetにする流れ。あとは
import data
units = 200
ndata = 10000
dataset = data.make_dataset(ndata,units)
などとすれば、Chainerに食わせられるデータセットを得る。make_data関数だけ適切書き換えれば、どんなデータにも対応できるはず。
モデルの設定
まず、Chainerのモデルを適当にラップしたクラスを作った。こんな感じ。
import chainer
import chainer.functions as F
import chainer.links as L
import collections
import struct
from chainer import training
from chainer.training import extensions
class MLP(chainer.Chain):
def __init__(self, n_units, n_out):
super(MLP, self).__init__(
l1 = L.Linear(None, n_units),
l2 = L.Linear(None, n_out)
)
def __call__(self, x):
return self.l2(F.relu(self.l1(x)))
class Model:
def __init__(self,n_unit):
self.unit = n_unit
self.model = L.Classifier(MLP(n_unit, 2))
def load(self,filename):
chainer.serializers.load_npz(filename, self.model)
def save(self,filename):
chainer.serializers.save_npz(filename, self.model)
def predictor(self, x):
return self.model.predictor(x)
def get_model(self):
return self.model
def export(self,filename):
p = self.model.predictor
l1W = p.l1.W.data
l1b = p.l1.b.data
l2W = p.l2.W.data
l2b = p.l2.b.data
d = bytearray()
for v in l1W.reshape(l1W.size):
d += struct.pack('f',v)
for v in l1b:
d += struct.pack('f',v)
for v in l2W.reshape(l2W.size):
d += struct.pack('f',v)
for v in l2b:
d += struct.pack('f',v)
open(filename,'w').write(d)
ごちゃごちゃ書いてあるけど、使い方としては、
m = Model(units) # モデルのラッパークラス作成
model = m.get_model() # モデルオブジェクトを取得(訓練に使う)
m.save("baker.model") # モデルの保存(シリアライズ)
m.load("baker.model") # モデルの読込(デシリアライズ)
m.export("baker.dat") # C++用にエクスポート
として使う。
学習
学習は、Modelクラスのmodelを受け取れば、あとはChainerのサンプルそのままなので特に問題ないと思う。とりあえずtrain.pyを見れば、そのままなのがわかる。ただ、学習後にモデルをシリアライズしている。
学習後のテスト
学習後のモデルをテストする、test.pyはこんな感じ。
from model import Model
import numpy as np
import random
import data
import math
def main():
ndata = 1000
unit = 200
model = Model(unit)
model.load("baker.model")
d = data.make_data(ndata,unit)
x = np.array([v[0] for v in d], dtype=np.float32)
y = model.predictor(x).data
r = [np.argmax(v) for v in y]
bs = sum(r[:ndata])
rs = sum(r[ndata:])
print("Check Baker")
print "Success/Fail",ndata-bs,"/",bs
print("Check Random")
print "Success/Fail",rs,"/",ndata-rs
def test():
unit = 200
model = Model(unit)
model.load("baker.model")
a = []
for i in range(unit):
a.append(0.5)
x = np.array([a], dtype=np.float32)
y = model.predictor(x).data
print(y)
if __name__ == '__main__':
random.seed(2)
np.random.seed(2)
test()
main()
単にModelクラスのインスタンスを作って、デシリアライズしてテストしているだけ1。
実行結果はこんな感じになる。
$ python test.py
[[-0.84465003 0.10021734]]
Check Baker
Success/Fail 929 / 71
Check Random
Success/Fail 913 / 87
最初の
[[-0.84465003 0.10021734]]
という出力は「200個全部0.5」というデータを食わせたときの重みを出力している。これは、0、すなわちパイこね変換と認識する重みが-0.84465003、乱数と認識する重みが0.10021734であるということ。つまり、定数を食わせたときは、ランダムだと認識する2。これは後でC++で読み込んだモデルが正しく機能しているかどうかのチェックに使う。
その後の
Check Baker
Success/Fail 929 / 71
という出力は、パイこね変換の数列を1000セット食わせたところ、929セットはパイこね変換と認識し、71セットをランダムと誤認識した、ということ。
さらにその後の
Check Random
Success/Fail 913 / 87
は、乱数を1000セット食わせて、913セットは乱数と正しく認識した、ということ。
エクスポート+C++へのインポート
エクスポートとC++へのインポートについては別記事参照。エクスポートはラッパークラスに任せてあるので、簡単にできる。
from model import Model
def main():
unit = 200
model = Model(unit)
model.load("baker.model")
model.export("baker.dat")
if __name__ == '__main__':
main()
baker.modelを読み込んで、baker.datを吐いている。
インポートも簡単だが、後の便利のためにクラス化しておこう。こんな感じ。
# pragma once
# include <iostream>
# include <fstream>
# include <vector>
# include <math.h>
# include <algorithm>
//------------------------------------------------------------------------
typedef std::vector<float> vf;
//------------------------------------------------------------------------
class Link {
private:
vf W;
vf b;
float relu(float x) {
return (x > 0) ? x : 0;
}
const int n_in, n_out;
public:
Link(int in, int out) : n_in(in), n_out(out) {
W.resize(n_in * n_out);
b.resize(n_out);
}
void read(std::ifstream &ifs) {
ifs.read((char*)W.data(), sizeof(float)*n_in * n_out);
ifs.read((char*)b.data(), sizeof(float)*n_out);
}
vf get(vf x) {
vf y(n_out);
for (int i = 0; i < n_out; i++) {
y[i] = 0.0;
for (int j = 0; j < n_in; j++) {
y[i] += W[i * n_in + j] * x[j];
}
y[i] += b[i];
}
return y;
}
vf get_relu(vf x) {
vf y = get(x);
for (int i = 0; i < n_out; i++) {
y[i] = relu(y[i]);
}
return y;
}
};
//------------------------------------------------------------------------
class Model {
private:
Link l1, l2;
public:
const int n_in, n_out;
Model(int in, int n_units, int out):
n_in(in), n_out(out),
l1(in, n_units), l2(n_units, out) {
}
void load(const char* filename) {
std::ifstream ifs(filename);
l1.read(ifs);
l2.read(ifs);
}
vf predict(vf &x) {
return l2.get(l1.get_relu(x));
}
int argmax(vf &x) {
vf y = predict(x);
auto it = std::max_element(y.begin(), y.end());
auto index = std::distance(y.begin(), it);
return index;
}
};
//------------------------------------------------------------------------
これで、
# include "model.hpp"
int
main(void){
const int n_in = 200;
const int n_units = 200;
const int n_out = 2;
Model model(n_in, n_units, n_out);
model.load("baker.dat");
}
としてモデルを読み込める。
インポートテスト
まず、同じものを食わせてまったく同じ重みを吐くかどうか試す。
こんなコードを書こう。
void
test(Model &model) {
vf x;
for (int i = 0; i < model.n_in; i++) {
x.push_back(0.5);
}
vf y = model.predict(x);
printf("%f %f\n", y[0], y[1]);
}
ただし、
typedef std::vector<float> vf;
である。実行結果は
-0.844650 0.100217
となって、ちゃんとPythonの結果と一致することがわかる。
さらに、パイこね変換と乱数を食わせた場合の正答率も調べる。
int
test_baker(Model &model) {
static std::mt19937 mt;
std::uniform_real_distribution<float> ud(0.0, 1.0);
vf x;
float v = ud(mt);
for (int i = 0; i < model.n_in; i++) {
x.push_back(v);
v = v * 3.0;
v = v - int(v);
}
return model.argmax(x);
}
//------------------------------------------------------------------------
int
test_random(Model &model) {
static std::mt19937 mt;
std::uniform_real_distribution<float> ud(0.0, 1.0);
vf x;
for (int i = 0; i < model.n_in; i++) {
x.push_back(ud(mt));
}
return model.argmax(x);
}
//------------------------------------------------------------------------
int
main(void) {
const int n_in = 200;
const int n_units = 200;
const int n_out = 2;
Model model(n_in, n_units, n_out);
model.load("baker.dat");
test(model);
const int TOTAL = 1000;
int bn = 0;
for (int i = 0; i < TOTAL; i++) {
bn += test_baker(model);
}
std::cout << "Check Baker" << std::endl;
std::cout << "Success/Fail:" << (TOTAL - bn) << "/" << bn << std::endl;
int rn = 0;
for (int i = 0; i < TOTAL; i++) {
rn += test_random(model);
}
std::cout << "Check Random" << std::endl;
std::cout << "Success/Fail:" << rn << "/" << (TOTAL - rn) << std::endl;
}
それぞれの実行結果はこんな感じ。
Check Baker
Success/Fail:940/60
Check Random
Success/Fail:923/77
だいたい似たような正答率になってるようだ。
まとめ
Chainerを使って、パイこね変換によって得られた数列と、標準乱数を見分けるテストをしてみた。もっと楽勝で見分けられると思ったけれど、3層で200ユニット/層だとこんなものなんですかね?とりあえずPythonで学習→C++で使うという流れは作ることができたので、これでいろいろ応用してみたい。
参考
自分が書いた記事で恐縮だけど。