この記事では、既存の機械学習を用いて3次元の空間を分類するコードを載せる。
概要
以下の2つのサイトから、引用している。
-
CS231n Deep Learning for Computer Vision
2次元の渦巻きデータから、領域分類を行う機械学習を示している。1
-
ニューラルネットのうずまきサンプル
上記のサイトを参考にして、隠れ層を2層としている。2
今回、2つ目の記事2を参考にして、3次元のデータを学習させる。
次に、学習したニューラルネットワークが、同じ次元(3次元)の他のデータを正しく領域分類できるかを調べる。
実行環境
Ubuntu 20.04.6 LTS
python 3.8.10
git vesion 2.25.1
vscode
$ cat /etc/lsb-release
DISTRIB_ID=Ubuntu
DISTRIB_RELEASE=20.04
DISTRIB_CODENAME=focal
DISTRIB_DESCRIPTION="Ubuntu 20.04.6 LTS"
$ python --version
Python 3.8.10
$ git --version
git version 2.25.1
$ pip list
Package Version
---------------------- -----------
antlr4-python3-runtime 4.9.3
contourpy 1.1.1
cycler 0.12.1
fonttools 4.56.0
hydra-core 1.3.2
importlib-resources 6.4.5
kiwisolver 1.4.7
matplotlib 3.7.5
narwhals 1.34.0
numpy 1.24.4
omegaconf 2.3.0
packaging 24.2
pandas 2.0.3
pillow 10.4.0
pip 20.0.2
pkg-resources 0.0.0
plotly 6.0.1
pyparsing 3.1.4
python-dateutil 2.9.0.post0
pytz 2025.2
PyYAML 6.0.2
setuptools 44.0.0
six 1.17.0
tzdata 2025.2
zipp 3.20.2
蛇足
以下のテキストをテキストファイルに保存して、
(ファイル名なんでもいいが今回は requierments.txt)
antlr4-python3-runtime==4.9.3
contourpy==1.1.1
cycler==0.12.1
fonttools==4.56.0
hydra-core==1.3.2
importlib-resources==6.4.5
kiwisolver==1.4.7
matplotlib==3.7.5
narwhals==1.34.0
numpy==1.24.4
omegaconf==2.3.0
packaging==24.2
pandas==2.0.3
pillow==10.4.0
plotly==6.0.1
pyparsing==3.1.4
python-dateutil==2.9.0.post0
pytz==2025.2
PyYAML==6.0.2
six==1.17.0
tzdata==2025.2
zipp==3.20.2
pip install -r requirements.txt
同じライブラリを一括インストールできる。
データの作成(3次元)
引用元の渦巻きをz方向にそのまま伸ばすとあまり拡張したことにならない。よって、3次元のデータは3重螺旋の構造をとった。
import numpy as np
import matplotlib.pyplot as plt
import plotly.express as px
import plotly.graph_objects as go
from matplotlib import cm
# Generate sample data 乱数のシード
np.random.seed(0) # 再現性を確保するために乱数のシードを固定
# パラメータの設定
N = 30 # クラスごとのデータ点数
D = 3 # データの次元数
K = 3 # クラス数
R = 0.5 # 基本半径
R_NOISE = 0.2 # 半径に加えるノイズの強さ
# データとラベルを格納する配列を初期化
X = np.zeros((N*K, D)) # データ点の座標を格納する配列
y = np.zeros((N*K), dtype='int') # クラスラベルを格納する配列
# 各クラスのデータを生成
for j in range(K):
ix = range(N*j, N*(j+1)) # クラス j に対応するインデックス範囲
r = R + np.random.randn(N) * R_NOISE # 半径にノイズを加える
t = np.linspace(j*2*np.pi/K, j*2*np.pi/K + 2.5*np.pi, N) + j*8*np.pi/K # 角度(スパイラルの形状を作る)
z = np.linspace(0.0, 2, N) # z軸方向の値(線形に増加)
# データ点を生成(スパイラル状に配置)
X[ix] = np.c_[r*np.sin(t), r*np.cos(t), z]
y[ix] = j # クラスラベルを設定
# 3D散布図を作成
fig = px.scatter_3d(
x = X[:, 0], # x座標
y = X[:, 1], # y座標
z = X[:, 2], # z座標
title = "3D Spiral Dataset", # グラフのタイトル
labels = {'x': 'X', 'y': 'Y', 'z': 'Z'}, # 軸ラベル
)
# グラフのレイアウトを調整
fig.update_layout(
scene = dict(
xaxis=dict(range = [-1.2, 1.2], nticks = 5), # x軸の範囲と目盛り数
yaxis=dict(range = [-1.2, 1.2], nticks = 5), # y軸の範囲と目盛り数
zaxis=dict(range = [-0.5, 2.5], nticks = 5), # z軸の範囲と目盛り数
)
)
# データ点のプロット設定
fig.update_traces(
marker = dict(
size = 5, # マーカーサイズ
color = y, # クラスラベルに基づく色付け
colorscale = 'jet'
)
)
# グラフをHTMLファイルとして保存
fig.write_html('spiral_dataset_3d.html') # 出力ファイル名
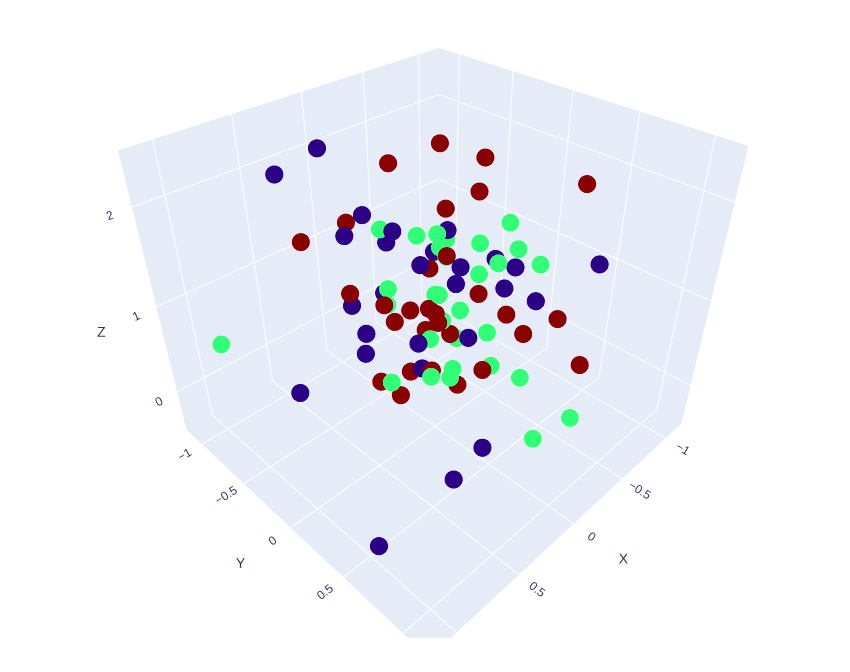
以下plotlyで出力したデータセットの図
https://ecalj.sakura.ne.jp/data/educational/spiral_dataset.html
螺旋であることがわかりにくいが、螺旋になっている。
これは、螺旋構造にした後、半径方向にランダムな値を加えているためだ。
ニューラルネットワークの学習
2つ目のコードより引用した、隠れ層が2層からなるニューラルネットワークである。
この記事上では、詳しいニューラルネットワークの説明は省く。
自分が参考にした動画を以下を添付する。
https://www.youtube.com/watch?v=tc8RTtwvd5U 3
https://www.youtube.com/watch?v=0AX3KSKjyog 3
https://www.youtube.com/watch?v=SgBDx8DqBZw 3
h1 = 100 # size of hidden layer1
h2 = 50 # size of hidden layer2
#initialization
W = 0.01 * np.random.randn(D,h1)
b = np.zeros((1,h1))
W1 = 0.01 * np.random.randn(h1,h2)
b1 = np.zeros((1,h2))
W2 = 0.01 * np.random.randn(h2,K)
b2 = np.zeros((1,K))
print(' W.shape b.shape',W.shape,b.shape)
print('W1.shape b1.shape',W1.shape,b1.shape)
print('W2.shape b2.shape',W2.shape,b2.shape)
# Set some hyperparameters
step_size = 1e-0
reg = 1e-3 # regularization strength
# Gradient descent loop
ndata = X.shape[0]
print('training data',ndata)
for i in range(1000):
hidden_layer1 = np.maximum(0, np.dot(X, W) + b) # X ---> hidden layer1
hidden_layer2 = np.maximum(0, np.dot(hidden_layer1, W1) + b1) # hidden layer1 ---> hidden layer2
# evaluate class scores, [N x K]
scores = np.dot(hidden_layer2, W2) + b2 # hidden layer2 ---> scores (logit)
# compute the class probabilities by softmax function
exp_scores = np.exp(scores)
probs = exp_scores / np.sum(exp_scores, axis=1, keepdims=True) # [N x K]
# compute the loss: average cross-entropy loss and regularization
correct_logprobs = np.array([-np.log(probs[i,y[i]]) for i in range(ndata)]) #i番目のデータの正解確率(対数をとって−1倍する)。
if(i==1):
print('probs.shape=',probs.shape)
print('corect_logprobs.shape=',correct_logprobs.shape)
# loss function
data_loss = np.sum(correct_logprobs)/ndata #正解確率(-logしたもの)の平均値
reg_loss = 0.5*reg*np.sum(W*W) + 0.5*reg*np.sum(W1*W1)+ 0.5*reg*np.sum(W2*W2) #正規化関数(Wなどの値を小さくするようにする)
loss = data_loss + reg_loss
if i % 100 == 0: print('iteration {}: loss {}'.format(i, loss))
# compute the gradient on scores
dscores = probs
for i in range(ndata):
dscores[i,y[i]] -= 1
dscores /= ndata
dW2 = np.dot(hidden_layer2.T, dscores) # backpropate the gradient to the parameters
db2 = np.sum(dscores, axis=0, keepdims=True) # first backprop into parameters W2 and b2
dhidden2 = np.dot(dscores, W2.T)
dhidden2[hidden_layer2 <= 0] = 0 # backprop the ReLU non-linearity
dW1 = np.dot(hidden_layer1.T, dhidden2)
db1 = np.sum(dhidden2, axis=0, keepdims=True)
dhidden1 = np.dot(dhidden2, W1.T)
dhidden1[hidden_layer1 <= 0] = 0
dW = np.dot(X.T, dhidden1)
db = np.sum(dhidden1, axis=0, keepdims=True)
# add regularization gradient contribution
dW2 += reg * W2
dW1 += reg * W1
dW += reg * W
# perform a parameter update
W += -step_size * dW
b += -step_size * db
W1 += -step_size * dW1
b1 += -step_size * db1
W2 += -step_size * dW2
b2 += -step_size * db2
# 学習済みモデルの保存
np.savez('trained_model_parameters.npz',
W=W, b=b,
W1=W1, b1=b1,
W2=W2, b2=b2)
このコードで入力した3重螺旋のデータを学習させる。2
学習データの確認
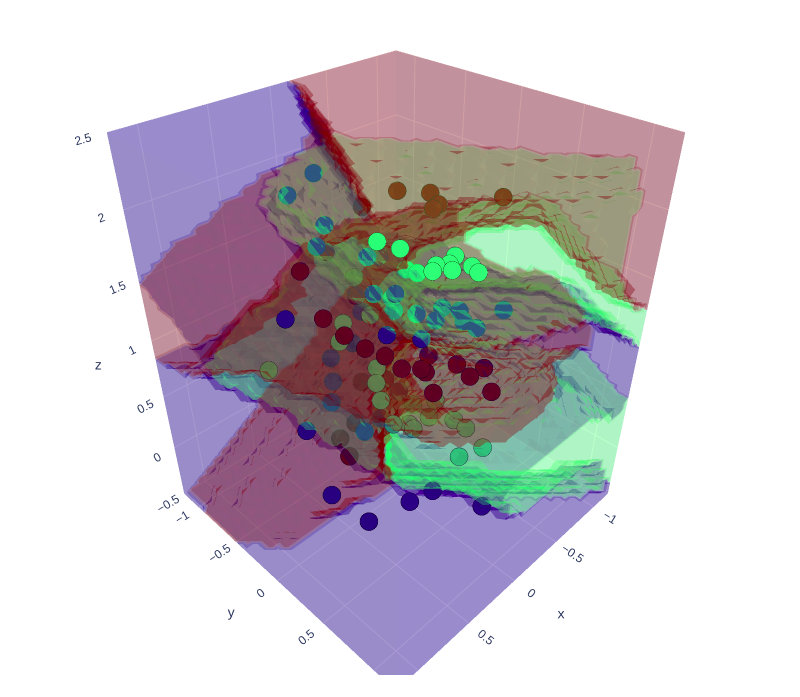
領域を分類して元のデータセットに重ねて表示させる。
領域分類が正しく機能しているかを確認する。
# === 3D空間の塗り分け予測可視化(元データも重ねる) ===
# グリッド範囲と解像度
x_range = np.linspace(-1.2, 1.2, 30)
y_range = np.linspace(-1.2, 1.2, 30)
z_range = np.linspace(-0.5, 2.5, 60)
xx, yy, zz = np.meshgrid(x_range, y_range, z_range)
grid_points = np.c_[xx.ravel(), yy.ravel(), zz.ravel()]
# 各点に対して予測
h1_grid = np.maximum(0, np.dot(grid_points, W) + b)
h2_grid = np.maximum(0, np.dot(h1_grid, W1) + b1)
scores_grid = np.dot(h2_grid, W2) + b2
pred_grid = np.argmax(scores_grid, axis=1)
pred_3d = pred_grid.reshape(xx.shape)
# クラスごとの色を、matplotlibの 'jet' カラーマップで対応
unique_classes = np.unique(y)
num_classes = len(unique_classes)
# jet カラーマップから [0,1] に正規化した色を取り出す
cmap = cm.get_cmap('jet', num_classes)
class_colors = {
cls: f'rgb({int(r*255)}, {int(g*255)}, {int(b*255)})'
for cls, (r, g, b, _) in zip(unique_classes, cmap(np.linspace(0, 1, num_classes)))
}
# 描画用フィギュア
fig_combined = go.Figure()
# クラスごとの領域描画(Isosurface)
for cls in unique_classes:
mask = (pred_3d == cls).astype(np.int8)
fig_combined.add_trace(go.Isosurface(
x = xx.ravel(), y = yy.ravel(), z = zz.ravel(),
value = mask.ravel(),
isomin = 0.5, isomax = 1.0,
opacity = 0.2,
surface_count = 1,
colorscale = [[0, class_colors[cls]], [1, class_colors[cls]]],
showscale = False,
name = f"Class {cls}"
))
# 元の学習データ(正解ラベルに基づいて色付け)
fig_combined.add_trace(go.Scatter3d(
x = X[:, 0], y = X[:, 1], z = X[:, 2],
mode = 'markers',
marker = dict(
size = 5,
color = y,
colorscale = 'Jet',
line = dict(width=0.5, color='black'),
opacity = 1.0,
showscale = False
),
name = 'Training Data'
))
# レイアウト調整
fig_combined.update_layout(
title = '3D Region Classification',
scene = dict(
xaxis = dict(range=[-1.2, 1.2]),
yaxis = dict(range=[-1.2, 1.2]),
zaxis = dict(range=[-0.5, 2.5])
)
)
# 書き出し
fig_combined.write_html("spiral_net_3d_with_dataset.html")
出力したファイルが以下より
https://ecalj.sakura.ne.jp/data/educational/spiral_region_classification_with_dataset.html
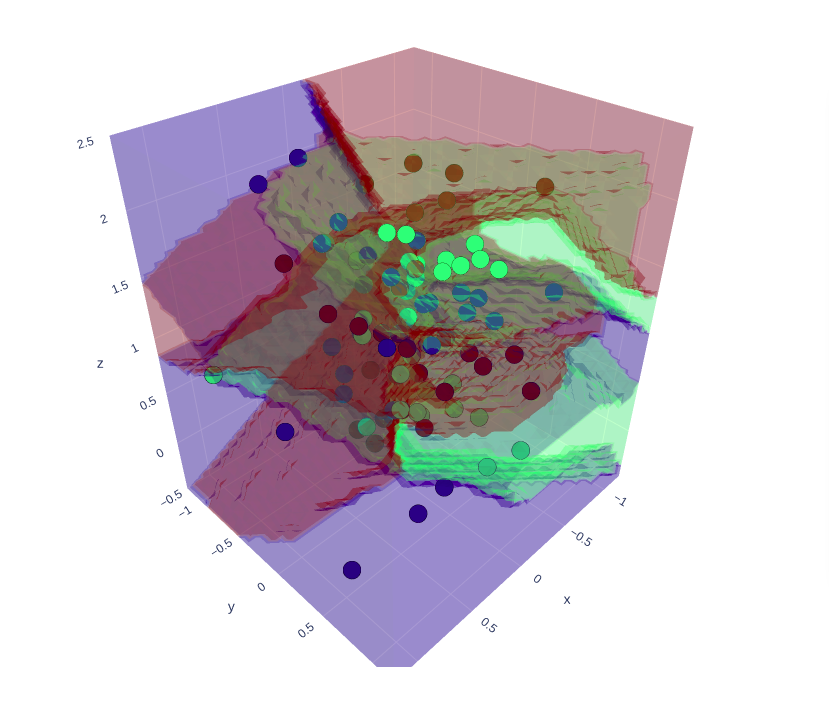
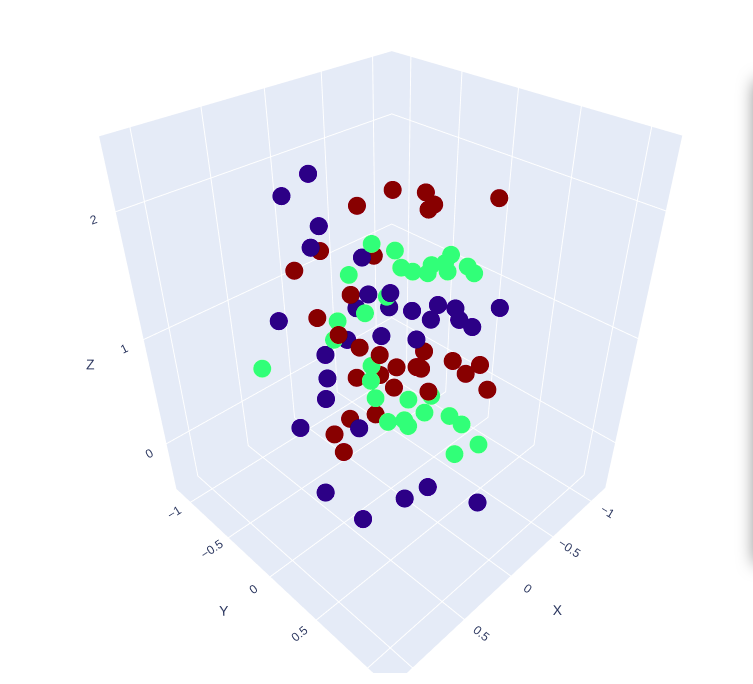
学習データを利用して他の3Dデータも試してみる。
学習したデータからパラメータを変えた他の3Dデータも試す。
import numpy as np
import plotly.graph_objects as go
import matplotlib
import plotly.express as px
from matplotlib import cm
# === 新しい3Dスパイラルデータを生成(あなたのコード) ===
# Generate sample data 乱数のシード
np.random.seed(0)
N = 30 # number of points per class
D = 3 # dimensionality
K = 3 # number of classes
R = 0.5 # radius
R_NOISE = 0.4 # noise
X = np.zeros((N*K,D))
y = np.zeros((N*K),dtype = 'int')
# z = np.zeros((N*K),dtype = 'int')
for j in range(K):
ix = range(N*j,N*(j+1))
r = R + np.random.randn(N) * R_NOISE # radius
t = np.linspace(j*2*np.pi/K, j*2*np.pi/K + 3*np.pi, N) + j*8*np.pi/K # theta
z = np.linspace(0.0,2,N)
X[ix] = np.c_[r*np.sin(t), r*np.cos(t), z] #データ点
y[ix] = j #正解ラベル K = 0,1,2
# z[ix] = j #正解ラベル K = 0,1,2
fig = px.scatter_3d(
x = X[:, 0],
y = X[:, 1],
z = X[:, 2],
title = "3D Spiral Dataset",
labels = {'x':'X','y':'Y','z':'Z'},
)
fig.update_layout(
scene = dict(
xaxis = dict(range = [-1.2, 1.2], nticks = 5),
yaxis = dict(range = [-1.2, 1.2], nticks = 5),
zaxis = dict(range = [-0.5, 2.5], nticks = 5),
)
)
fig.update_traces(
marker = dict(
size = 5,
color = y,
colorscale = 'jet'
)
)
fig.write_html('spiral_dataset_more_ramdomly.html')
# === 学習済みモデルの読み込み ===
data = np.load('trained_model_parameters.npz')
W = data['W']
b = data['b']
W1 = data['W1']
b1 = data['b1']
W2 = data['W2']
b2 = data['b2']
# === 新データへの順伝播による予測 ===
h1 = np.maximum(0, np.dot(X, W) + b)
h2 = np.maximum(0, np.dot(h1, W1) + b1)
scores = np.dot(h2, W2) + b2
predicted_class = np.argmax(scores, axis = 1)
accuracy = np.mean(predicted_class == y)
# === 可視化(予測クラスによって色分け) ===
# === 3D空間の塗り分け予測可視化(元データも重ねる) ===
# グリッド範囲と解像度
x_range = np.linspace(-1.2, 1.2, 30)
y_range = np.linspace(-1.2, 1.2, 30)
z_range = np.linspace(-0.5, 2.5, 60)
xx, yy, zz = np.meshgrid(x_range, y_range, z_range)
grid_points = np.c_[xx.ravel(), yy.ravel(), zz.ravel()]
# 各点に対して予測
h1_grid = np.maximum(0, np.dot(grid_points, W) + b)
h2_grid = np.maximum(0, np.dot(h1_grid, W1) + b1)
scores_grid = np.dot(h2_grid, W2) + b2
pred_grid = np.argmax(scores_grid, axis = 1)
pred_3d = pred_grid.reshape(xx.shape)
accuracy_text = f"Training Accuracy: {accuracy*100:.2f}%"
# クラスごとの色を、matplotlibの 'jet' カラーマップで対応
unique_classes = np.unique(y)
num_classes = len(unique_classes)
# jet カラーマップから [0,1] に正規化した色を取り出す
cmap = cm.get_cmap('jet', num_classes)
class_colors = {
cls: f'rgb({int(r*255)}, {int(g*255)}, {int(b*255)})'
for cls, (r, g, b, _) in zip(unique_classes, cmap(np.linspace(0, 1, num_classes)))
}
# 描画用フィギュア
fig_combined = go.Figure()
# クラスごとの領域描画(Isosurface)
for cls in unique_classes:
mask = (pred_3d == cls).astype(np.int8)
fig_combined.add_trace(go.Isosurface(
x = xx.ravel(), y = yy.ravel(), z = zz.ravel(),
value = mask.ravel(),
isomin = 0.5, isomax = 1.0,
opacity = 0.2,
surface_count = 1,
colorscale = [[0, class_colors[cls]], [1, class_colors[cls]]],
showscale = False,
name = f"Class {cls}"
))
# 元の学習データ(正解ラベルに基づいて色付け)
fig_combined.add_trace(go.Scatter3d(
x = X[:, 0], y = X[:, 1], z = X[:, 2],
mode = 'markers',
marker = dict(
size = 5,
color = y,
colorscale = 'Jet',
line = dict(width = 0.5, color = 'black'),
opacity = 1.0,
showscale = False
),
name = 'Training Data'
))
# レイアウト調整
fig_combined.update_layout(
title = '3D Region Coloring with Class-Matched Colors',
scene = dict(
xaxis = dict(range = [-1.2, 1.2]),
yaxis = dict(range = [-1.2, 1.2]),
zaxis = dict(range = [-0.5, 2.5])
)
)
# 書き出し
fig_combined.write_html("spiral_region_classification_more_ramdomly.html")
作成したデータセット及び領域分類も重ねて表示した結果が以下
https://ecalj.sakura.ne.jp/data/educational/spiral_dataset_more_ramdomly.html
https://ecalj.sakura.ne.jp/data/educational/spiral_region_classification_more_ramdomly.html
これも分類がうまくできている。
まとめ
本記事では、ニューラルネットワークを用いた3次元空間の分類問題について、以下のステップを通して紹介した。
-
2次元の渦巻きデータ(spiral)の生成と分類(引用)
-
その拡張として3次元スパイラルデータの生成
-
ニューラルネットワークによる分類と学習の実装
-
Plotlyを用いた3D空間上での可視化(分類結果の重ね描き)
全体コード
import numpy as np
import matplotlib.pyplot as plt
import plotly.express as px
import plotly.graph_objects as go
from matplotlib import cm
# Generate sample data 乱数のシード
np.random.seed(0) # 再現性を確保するために乱数のシードを固定
# パラメータの設定
N = 30 # クラスごとのデータ点数
D = 3 # データの次元数
K = 3 # クラス数
R = 0.5 # 基本半径
R_NOISE = 0.2 # 半径に加えるノイズの強さ
# データとラベルを格納する配列を初期化
X = np.zeros((N*K, D)) # データ点の座標を格納する配列
y = np.zeros((N*K), dtype='int') # クラスラベルを格納する配列
# 各クラスのデータを生成
for j in range(K):
ix = range(N*j, N*(j+1)) # クラス j に対応するインデックス範囲
r = R + np.random.randn(N) * R_NOISE # 半径にノイズを加える
t = np.linspace(j*2*np.pi/K, j*2*np.pi/K + 2.5*np.pi, N) + j*8*np.pi/K # 角度(スパイラルの形状を作る)
z = np.linspace(0.0, 2, N) # z軸方向の値(線形に増加)
# データ点を生成(スパイラル状に配置)
X[ix] = np.c_[r*np.sin(t), r*np.cos(t), z]
y[ix] = j # クラスラベルを設定
# 3D散布図を作成
fig = px.scatter_3d(
x=X[:, 0], # x座標
y=X[:, 1], # y座標
z=X[:, 2], # z座標
title="3D Spiral Dataset", # グラフのタイトル
labels={'x': 'X', 'y': 'Y', 'z': 'Z'}, # 軸ラベル
)
# グラフのレイアウトを調整
fig.update_layout(
scene=dict(
xaxis=dict(range=[-1.2, 1.2], nticks=5), # x軸の範囲と目盛り数
yaxis=dict(range=[-1.2, 1.2], nticks=5), # y軸の範囲と目盛り数
zaxis=dict(range=[-0.5, 2.5], nticks=5), # z軸の範囲と目盛り数
)
)
# データ点のプロット設定
fig.update_traces(
marker=dict(
size=5, # マーカーサイズ
color=y, # クラスラベルに基づく色付け
colorscale='jet'
)
)
# グラフをHTMLファイルとして保存
fig.write_html('spiral_dataset_3d.html') # 出力ファイル名
# NN Architecture input, hidden1, hidden2
# input X -->W,b-->ReLU--> hidden layer1 -->W1,b1-->ReLU=> hidden layer2 -->W2,b2---> scores
# initialize parameters randomly
h1 = 100 # size of hidden layer1
h2 = 50 # size of hidden layer2
#initialization
W = 0.01 * np.random.randn(D,h1)
b = np.zeros((1,h1))
W1 = 0.01 * np.random.randn(h1,h2)
b1 = np.zeros((1,h2))
W2 = 0.01 * np.random.randn(h2,K)
b2 = np.zeros((1,K))
print(' W.shape b.shape',W.shape,b.shape)
print('W1.shape b1.shape',W1.shape,b1.shape)
print('W2.shape b2.shape',W2.shape,b2.shape)
# Set some hyperparameters
step_size = 1e-0
reg = 1e-3 # regularization strength
# Gradient descent loop
ndata = X.shape[0]
print('training data',ndata)
for i in range(1000):
hidden_layer1 = np.maximum(0, np.dot(X, W) + b) # X ---> hidden layer1
hidden_layer2 = np.maximum(0, np.dot(hidden_layer1, W1) + b1) # hidden layer1 ---> hidden layer2
# evaluate class scores, [N x K]
scores = np.dot(hidden_layer2, W2) + b2 # hidden layer2 ---> scores (logit)
# compute the class probabilities by softmax function
exp_scores = np.exp(scores)
probs = exp_scores / np.sum(exp_scores, axis=1, keepdims=True) # [N x K]
# compute the loss: average cross-entropy loss and regularization
correct_logprobs = np.array([-np.log(probs[i,y[i]]) for i in range(ndata)]) #i番目のデータの正解確率(対数をとって−1倍する)。
if(i==1):
print('probs.shape=',probs.shape)
print('corect_logprobs.shape=',correct_logprobs.shape)
# loss function
data_loss = np.sum(correct_logprobs)/ndata #正解確率(-logしたもの)の平均値
reg_loss = 0.5*reg*np.sum(W*W) + 0.5*reg*np.sum(W1*W1)+ 0.5*reg*np.sum(W2*W2) #正規化関数(Wなどの値を小さくするようにする)
loss = data_loss + reg_loss
if i % 100 == 0: print('iteration {}: loss {}'.format(i, loss))
# compute the gradient on scores
dscores = probs
for i in range(ndata):
dscores[i,y[i]] -= 1
dscores /= ndata
dW2 = np.dot(hidden_layer2.T, dscores) # backpropate the gradient to the parameters
db2 = np.sum(dscores, axis=0, keepdims=True) # first backprop into parameters W2 and b2
dhidden2 = np.dot(dscores, W2.T)
dhidden2[hidden_layer2 <= 0] = 0 # backprop the ReLU non-linearity
dW1 = np.dot(hidden_layer1.T, dhidden2)
db1 = np.sum(dhidden2, axis=0, keepdims=True)
dhidden1 = np.dot(dhidden2, W1.T)
dhidden1[hidden_layer1 <= 0] = 0
dW = np.dot(X.T, dhidden1)
db = np.sum(dhidden1, axis=0, keepdims=True)
# add regularization gradient contribution
dW2 += reg * W2
dW1 += reg * W1
dW += reg * W
# perform a parameter update
W += -step_size * dW
b += -step_size * db
W1 += -step_size * dW1
b1 += -step_size * db1
W2 += -step_size * dW2
b2 += -step_size * db2
"""学習終了。以下で学習したNNによりすべての点での値を予想する(塗り分けることになる)"""
"""平面上でのすべての点での予想をおこなった。なるほど、うまく予想している。"""
# === 3D空間の塗り分け予測可視化(元データも重ねる) ===
# グリッド範囲と解像度
x_range = np.linspace(-1.2, 1.2, 30)
y_range = np.linspace(-1.2, 1.2, 30)
z_range = np.linspace(-0.5, 2.5, 60)
xx, yy, zz = np.meshgrid(x_range, y_range, z_range)
grid_points = np.c_[xx.ravel(), yy.ravel(), zz.ravel()]
# 各点に対して予測
h1_grid = np.maximum(0, np.dot(grid_points, W) + b)
h2_grid = np.maximum(0, np.dot(h1_grid, W1) + b1)
scores_grid = np.dot(h2_grid, W2) + b2
pred_grid = np.argmax(scores_grid, axis=1)
pred_3d = pred_grid.reshape(xx.shape)
# クラスごとの色を、matplotlibの 'jet' カラーマップで対応
unique_classes = np.unique(y)
num_classes = len(unique_classes)
# jet カラーマップから [0,1] に正規化した色を取り出す
cmap = cm.get_cmap('jet', num_classes)
class_colors = {
cls: f'rgb({int(r*255)}, {int(g*255)}, {int(b*255)})'
for cls, (r, g, b, _) in zip(unique_classes, cmap(np.linspace(0, 1, num_classes)))
}
# 描画用フィギュア
fig_combined = go.Figure()
# クラスごとの領域描画(Isosurface)
for cls in unique_classes:
mask = (pred_3d == cls).astype(np.int8)
fig_combined.add_trace(go.Isosurface(
x=xx.ravel(), y=yy.ravel(), z=zz.ravel(),
value=mask.ravel(),
isomin=0.5, isomax=1.0,
opacity=0.2,
surface_count=1,
colorscale=[[0, class_colors[cls]], [1, class_colors[cls]]],
showscale=False,
name=f"Class {cls}"
))
# 元の学習データ(正解ラベルに基づいて色付け)
fig_combined.add_trace(go.Scatter3d(
x=X[:, 0], y=X[:, 1], z=X[:, 2],
mode='markers',
marker=dict(
size=5,
color=y,
colorscale='Jet',
line=dict(width=0.5, color='black'),
opacity=1.0,
showscale=False
),
name='Training Data'
))
# レイアウト調整
fig_combined.update_layout(
title='3D Region Coloring with Class-Matched Colors',
scene=dict(
xaxis=dict(range=[-1.2, 1.2]),
yaxis=dict(range=[-1.2, 1.2]),
zaxis=dict(range=[-0.5, 2.5])
)
)
# 書き出し
fig_combined.write_html("spiral_net_3d_with_matched_colors.html")
-
Stanford CS231n: https://cs231n.github.io/neural-networks-case-study/ ↩
-
Qiita記事「ニューラルネットのうずまきサンプル」: https://qiita.com/takaokotani/items/ed5b02dc3e7ae75e6002 ↩ ↩2 ↩3