1.はじめに
Python機械学習プログラミング[第2版] という本を買って勉強を始めました。効率的な学習を進める為には、勉強した内容をアウトプットするのが効果的ということで、勉強した内容を備忘録の形でここに残すことを進めています。
なお、本ではコードを一部 jupyter notebook形式で記載しているようですが、ここでは.py形式でも実行できるような形式で記載したいと思います。
今回は、第5章次元削減の中の線形判別分析です。
2.線形判別分析(wineデータ)
前回の主成分分析は、クラス情報は一切参照せずに行う分析で、いわゆる教師なし分析でした。今回は、クラス情報も使った、線形判別分析です。テキストのコードを実行してみましょう。
import pandas as pd
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import StandardScaler
import numpy as np
import matplotlib.pyplot as plt
# データセットの読み込みとサマリー表示
df_wine = pd.read_csv('https://archive.ics.uci.edu/ml/'
'machine-learning-databases/wine/wine.data',
header=None)
df_wine.columns = ['Class label', 'Alcohol', 'Malic acid', 'Ash',
'Alcalinity of ash', 'Magnesium', 'Total phenols',
'Flavanoids', 'Nonflavanoid phenols', 'Proanthocyanins',
'Color intensity', 'Hue',
'OD280/OD315 of diluted wines', 'Proline']
print(df_wine.head())
print()
# データセットのトレーニングデータとテストデータへの分割(7:3)
X, y = df_wine.iloc[:, 1:].values, df_wine.iloc[:, 0].values
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.3,
stratify=y, random_state=0)
print('X_train.shape = ', X_train.shape)
print('y_train.shape = ', y_train.shape)
print()
# Xデータの標準化
sc = StandardScaler()
X_train_std = sc.fit_transform(X_train)
X_test_std = sc.transform(X_test)
# 平均ベクトルの計算
np.set_printoptions(precision=4)
mean_vecs = []
for label in range(1, 4):
mean_vecs.append(np.mean(X_train_std[y_train == label], axis=0))
print('MV %s: %s\n' % (label, mean_vecs[label - 1]))
# クラス内変動の計算
d = 13
S_W = np.zeros((d, d))
for label, mv in zip(range(1, 4), mean_vecs):
class_scatter = np.zeros((d, d))
for row in X_train_std[y_train == label]:
row, mv = row.reshape(d, 1), mv.reshape(d, 1)
class_scatter += (row - mv).dot((row - mv).T)
S_W += class_scatter
print('Within-class scatter matrix: %sx%s' % (S_W.shape[0], S_W.shape[1]))
print('Class label distribution: %s' % np.bincount(y_train)[1:])
# スケーリング
d = 13
S_W = np.zeros((d, d))
for label, mv in zip(range(1, 4), mean_vecs):
class_scatter = np.cov(X_train_std[y_train == label].T)
S_W += class_scatter
print('Scaled within-class scatter matrix: %sx%s' % (S_W.shape[0], S_W.shape[1]))
# クラス間変動の計算
mean_overall = np.mean(X_train_std, axis=0)
d = 13
S_B = np.zeros((d, d))
for i, mean_vec in enumerate(mean_vecs):
n = X_train[y_train == i + 1, :].shape[0]
mean_vec = mean_vec.reshape(d, 1)
mean_overall = mean_overall.reshape(d, 1)
S_B += n * (mean_vec - mean_overall).dot((mean_vec - mean_overall).T)
print('Between-class scatter matrix: %sx%s' % (S_B.shape[0], S_B.shape[1]))
# 固有値を降順に表示
eigen_vals, eigen_vecs = np.linalg.eig(np.linalg.inv(S_W).dot(S_B))
eigen_pairs = [(np.abs(eigen_vals[i]), eigen_vecs[:, i])
for i in range(len(eigen_vals))]
eigen_pairs = sorted(eigen_pairs, key=lambda k: k[0], reverse=True)
print('\nEigenvalues in descending order:\n')
for eigen_val in eigen_pairs:
print(eigen_val[0])
# 固有値の寄与度をパレート図に表示
tot = sum(eigen_vals.real)
discr = [(i / tot) for i in sorted(eigen_vals.real, reverse=True)]
cum_discr = np.cumsum(discr)
plt.bar(range(1, 14), discr, alpha=0.5, align='center',
label='individual "discriminability"')
plt.step(range(1, 14), cum_discr, where='mid',
label='cumulative "discriminability"')
plt.ylabel('"discriminability" ratio')
plt.xlabel('Linear Discriminants')
plt.ylim([-0.1, 1.1])
plt.legend(loc='best')
plt.tight_layout()
plt.show()
# 射影行列wを設定
w = np.hstack((eigen_pairs[0][1][:, np.newaxis].real,
eigen_pairs[1][1][:, np.newaxis].real))
print('Matrix W:\n', w)
# トレインデータを射影行列wで変換
X_train_lda = X_train_std.dot(w)
colors = ['r', 'b', 'g']
markers = ['s', 'x', 'o']
for l, c, m in zip(np.unique(y_train), colors, markers):
plt.scatter(X_train_lda[y_train == l, 0],
X_train_lda[y_train == l, 1] * (-1),
c=c, label=l, marker=m)
plt.xlabel('LD 1')
plt.ylabel('LD 2')
plt.legend(loc='lower right')
plt.tight_layout()
plt.show()

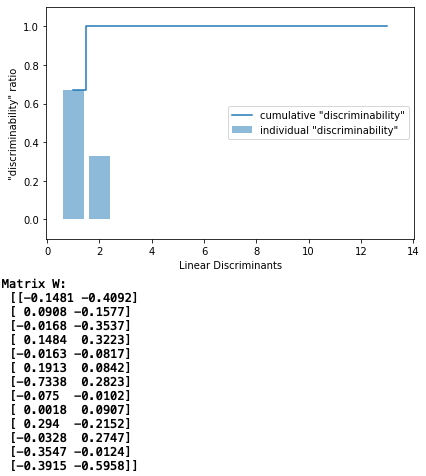
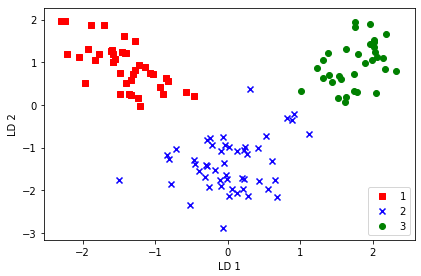
さすがに、クラス情報も加味した分析なので、主成分分析より明らかに線形分離性が高い様です。主成分の上位2つの寄与率は、ほぼ100%です。
3.線形判別分析(wineデータ by sklearnライブラリー)
当然、線形判別分析も、sklearnのライブラリーにありますので、wineデータの決定領域表示をしてみます。
from matplotlib.colors import ListedColormap
from sklearn.discriminant_analysis import LinearDiscriminantAnalysis as LDA
from sklearn.linear_model import LogisticRegression
# 決定領域表示関数
def plot_decision_regions(X, y, classifier, resolution=0.02):
# setup marker generator and color map
markers = ('s', 'x', 'o', '^', 'v')
colors = ('red', 'blue', 'lightgreen', 'gray', 'cyan')
cmap = ListedColormap(colors[:len(np.unique(y))])
# plot the decision surface
x1_min, x1_max = X[:, 0].min() - 1, X[:, 0].max() + 1
x2_min, x2_max = X[:, 1].min() - 1, X[:, 1].max() + 1
xx1, xx2 = np.meshgrid(np.arange(x1_min, x1_max, resolution),
np.arange(x2_min, x2_max, resolution))
Z = classifier.predict(np.array([xx1.ravel(), xx2.ravel()]).T)
Z = Z.reshape(xx1.shape)
plt.contourf(xx1, xx2, Z, alpha=0.4, cmap=cmap)
plt.xlim(xx1.min(), xx1.max())
plt.ylim(xx2.min(), xx2.max())
# plot class samples
for idx, cl in enumerate(np.unique(y)):
plt.scatter(x=X[y == cl, 0],
y=X[y == cl, 1],
alpha=0.6,
c=cmap(idx),
edgecolor='black',
marker=markers[idx],
label=cl)
# sklearnによる線形判別分析
lda = LDA(n_components=2)
X_train_lda = lda.fit_transform(X_train_std, y_train)
X_test_lda = lda.transform(X_test_std)
lr = LogisticRegression(solver='lbfgs', multi_class='auto') # 引数追加
lr = lr.fit(X_train_lda, y_train)
# トレインデータによる決定領域表示
plot_decision_regions(X_train_lda, y_train, classifier=lr)
plt.title('LDA for train_data')
plt.xlabel('LD 1')
plt.ylabel('LD 2')
plt.legend(loc='lower left')
plt.tight_layout()
plt.show()
# テストデータによる決定領域表示
plot_decision_regions(X_test_lda, y_test, classifier=lr)
plt.title('LDA for test_data')
plt.xlabel('LD 1')
plt.ylabel('LD 2')
plt.legend(loc='lower left')
plt.tight_layout()
plt.show()
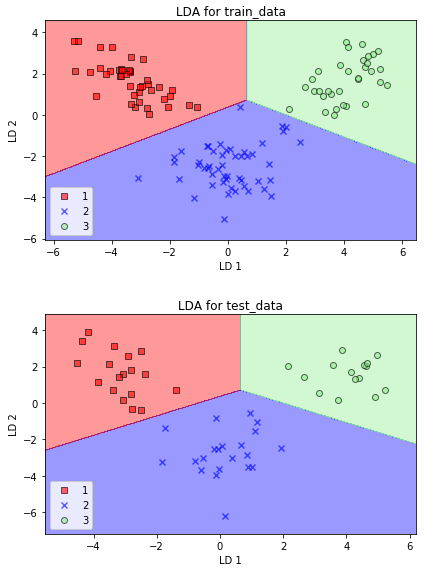
私のsklearnのバージョン(0.21.3)の関係で、ワーニングが出ましたので、lr = LogisticRegression()の引数を追加しています。