skimage.feature.hog (http://scikit-image.org/docs/dev/api/skimage.feature.html#hog) は入力画像からHOG特徴量を抽出してくれるのだが,出力結果は1d-arrayであり,何がなんだかわからないという問題がある.https://github.com/holtzhau/scikits.image/blob/master/skimage/feature/hog.py にあるコードを読むと,このarrayをどう解釈すれば良いかがわかる.
入力引数で重要なもの
-
orientations: ヒストグラムのビン数(default=9) -
pixels_per_cell: セルのサイズ(default=(8,8)) -
cells_per_block: ブロックごとのセル数(default=(3, 3))
出力を見てみる
180行目
hog.py
return normalised_blocks.ravel()
ここravelせずに返して欲しかった.normalised_blocksの宣言を見てみると,
160行目
hog.py
n_blocksx = (n_cellsx - bx) + 1
n_blocksy = (n_cellsy - by) + 1
normalised_blocks = np.zeros((n_blocksx, n_blocksy, bx, by, orientations))
ここで使われている変数は100行目あたりで宣言されている
hog.py
sx, sy = image.shape
cx, cy = pixels_per_cell
bx, by = cells_per_block
n_cellsx = int(np.floor(sx // cx)) # number of cells in x
n_cellsy = int(np.floor(sy // cy)) # number of cells in y
というわけで,hogを回して得られる1-d array(retvalとする)は,
retval.reshape((n_blocksx, n_blocksy, bx, by, orientations))
としてあげると,元の形に戻すことができる.
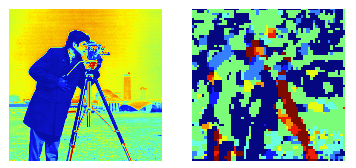
活用例
たとえば,blockごとの最大勾配方向を可視化することができる.
visualize_argmaxhog.py
from skimage.feature import hog
from skimage.data import camera
import matplotlib.pyplot as plt
img = camera()
orientations = 9
pixels_per_cell = (8, 8)
cells_per_block = (3, 3)
h = hog(img, orientations=orientations, pixels_per_cell=pixels_per_cell, cells_per_block=cells_per_block)
sx, sy = img.shape[:2]
cx, cy = (8, 8)
bx, by = (3, 3)
n_cellsx = int(np.floor(sx // cx)) # number of cells in x
n_cellsy = int(np.floor(sy // cy)) # number of cells in y
n_blocksx = (n_cellsx - bx) + 1
n_blocksy = (n_cellsy - by) + 1
himg = h.reshape((n_blocksx * n_blocksy, bx, by, orientations))
vis = np.array([np.argmax(x.sum(axis=(0, 1))) for x in himg]).reshape((n_blocksx, n_blocksy))
plt.subplot(1, 2, 1)
plt.imshow(img)
plt.axis('off')
plt.subplot(1, 2, 2)
plt.imshow(vis, interpolation='nearest')
plt.axis('off')