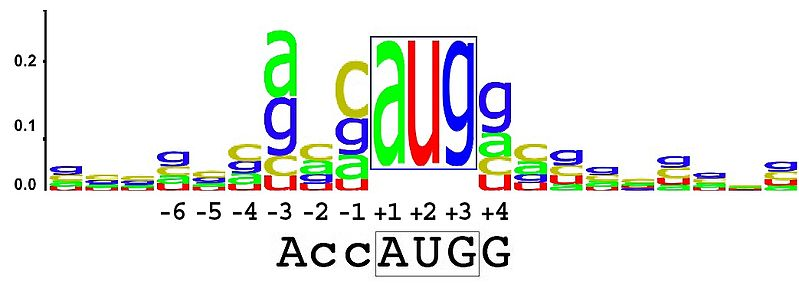Wordleの不満点
引き続き、Wordleにハマっています。
しかしWordle、面白いのは確かなんですが、不満な点もあります。
それは、最後に答えの英単語が明らかにされても、「し…知らね~~」となることがあまりに多いことです。
なんだよ、KNOLLとかWRUNGって。
確かに前回の記事では語彙力をつける勉強になると言いましたが、いくらなんでも勉強になる率が高すぎます。
最初の数手でヒントを貰うところまでは良いんです。
しかし、ヒントを貰った後から、自分の語彙の中でヒントに当てはまる単語がないので、語感だけで「なんかこういう単語あるんじゃない?」って思いながら適当に文字を埋めることになりがちです。しかもそれで当たったりする。
考えてみれば、我々日本人にとって英語は母語ではないので、その点では最初から不利な勝負に挑まされているわけです。
そこで、敵(Wordle)との戦力差を埋めるためのチートシートを作ってみました。
ネタバレ注意
次の画像には、任意のWordleの答えが含まれています。

たとえば、CHEATとSHEETという単語は以下の場所にあります。

こんな感じで、Wordleのお題となりうる単語はすべて先の画像のどこかに隠されています。
これで語彙力の差は、幾分か埋められると思います。
ただ、このチートシートを使うと、Wordle本来の推理ゲームという要素が薄れ、代わりにウォーリーをさがせ!の要素がゲーム性に混じってきます。
具体的に何をやったの?
Wordleの解答候補となる2317個の英単語を、共通パターンを持つ単語のクラスタ100個に分け、それぞれを配列ロゴで可視化しました。
パターンとは
冒頭で述べた、
語感だけで「なんかこういう単語あるんじゃない?」って思いながら適当に文字を埋める
という行為について、我々の脳が実際に何をやっているか考えてみると、既知の英単語において頻出するパターンから演繹して、「こんな単語もあるんじゃないか?」と予想しているのではないかと思います。
実際、言語的にいえば、英語に限らず単語には、接辞と呼ばれるパターンが存在します。たとえば、
-
接頭辞「UN***」
-
UNSET(置く= SETの反対、外す)
-
UNTIE (結ぶ = TIEの反対、ほどく)
-
接尾辞「***ER」
-
EATER(食べる = EATする人)
-
DIVER(潜る = DIVEする人)
のようなものです。
接辞はこれらの他にも真ん中に来るもの(接中辞)、頭と尻の両方に来るもの(接周辞)など、様々です。
しかし、Wordleは五文字の単語という制約があるので、実際に使われている接辞の数は限られているでしょう。
また、接辞でないにしても、子音の後に母音が来やすいなど、隠されたパターンもありそうです。
そこで、五文字の単語に潜むパターンを、事前知識に頼ることなく、機械的に抽出することにしました。
クラスタとは
今回私が取った手法は、ある程度似ている単語でクラスタにわけて、そのクラスタの共通部分を取り出す、というものです。
そのためには、「似ている」ということを単語の類似度あるいは距離でちゃんと定義してやる必要があります。
文字列に対する距離というと、レーベンシュタイン距離(編集距離、同じ文字列にするための必要な作業の数)が有名です。
レーベンシュタイン距離は異なる長さの文字列に対しても定義できますが、今回は文字列の長さが5で固定という状況なので、もっと簡単なアプローチを取ってみました。
(おそらく、レーベンシュタイン距離を使っても似たような結果になるとは思いますが)
まず、各単語の位置×アルファベットを特徴量として、ワンホットエンコーディングします。
以下では前回に引き続き、df_charというデータフレームに適用します。
df_char_onehot = pd.get_dummies(df_char)
df_char_onehot
あとはこの行列に、従来のクラスタリング手法を適用するだけです。
今回は**階層型クラスタリング(ユークリッド距離 + Ward法)**を選びました。
コードと結果のデンドログラム(樹形図)は以下です。
from scipy.spatial import distance
from scipy.cluster import hierarchy
row_linkage = hierarchy.linkage(df_char_onehot,method='ward')
sns.clustermap(df_char_onehot, row_linkage=row_linkage, col_cluster=False)

なんとなく、行方向に似ている単語同士がまとまってくれている気がします。
しかしこのままでは、各クラスタがどういう特徴を持っているのかがわかりにくいですね。
配列ロゴとは
クラスタの特徴を視覚化するため、バイオインフォマティクスで使われる、配列ロゴというツールを借用しました。
配列ロゴは、進化的に保存されているDNA/RNA/アミノ酸などの配列を視覚化するときに、しばしば使われる手法です。
たとえば上記の配列ロゴは、Wikipediaから引用したものですが、ヒトの全mRNAの開始コドン周辺で最も保存された塩基を示す配列ロゴです。
配列ロゴは、位置ごとに異なる文字の積み重ねで構成されています。各位置において、文字の高さの合計は「その位置における保存度合い」、文字の相対的な大きさは「その位置で観察される頻度」を表しています。
pythonでは、Logomakerというライブラリで配列ロゴを作成できます。
階層型クラスタリングで得られたすべてのクラスタに対して、配列ロゴを作成しましょう。
**配列ロゴ作成コード**
import logomaker
# Scipy treeのノードとクラスタIDを対応させる関数
def Node2ID(node):
dictID2Node = dict()
dictID2Node[node.id] = node
if not node.is_leaf():
dictID2Node.update(Node2ID(node.get_left()))
dictID2Node.update(Node2ID(node.get_right()))
return dictID2Node
# ノードに対してすべての子孫を得る関数
def getDescendants(node):
Descendants = []
if node.is_leaf():
Descendants.append(node.id)
return Descendants
else:
Descendants = getDescendants(node.get_left())
Descendants.extend(getDescendants(node.get_right()))
return Descendants
# ひとつのクラスタを画像出力する関数
def show_single_cluster(cluster_member_list, output_file):
color_scheme = {key:plt.cm.get_cmap('hsv')(i/26) for i,key in enumerate(list("ABCDEFGHIJKLMNOPQRSTUVWXYZ"))}
df_cluster_string = df_char_onehot.iloc[cluster_member_list]
df_cluster_char = pd.DataFrame([list(index) for index in df_cluster_string.index])
df_count = pd.DataFrame(0,index=range(5),columns=list("ABCDEFGHIJKLMNOPQRSTUVWXYZ"))
for pos in range(5):
dict_char_freq = df_cluster_char[pos].value_counts().to_dict()
for key in dict_char_freq.keys():
df_count.loc[pos,key] = dict_char_freq[key]
fig, ax = plt.subplots(1, 1, figsize=(1, 0.5))
logomaker.Logo(df_count,color_scheme=color_scheme,show_spines=False,ax=ax);
ax.set_xticks([])
ax.set_yticks([])
fig.savefig(output_file)
return
# クラスタのIDから画像を出力する関数
def show_by_id(ID,output_file):
return show_single_cluster(getDescendants(dictID2Node[ID]),output_file)
# すべての画像を出力しておく
scipy_tree = hierarchy.to_tree(row_linkage,False)
dictID2Node = Node2ID(scipy_tree)
output_dir="fig"
if not os.path.isdir(output_dir):
os.mkdir(output_dir)
for ID in dictID2Node.keys():
output_file = os.path.join(output_dir, "%d.png" % ID)
show_by_id(ID,output_file);
出力フォルダがこんな感じになるはずです。
木の比較的深い部分を表示していますが、似ている単語が同じクラスタに入っているということが一目瞭然ですね。
次に、Tree構造をグラフィカルに可視化するライブラリであるete3を使って、デンドログラムに配列ロゴを加えましょう。
from ete3 import Tree, TreeStyle, TextFace, ImgFace, faces, AttrFace
# scipyのtreeからNewick木をつくる関数
def getNewick(node, newick, parentdist, depth=0, max_depth=3, max_size=100):
n_children = len(getDescendants(node))
if node.is_leaf()|(depth>=max_depth)|(n_children<max_size):
return "%s:%.2f%s" % ("#" + str(node.id) + " \n[" + str(len(getDescendants(node)))+ "words]"
, parentdist - node.dist, newick)
else:
if len(newick) > 1:
newick = ")%s:%.2f%s" % ("#" + str(node.id) + " \n[" + str(n_children)+ "words]"
, parentdist - node.dist, newick)
else:
newick = ");"
newick = getNewick(node.get_left(), newick, node.dist, depth=depth+1, max_depth=max_depth, max_size=max_size)
newick = getNewick(node.get_right(), ",%s" % (newick), node.dist, depth=depth+1, max_depth=max_depth, max_size=max_size)
newick = "(%s" % (newick)
return newick
Newick = getNewick(scipy_tree, "", scipy_tree.dist, max_depth=20,max_size=35).replace(";","#4628 [2315words];")
ete3_tree = Tree(Newick, format=1)
ete3_tree.render("%%inline", tree_style = ts)
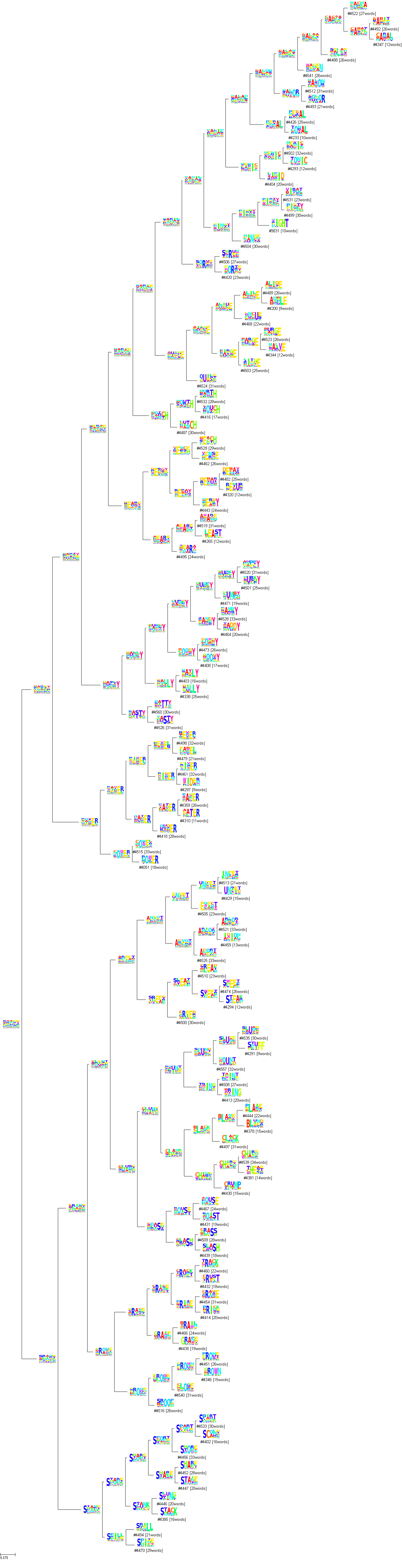
ete3を使った結果は以下です。末端のクラスタに、そのクラスタに含まれる単語の数を記しておきました。
実際の葉(末端ノード)まで表示すると大変な大きさのデンドログラムになってしまうので、今回はクラスタの大きさが35を切った時点で、それ以下の子ノードを表示しないようにしました。これでちょうど、100個のクラスタが得られます。
(冒頭に出したチートシートは、この100個のクラスタの配列ロゴを上下左右10*10に並べただけのものです)
こうして見ると、Sで始まる単語、Yで終わる単語の多さに気づかされますね。
また、-LLY、-TTY、-INT、-IGHTなどの、親しみのあるパターンもちゃんと抽出できていることがわかります。
冒頭のチートシートを使わないにしても、こうしたパターンがあることだけでも頭に入れておくと、Wordleでいざというときに役立つかもしれません(LとYが黄色になったら、-LY形かもと推測したり)。
パターンだけを抽出したい場合は
階層型クラスタリングの欠点は、「1つの単語が2つのクラスタに所属する」ことを許さない点です。
例えば仮に、「ST-」パターンのクラスタと、「-ING」パターンのクラスタがあるとしましょう。
このとき、「STING」という単語は両方のパターンを持っているわけですが、階層型クラスタリングでは必ずどちらかの一方のクラスタだけに所属しなければなりません。
結果的には、2つのパターンのどちらかの構成員が実際よりも一人減って見えることになってしまいます。
こうした問題は、行列分解系の手法を使うと解決できるでしょう。
例えば、「STING」という単語は、「ST-」パターンと「-ING」パターンを0.5ずつ持つ、というように分解されるはずです。
そこで試しに、非負値行列分解を適用してみました。
from sklearn.decomposition import NMF
model = NMF(n_components=15, init="nndsvd",alpha_W=0.0)
W = pd.DataFrame(model.fit_transform(df_char_onehot), index=df_char_onehot.index)
H = pd.DataFrame(model.components_, columns=df_char_onehot.columns)
color_scheme = {key:plt.cm.get_cmap('hsv')(i/26) for i,key in enumerate(list("ABCDEFGHIJKLMNOPQRSTUVWXYZ"))}
ncol = 5
nrow = np.int64(np.ceil(len(H.index) * 1.0 / ncol))
fig, axes = plt.subplots(nrow, ncol, figsize=(ncol * 5, nrow*5))
axes = axes.flatten()
for i,index in enumerate(H.index):
df_count = pd.DataFrame(0,index=range(1,6),columns=list("ABCDEFGHIJKLMNOPQRSTUVWXYZ"))
for column in H.columns:
m = re.match(r'([0-9])_([A-Z])', column)
df_count.loc[int(m[1]),m[2]] = H.loc[index, column]
logomaker.Logo(df_count,color_scheme=color_scheme,show_spines=True,ax=axes[i])
axes[i].set_xticks([])
axes[i].set_yticks([])
うまくパターンの部分だけが抽出されていることがわかります。
(ただ、NMF内部のmethodやパラメータを変えたら、共通するパターンもありつつ、違うパターンも結構出てきます。特に正解とかはないので、どういうパラメータを使うべきかが悩ましいところだと感じました。)
以上。