はじめに
仕事で統計遺伝学が必要になりそうなので、GWAS結果からマンハッタンプロットを描いてみた。
備忘録的なやつ。
qqmanパッケージを使います。
ライブラリの読み込み
qqmanパッケージの読み込み
library(qqman)
データの読み込みと確認
使用するデータはqqmanパッケージの内部データの
gwasResults
で。
DF <- gwasResults
str(DF)
table(DF$CHR)
実行結果
> DF <- gwasResults
> str(DF)
'data.frame': 16470 obs. of 4 variables:
$ SNP: chr "rs1" "rs2" "rs3" "rs4" ...
$ CHR: int 1 1 1 1 1 1 1 1 1 1 ...
$ BP : int 1 2 3 4 5 6 7 8 9 10 ...
$ P : num 0.915 0.937 0.286 0.83 0.642 ...
> table(DF$CHR)
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22
1500 1191 1040 945 877 825 784 750 721 696 674 655 638 622 608 595 583 572 562 553 544 535
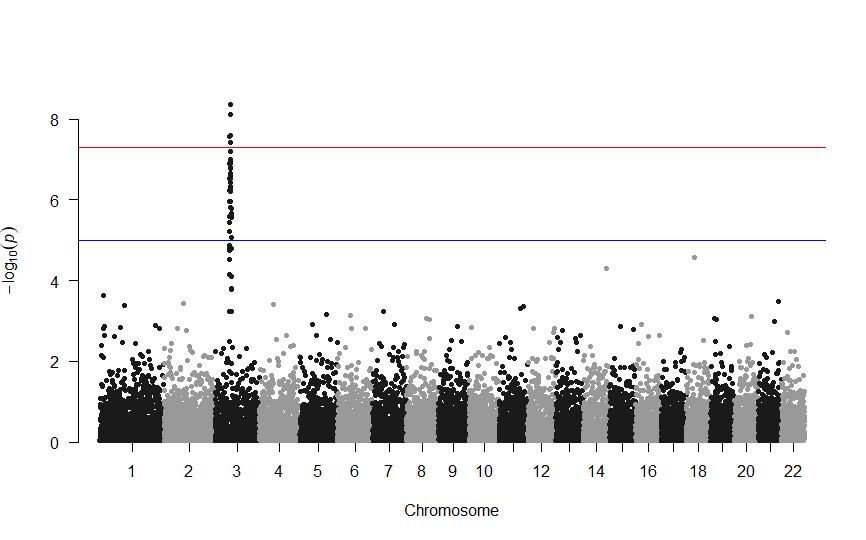
とりあえずマンハッタンプロット
とりあえず描いてみる。
描画は、**manhattan()**関数で。
manhattan(DF)
青色のラインはp値が1.0×10-5のレベルであり、suggestive level(有意ではないが、なんらかの関連がありそうなレベル)を示している。
赤色のラインはp値が5.0×10-8のレベルであり、GWASでな有意レベルを示している。
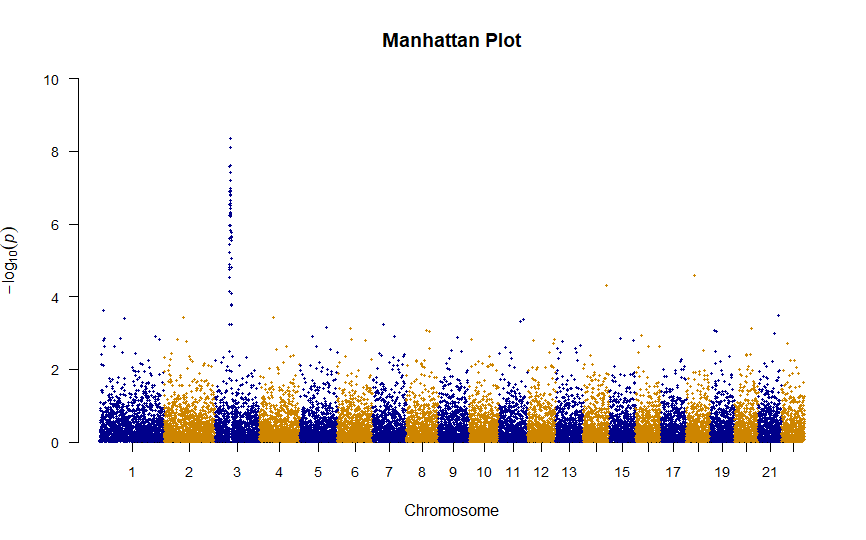
マンハッタンプロットを色々いじってみた①
色をカラフル、プロットのサイズ、などなど条件をいじってみた。
manhattan(DF,
main = "Manhattan Plot", #タイトルを付ける
ylim = c(0, 10), #y軸の範囲
cex = 0.6, #プロットの大きさ
cex.axis = 0.9, #軸のフォントサイズ
col = c("blue4", "orange3"), #プロットの色
suggestiveline = F, #suggestivelineを消す
genomewideline = F) #genomewidelineを消す
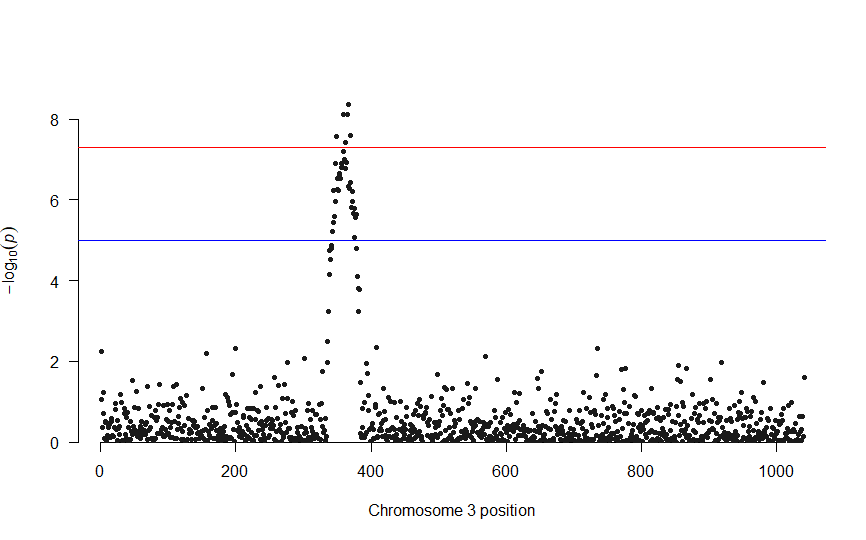
マンハッタンプロットを色々いじってみた②
染色体一本にフォーカスする方法。
3番染色体にフォーカス。
manhattan(subset(DF, CHR == 3))
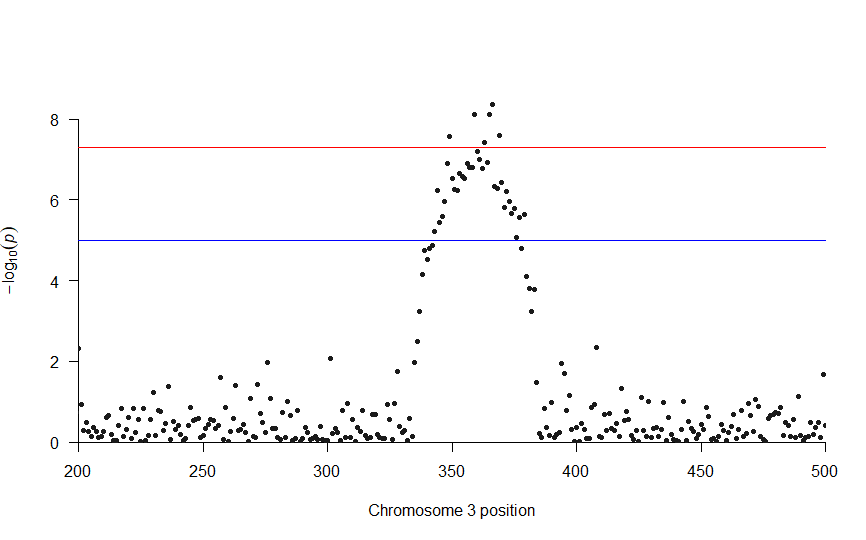
3番染色体をさらにフォーカス
manhattan(subset(DF, CHR == 3),
xlim = c(200, 500))
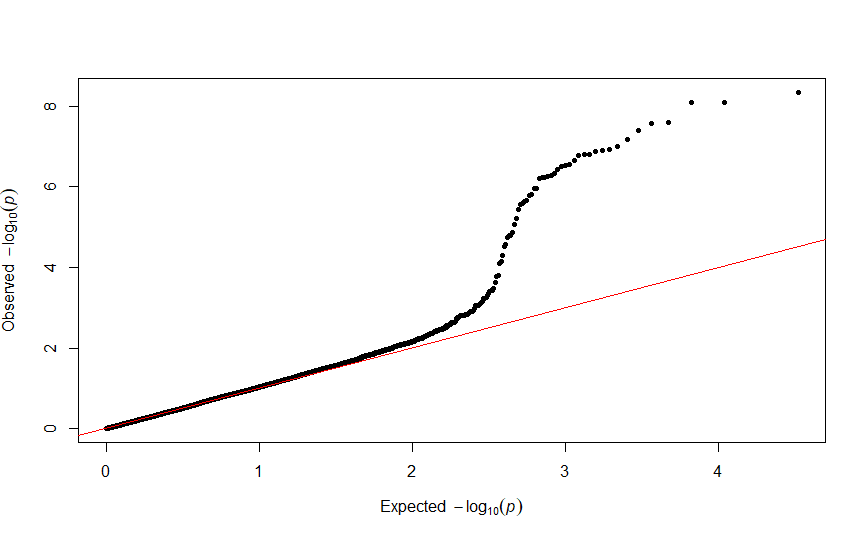
QQプロットも描いてみた
qqmanパッケージの**qq()**関数でQQプロットが書けちゃいます。
データフレームのp値を指定。
qq(DF$P)
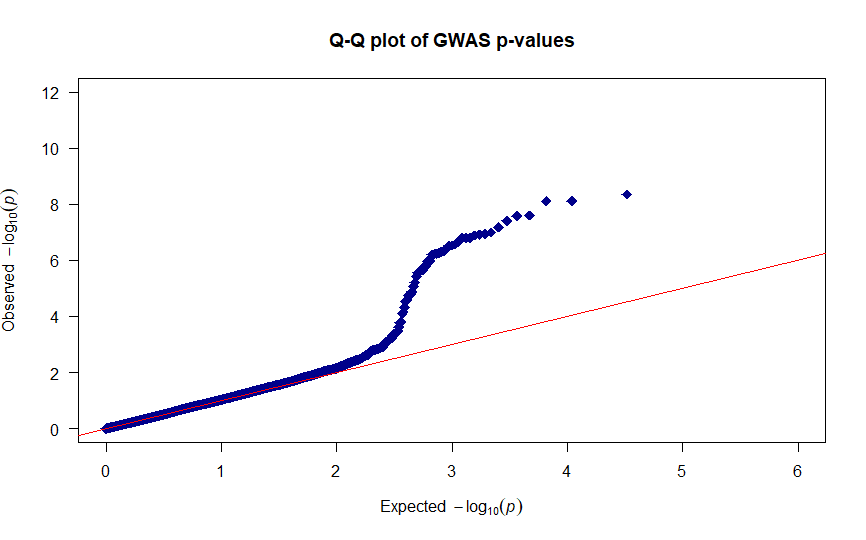
QQプロットも色々いじります。
# QQプロット改
qq(DF$P,
main = "Q-Q plot of GWAS p-values", #タイトルを付ける
xlim = c(0, 6), #x軸の範囲
ylim = c(0, 12), #y軸の範囲
col = "blue4", #プロットの色
cex = 1.5, #プロットの大きさ
las = 1, #y軸のメモリを縦にする
pch = 18) #プロットの種類
最終スクリプト
# Rをきれいにする
rm(list = ls())
# ライブラリの読み込み
library(qqman)
# データの読み込みと確認
DF <- gwasResults
str(DF)
table(DF$CHR)
# マンハッタンプロット
manhattan(DF)
# マンハッタンプロット改
manhattan(DF,
main = "Manhattan Plot", #タイトルを付ける
ylim = c(0, 10), #y軸の範囲
cex = 0.6, #プロットの大きさ
cex.axis = 0.9, #軸のフォントサイズ
col = c("blue4", "orange3"), #プロットの色
suggestiveline = F, #suggestivelineを消す
genomewideline = F) #genomewidelineを消す
# 染色体3のマンハッタンプロット
manhattan(subset(DF, CHR == 3))
# 染色体3のマンハッタンプロット改
manhattan(subset(DF, CHR == 3),
xlim = c(200, 500))
# QQプロット
qq(DF$P)
# QQプロット改
qq(DF$P,
main = "Q-Q plot of GWAS p-values", #タイトルを付ける
xlim = c(0, 6), #x軸の範囲
ylim = c(0, 12), #y軸の範囲
col = "blue4", #プロットの色
cex = 1.5, #プロットの大きさ
las = 1, #y軸のメモリを縦にする
pch = 18) #プロットの種類