はじめに
Ristでは、今年から技術ブログを立ち上げました。
記念すべき第1回目の記事として、2019年9月~2019年11月にKaggleで開催された「RSNA Intracranial Hemorrhage Detection」というコンペの上位解法について紹介させてもらいます。
もし、この大会が開催されたオンラインコンペサイトであるKaggleについてご存じない方は、下記の記事でとてもわかりやすく説明されていますので、一度ご覧いただければと思います。
「結局、Kagglerは何を必死にやっているのか?」というLTをしました
Update 2020年1月21日
「CT画像」を誤って「MRI画像」と書いていたのを訂正しました。
「脳出血」という表現も正確ではなかったので訂正しました。正しくは「頭蓋内出血」です。
ご指摘いただいた方、本当にありがとうございました。
コンペの概要
さっそく今回の紹介するコンペについて説明していきたいと思います。
主催者である北米放射線学会、The Radiological Society of North America (RSNA)は、100年以上の歴史と5400人以上の会員が所属する学会で、毎年世界最大規模の年次医療会議を開催しています。RSNAは2017年、2018年に続き3年連続でKaggleでコンペティションを開催しました。
今回は、脳CTの連続スライスの画像から頭蓋内出血があるかないか(any)、さらに下図のように頭蓋内出血のタイプを5種類(Intraparenchymal、 Intraventricular、 Subarachnoid、 Subdural、 Epidural)の中から予測するといった課題でした。評価指標はloglossで、ラベル「any」に対しては大きめのWeightがかけられていました。
 (図はコンペの[Overview](https://www.kaggle.com/c/rsna-intracranial-hemorrhage-detection/overview/hemorrhage-types)より引用)
(図はコンペの[Overview](https://www.kaggle.com/c/rsna-intracranial-hemorrhage-detection/overview/hemorrhage-types)より引用)
画像はDICOM形式で与えられており画像の情報だけでなく患者IDや脳切片の位置などの情報も利用できました(注釈:最初はピクセルデータのみ使用可能で、メタデータの利用は禁止されていたが途中から使えるようになった)。余談ですが、DICOMとはDigital Imaging and COmmunication in Medicineの略で、「『医用画像情報』ならびに『医用通信プロトコル』に関する国際的な標準規格」のことです。ですので、CT画像以外でも使用されております。本コンペの数ヶ月前に行われたSIIM-ACR Pneumothorax Segmentationという肺のX線画像を扱ったKaggleのコンペでも、同じようにDICOM形式が使われていました。
今回のコンペのポイントは以下の2つだと思います。
- CT画像の扱い方
- Meta情報の使い方
1.CT画像の扱い方
通常の画像は、1 pixelにつき0~255の値をRGBの3チャンネル分もっています。しかし、CTの画像は少し特殊で濃淡情報のみを持っています。その値を「CT値」と呼び、空気が-1000で水が0の相対値で表されます。詳細については、以下のサイトを参考にしてください。
CT値とは?_CT適塾
CT値は範囲を絞ることで特定の組織の画像を得ることができます。例えば、脳の場合は、CT値の中心が40で幅が80の領域、つまり0~80のCT値を取ってくることで脳組織の画像が得られます。これを「〜のWindowで切り取ってくる」と言います。以下のサイトや、Kernelをご覧いただけるとわかりやすいかと思います。
- Windowing (CT), Julia Kube and Andrew Murphy et al.
- Kernel: See like a Radiologist with Systematic Windowing
特定の組織がよく見えるWindowを3つ使い、切り出してきた画像を3チャンネルに1つづつ割り当てて、入力画像を作成して学習させているチームが多かったように思います。ここに関してはコンペの途中でAppianさんがとても素晴らしいDiscussionを投稿してくださり、多くのチームがこれを参考にWindowを取り入れていたので、上位チームの差を分けたわけではないと思います。
2.Meta情報の扱い方
先ほど述べた通り、DICOM形式にはCT値以外にも20以上の情報が含まれていました。以下のKernelを見ていただければ、どういう情報があるかわかります。
そして、今回のコンペで特に大事だったのはCT画像の位置情報です。Meta情報を使えば患者ごとに画像を集めてそれを順番に並び替えることができます。こちらのDiscussionの映像が非常にわかりやすいです。ご覧いただけるとわかる通り、連続する画像はとても似ています。
実際に、ある患者のデータに対してラベルを位置情報を元に並び替えると、その前後の画像にも同じタイプの頭蓋内脳出血が並んでいるのがわかります。

解法について
それでは、上位のチームで使用されていた手法を簡単に紹介していきます。
以下、画像は全て上位チームのDiscussionから引用しています。
3位 takuokoさん

3位の方の解法はこちらから見れます。Githubはこちらです。
takuokoさんの解法のポイントは3つあると思います。1つ目が様々な種類の入力画像を使っているところ、2つ目がマルチタスク学習しているところ、最後3つ目がUser Stackingという手法です。
1つ目の入力画像について説明します。まず患者ごとにCT画像(以下、スライス)をポジション順に並べます。そして、1つ前のポジションのスライスと1つ後のポジションのスライスを結合して1つの入力とし真ん中のスライスのラベルを予測しています。この発想自体は他のチームでも使用されていたのですが、takuokoさんの解法の興味深いところは、**前後1枚のスライスだけではなくて、さらに多くのスライスを組み込んでいたところです。**1つ前と2つ前のスライス、1つ後と2つ後のスライス、2つ前と2つ後のスライス、1〜3つ前のスライスの平均と1〜3つ後のスライスの平均、1〜5つ前後のスライスの前後それぞれの平均、前後にある全てのスライスの前後それぞれの平均、このような様々な入力を用いてモデルを学習されていました。
2つ目のポイントは、ターゲットに関する工夫です。先ほど説明した様々な入力に対して、基本的には他のチーム同様、予測するラベルは真ん中のスライスのラベルだけでした。しかし、ここで予測するラベルを真ん中だけでなく、入力に用いた前後のスライスのラベルも同時に学習することで、シングルモデルでのベストのスコアとなり、アンサンブルすることで1st stage Public 2位まできたそうです。
最後に上記の方法で予測ラベルを出した後、結果をアンサンブルするところでも他のチームにはない方法を用いていました。User Stackingと名付けられたその手法は、各スライスの予測ラベルに加えて前後の20枚のスライスの予測ラベルを特徴量として加えたテーブルを作成し、勾配ブーストを使いラベルの予測を行なっています。それを最終ラベルの予測のアンサンブルに加えていました。
全体を通して、シンプルだけど他のチームにはない発想でとても勉強になりました。
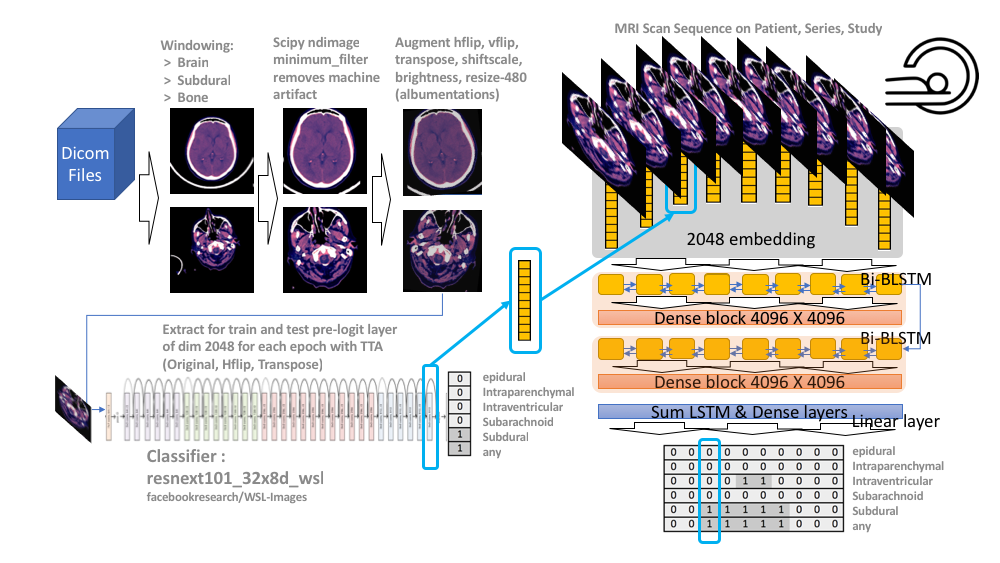
2位 NoBrainerチーム
2位のチームの解法はこちらから見れます。Githubはこちらです。
2位のチームは入力ではなくて出力側での工夫がありました。まずResNeXt101で学習を行います。その後、予測ラベルではなくCNNの直後のGlobal Average Pooling層のembeddingをデータごとにとってきて、ポジションの順番に並べてLSTMのモデルで学習しています。さらにこのembeddingにも一工夫あり、自分のポジションの前後のembeddingの差分も特徴量として加えているのが印象的でした。
そして、LSTMのモデルでは、
- LSTM1回
- LSTM1回+全結合
- LSTM2回
- LSTM2回+全結合
この4つのベクトルを作成した後、これらを全て足し合わせて、それをもう一度全結合層に通してラベルを予測していました。1st stageのPublic Leaderboardで銀メダル圏のあたりからLSTMの工夫をこらすことでPublic 5位 / 2nd stage 2位まで順位を上げています。Githubにスコアの変遷が書かれているのですが、試行錯誤がよくわかり学ぶところが多かったです。
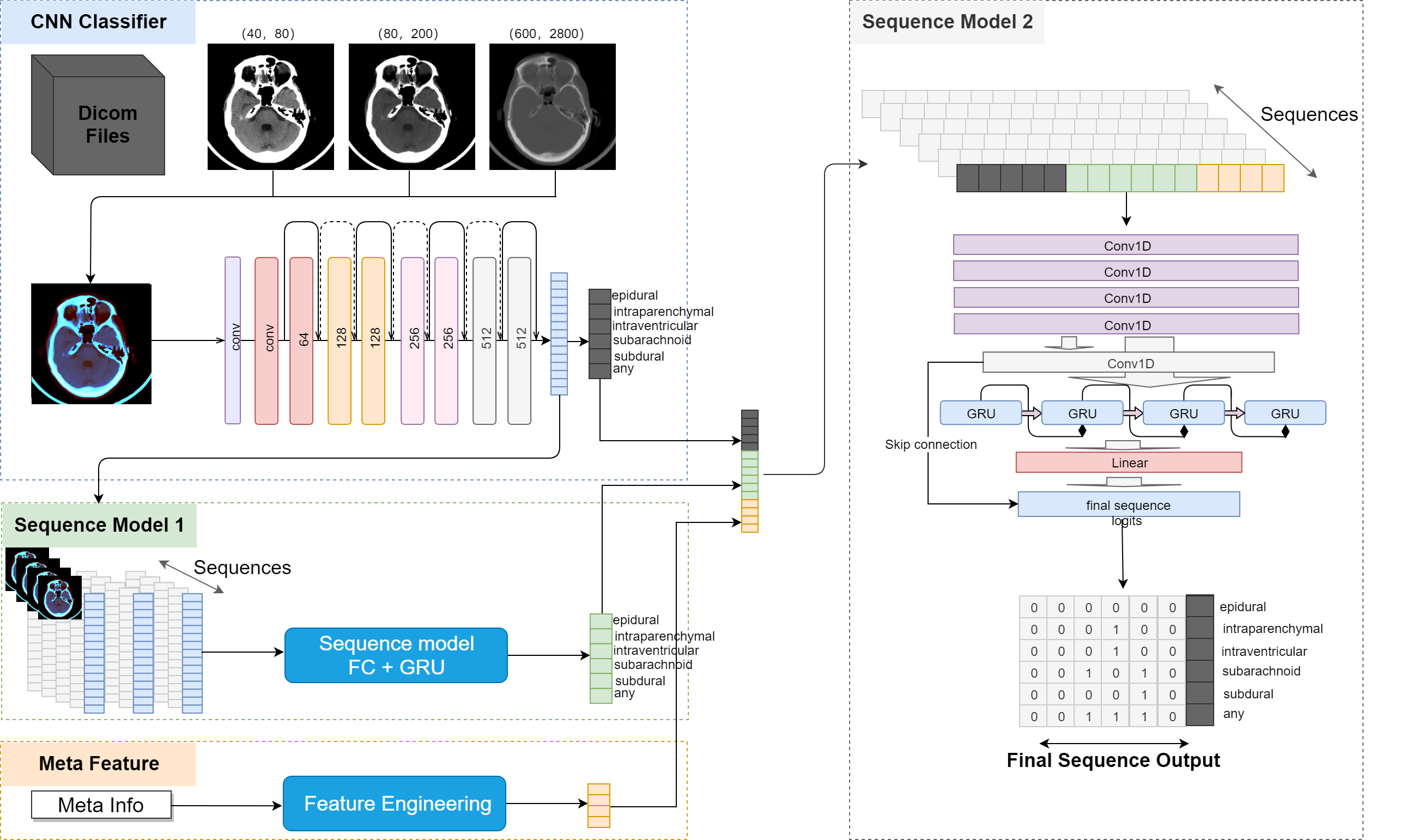
1位 SeuTaoチーム
1位のチームの解法はこちらから見れます。Githubはこちらです。
まず入力画像について説明します。入力画像は3つの方法で作成していました。
1. 1つのポジションに対して3つのWindowで切り取る
2. 前後も含めた3つのポジションに対して1つのWindowで切り取る
3. 3つ目が1と2を合わせたもの(3つのポジションに対して3つのWindowで切り取る)
入力画像の処理は他の上位チームと大差はなかったのではないかと思います。
1位のチームで特に印象的だったのは、**CNNを学習させて一度ラベルを予測したあとの2段階のSequenceモデルです。**図を見ると良くわかるのですが、1段階目でCNNの出力層直前のembeddingをポジションの順番に並べ、それをGRUを使って学習し、新しいラベルの予測をしています。そして2段階目では、まず各データについてCNNの予測ラベルと1段階目のSequenceモデルの予測ラベル、さらに隣接するスライスの距離情報を結合したベクトルを作成しています。そのようにして作成したベクトルをポジションの順番に並べ、GRUのモデルで学習して最終的な予測ラベルを作成しています。隣接するスライスとの距離を特徴量に用いたのは、スライスの間隔が違うと画像のクオリティが異なるというドメイン知識があったから使用したそうです。
では、この2段階のSequenceモデルがどのくらいスコアに貢献したのでしょうか。それについてはDiscussionにコメントが上がっていました。まずCNNのモデルの結果だけだと1st stageのPublic Leaderboardでは25位あたりだったのが、2段階目のSequenceモデルのConv 1Dを加えることで9位相当まで上がっています。そしてこのSequenceモデルに隣接スライスの距離情報とGRUを加えることで4位相当まで順位が上がっています。最後に、1段階目のSequenceモデルを入れることでPublic 1位、そして2nd stageのPrivateも堂々1位に輝いています。
まとめ
今回のコンペティションはスライスのポジション情報をいかにうまくモデルに組み込んで予測の精度を上げるかが上位に食い込むための重要ポイントの1つだったと思います。そして上位入賞者の中でもその方法は様々であり、解法を読んでいてとても勉強になりました。また、このような社会的に意義のあるコンペで多様性のあるモデルが生まれるということは、とても素晴らしいことではないでしょうか。今後もこのようなコンペがどんどん開催されることを期待したいです。
もし間違っているところなどございましたら、教えていただけますと幸いです。