一次元の頂点検出
[-1, 1]で畳み込み処理を行うと上昇している箇所は0を超えた値となり下降している箇所は0未満となる、この事を利用して上昇フラグ、下降フラグを作成、そのフラグが重なりあった箇所を頂点とする。
import math
import matplotlib
import matplotlib.pyplot as plt
import numpy as np
import PIL.Image
import random
import scipy.ndimage
# 一次元の頂点検出
x = np.array([0, 0, 1, 0, 0])
print("x", x, "対象配列")
conv1 = np.convolve(x, [1, -1], mode="full")
print("conv1", conv1, "[-1, 1]のフィルタにより上昇箇所、下降箇所の目印を作る")
flag1 = (conv1 > 0).astype(int)
print("flag1", flag1, "上昇箇所のフラグ")
flag2 = (conv1 <= 0).astype(int)
print("flag2", flag2, "下降箇所のフラグ")
flag1 = flag1[:-1]
print("flag1", flag1, "上昇フラグの末尾を1つ削って長さをあわせる")
flag2 = flag2[1:]
print("flag2", flag2, "下降フラグの先頭を1つ削って頂点箇所と合わせ、長さも揃える")
flag3 = flag1 & flag2
print("flag3", flag3, "上昇フラグと下降フラグを AND した結果が頂点箇所となる")
実行結果
x [0 0 1 0 0] 対象配列
conv1 [ 0 0 1 -1 0 0] [-1, 1]のフィルタにより上昇箇所、下降箇所の目印を作る
flag1 [0 0 1 0 0 0] 上昇箇所のフラグ
flag2 [1 1 0 1 1 1] 下降箇所のフラグ
flag1 [0 0 1 0 0] 上昇フラグの末尾を1つ削って長さをあわせる
flag2 [1 0 1 1 1] 下降フラグの先頭を1つ削って頂点箇所と合わせ、長さも揃える
flag3 [0 0 1 0 0] 上昇フラグと下降フラグを AND した結果が頂点箇所となる
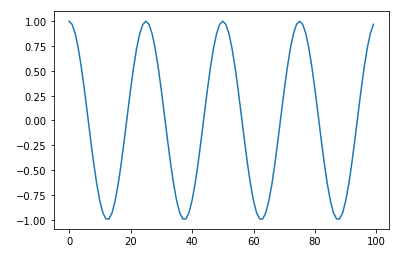
サンプルの作成
cycle = 4
data = np.zeros(100)
cycleWidth = len(data) / cycle
unit = math.pi / cycleWidth * 2
for i in range(cycle):
for j in range(int(cycleWidth)):
data[i * int(cycleWidth) + j] = math.cos(unit * float(j))
plt.plot(data)
plt.show()
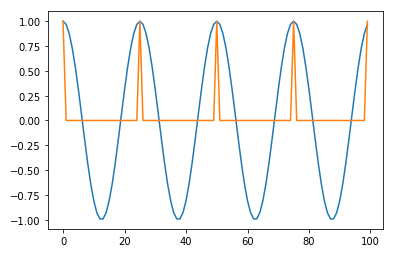
サンプルに対して頂点検出の実行
# 一次元の頂点検出を関数化しておく
def detectPeak1D(x):
conv1 = np.convolve(x, [1, -1], mode="full")
flag1 = (conv1 > 0).astype(int)
flag2 = (conv1 <= 0).astype(int)
flag1 = flag1[:-1]
flag2 = flag2[1:]
flag3 = flag1 & flag2
return flag3
peaks = detectPeak1D(data)
plt.plot(data)
plt.plot(peaks)
plt.show()
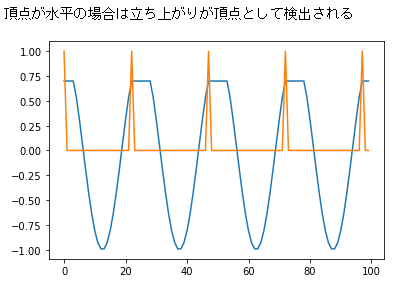
水平箇所については立ち上がりが頂点として検出される
data[data > 0.7] = 0.7
peaks = detectPeak1D(data)
print("頂点が水平の場合は立ち上がりが頂点として検出される")
plt.plot(data)
plt.plot(peaks)
plt.show()
二次元の頂点検出
一次元の頂点検出を「全ての行に対して行ったフラグ(二次元配列)」と「全ての列に対して行ったフラグ(二次元配列)」の2つをANDした結果を2次元データの頂点とする
# 2次元の頂点検出
x = np.array([
[0, 0, 1, 0, 0],
[0, 2, 3, 2, 0],
[1, 3, 5, 3, 1],
[0, 2, 3, 2, 0],
[0, 0, 1, 0, 0],
])
print(x, "対象データ")
# すべての行で頂点検出を実行
peaks1 = []
for ix in x:
peak = detectPeak1D(ix)
peaks1.append(peak)
peaks1 = np.array(peaks1)
print(peaks1, "横方向の頂点検出フラグ")
# すべての列で頂点検出を実行
peaks2 = []
for ix in x.transpose():
peak = detectPeak1D(ix)
peaks2.append(peak)
peaks2 = np.array(peaks2).transpose() # transposeにより検出を実行したのでもとに戻す
print(peaks2, "縦方向の頂点検出フラグ")
peaks3 = (peaks1 & peaks2).astype(int)
print(peaks3, "行、列の検出フラグをANDして残ったフラグが2次元の頂点フラグとなる")
実行結果
[[0 0 1 0 0]
[0 2 3 2 0]
[1 3 5 3 1]
[0 2 3 2 0]
[0 0 1 0 0]] 対象データ
[[0 0 1 0 0]
[0 0 1 0 0]
[0 0 1 0 0]
[0 0 1 0 0]
[0 0 1 0 0]] 横方向の頂点検出フラグ
[[0 0 0 0 0]
[0 0 0 0 0]
[1 1 1 1 1]
[0 0 0 0 0]
[0 0 0 0 0]] 縦方向の頂点検出フラグ
[[0 0 0 0 0]
[0 0 0 0 0]
[0 0 1 0 0]
[0 0 0 0 0]
[0 0 0 0 0]] 行、列の検出フラグをANDして残ったフラグが2次元の頂点フラグとなる
# 二次元の頂点検出を関数化しておく
def detectPeak2D(x):
peaks1 = []
for ix in x:
peak = detectPeak1D(ix)
peaks1.append(peak)
peaks1 = np.array(peaks1)
peaks2 = []
for ix in x.transpose():
peak = detectPeak1D(ix)
peaks2.append(peak)
peaks2 = np.array(peaks2).transpose()
flag = (peaks1 & peaks2).astype(int)
return flag
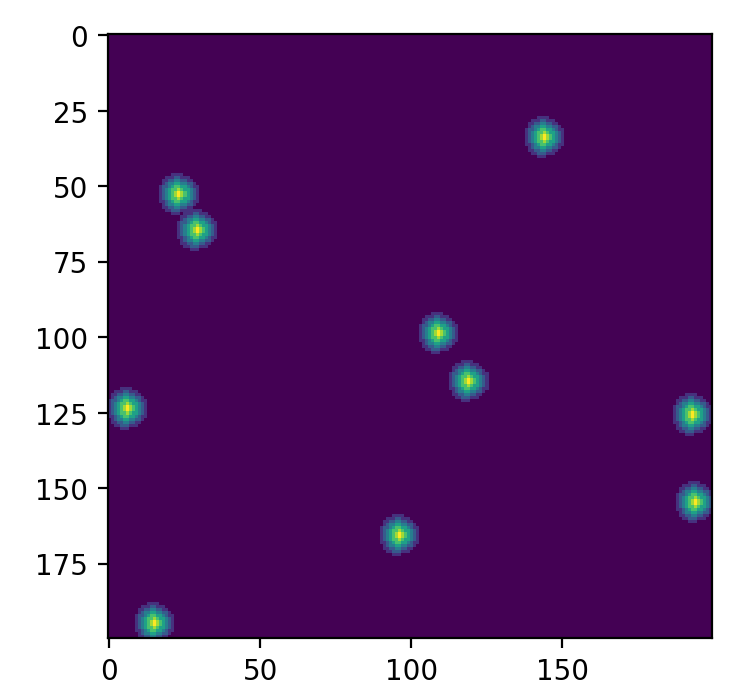
二次元データサンプルの作成
# 二次元頂点検出の試験用データを作成する、一次元の頂点のあるデータを回転させて作る
random.seed(1)
data2d = np.zeros((200, 200))
pattern = np.array([1, 2, 3, 4, 5, 6, 7, 6, 5, 4, 3, 2, 1])
for i in range(10):
ox, oy = random.randrange(200), random.randrange(200)
for j in range(50):
rad = (math.pi / 50) * j
for ix, v in enumerate(pattern):
pos = ix - len(pattern) / 2
x = ox + math.cos(rad) * pos
y = oy + math.sin(rad) * pos
if x < 0: continue
if x >= 200: continue
if y < 0: continue
if y >= 200: continue
data2d[int(x)][int(y)] = v
plt.figure(figsize=(10,4),dpi=200)
plt.imshow(data2d)
plt.show()
二次元サンプル画像
二次元頂点検出の実行
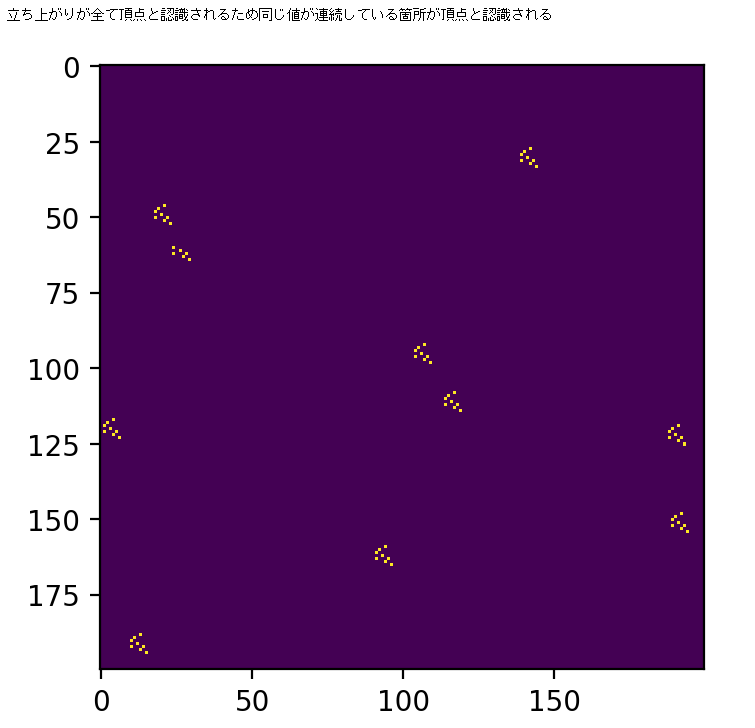
peaks = detectPeak2D(data2d)
print("立ち上がりが全て頂点と認識されるため同じ値が連続している箇所が頂点と認識される")
plt.figure(figsize=(10,4),dpi=200)
plt.imshow(peaks)
plt.show()
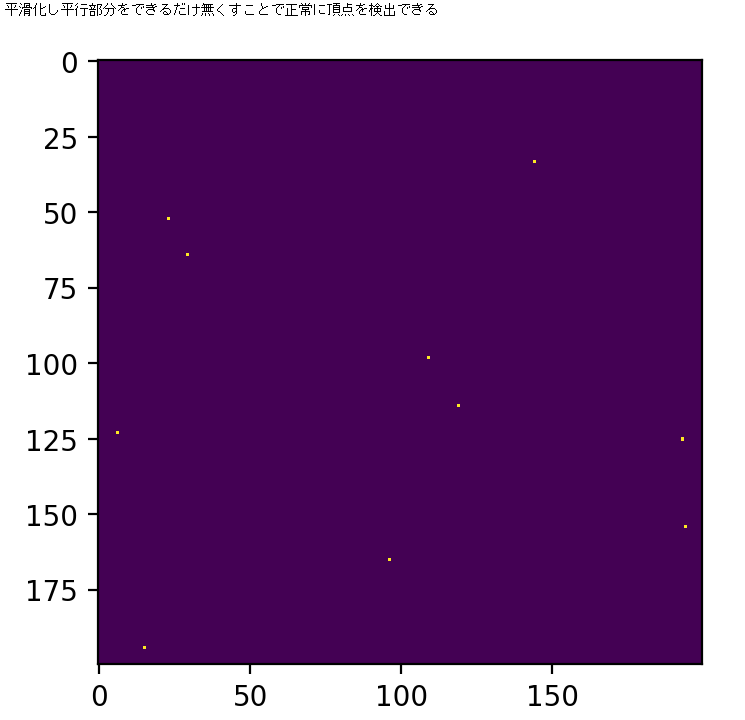
平滑化処理を施してから二次元頂点検出の実行
data2dGaussian = scipy.ndimage.gaussian_filter(data2d, sigma=1)
peaks = detectPeak2D(data2dGaussian)
print("平滑化し平行部分をできるだけ無くすことで正常に頂点を検出できる")
plt.figure(figsize=(10,4),dpi=200)
plt.imshow(peaks)
plt.show()
注意等
前処理も加工処理もなくキレイに山なりの形をしているデータを想定した処理です、基本的には平滑化してからでないと想定通りの動作をしない事が多いと思います、そのまま使うのではなく移動平均でもガウシアンフィルタでもなんでも良いので平滑化処理を行ってから使用します。
以上です。