100 万個くらいのラベルとそのラベルに対応する値があって、その中から 100 個くらいのラベルに対して、対応する値を取得したい。
説明のために適当にデータを生成してみる。
generate_random_labels <- function(num, length = 10) {
apply(Vectorize(sample, "size")(letters, rep(num, length), replace = TRUE), 1, function(row) paste(row, collapse = ""))
}
generate_random_values <- function(labels, digits = 1) {
format <- paste0("%s%0", digits, "d")
sprintf(format, labels, Vectorize(sample, "size")(seq_len(10^digits-1), rep(1,length(labels))))
}
N <- 1000
labels <- generate_random_labels(N)
values <- generate_random_values(labels)
df <- data.frame(labels, values, stringsAsFactors = FALSE)
head(df)
labels values
1 itfftodouk itfftodouk7
2 oalbnlpxnj oalbnlpxnj6
3 idtnwbwoss idtnwbwoss8
4 fgqqbnyzmv fgqqbnyzmv7
5 ioyqgcrbto ioyqgcrbto6
6 ypkcdfbjvr ypkcdfbjvr1
1000 個のラベルと値のペアを作成した。
この中から適当に 5 個のラベルを抽出する。
target <- sample(labels, 5)
target
[1] "lqogquxoql" "zeipgvkjvu" "snnqnsjeuw" "povvjfdaow" "kztoedvhju"
このラベルに対応した値を取得したいというのが今回の目的だ。
こういう場合はハッシュテーブルを使うと便利なのだが R言語にはハッシュテーブルが無い。
最初に思いつくのはデータフレームを使ったこんなやり方だ。
df[df$labels %in% target, ]$values
[1] "povvjfdaow8" "snnqnsjeuw4" "kztoedvhju1" "lqogquxoql9" "zeipgvkjvu7"
今回は結果の順序はどうでもいいので一応これで OK だ。
だが、実際に適用してみると非常に遅い。
この動作は、何千回、何万回と繰り返すため、できるだけ速い方法を探したい。
今回は R でハッシュテーブルぽいものを使う方法をいろいろと考えて、速度比較してみた。
1. 名前付きベクトル
R でハッシュテーブルを実現する一つのやり方は名前付きベクトルを使うことである。
named_values <- values
names(named_values) <- labels
named_values[target]
lqogquxoql zeipgvkjvu snnqnsjeuw povvjfdaow kztoedvhju
"lqogquxoql9" "zeipgvkjvu7" "snnqnsjeuw4" "povvjfdaow8" "kztoedvhju1"
非常にお手軽にできてわかりやすい。
2. 名前付きリスト
同様に、名前付きリストもハッシュテーブルとして使える。
named_values_list <- as.list(values)
names(named_values_list) <- labels
unlist(named_values_list[target])
lqogquxoql zeipgvkjvu snnqnsjeuw povvjfdaow kztoedvhju
"lqogquxoql9" "zeipgvkjvu7" "snnqnsjeuw4" "povvjfdaow8" "kztoedvhju1"
3. 環境
『R言語徹底解説』第8章(p.160)には、R における環境(environment)の特別な使用方法としてハッシュマップとして使えると書いてある。
実際に書いてみるとこんなコードになった。
my_env <- new.env(parent = emptyenv())
invisible(Vectorize(assign, c("x", "value"))(labels, values, envir = my_env))
unlist(mget(target, envir = my_env))
lqogquxoql zeipgvkjvu snnqnsjeuw povvjfdaow kztoedvhju
"lqogquxoql9" "zeipgvkjvu7" "snnqnsjeuw4" "povvjfdaow8" "kztoedvhju1"
コードは複雑になるがハッシュテーブルを実現できている。
4. hash パッケージ
『R言語徹底解説』第8章(p.160)では、環境をハッシュテーブルとして使うのに便利な hash パッケージについても言及がある。
library(hash)
h <- hash(labels, values)
values(h[target])
kztoedvhju lqogquxoql povvjfdaow snnqnsjeuw zeipgvkjvu
"kztoedvhju1" "lqogquxoql9" "povvjfdaow8" "snnqnsjeuw4" "zeipgvkjvu7"
非常に手軽である。順序が保持されないことには注意が必要である。
5. データベース(SQLite)
データベースでもハッシュテーブルと同じことができる。
一見オーバーヘッドが大きそうだが、データサイズが大きい場合は結局データベースを使った方が速くなるということもある。
dplyr パッケージと RSQLite パッケージを使えばかなり手軽に SQLite を使うことができるので軽く紹介したい。
# install.packages("RSQLite")
library(dplyr)
temp_tbl <- dplyr:::temp_load(temp_srcs("sqlite"), list(df=df))$sqlite$df
temp_tbl %>% filter(labels %in% target) %>% select(values) %>% collect %>% {.$values}
[1] "povvjfdaow8" "snnqnsjeuw4" "kztoedvhju1" "lqogquxoql9" "zeipgvkjvu7"
これも順序は保存されない。
速度比較
上で紹介した 6 つの方法について速度を比較してみる。
テーブルサイズ N を 100~1000000 まで変化させて速度を調べている。
library(magicfor)
magic_for()
for(N in 10^(2:6)) {
# Create Data -------------------------------------------------------------
labels <- generate_random_labels(N)
values <- generate_random_values(labels)
df <- data.frame(labels, values, stringsAsFactors = FALSE)
target <- sample(labels, 100)
# Preparation -------------------------------------------------------------
named_values <- values
names(named_values) <- labels
named_values_list <- as.list(values)
names(named_values_list) <- labels
my_env <- new.env(parent = emptyenv())
invisible(Vectorize(assign, c("x", "value"))(labels, values, envir = my_env))
library(hash)
h <- hash(labels, values)
library(dplyr)
# install.packages("RSQLite")
temp_tbl <- dplyr:::temp_load(temp_srcs("sqlite"), list(df=df))$sqlite$df
# Benchmark ---------------------------------------------------------------
library(microbenchmark)
mb_result <- microbenchmark(
data_frame = {
df[df$labels %in% target, ]$values
},
named_vector = {
named_values[target]
},
named_list ={
unlist(named_values_list[target])
},
environment = {
unlist(mget(target, envir = my_env))
},
hash = {
values(h[target])
},
sqlite = {
temp_tbl %>% filter(labels %in% target) %>% select(values) %>% collect %>% {.$values}
}
)
time_df <- data.frame(N=as.character(N), mb_result)
print(time_df)
}
result <- Reduce(rbind, magic_result()$time_df)
library(ggplot2)
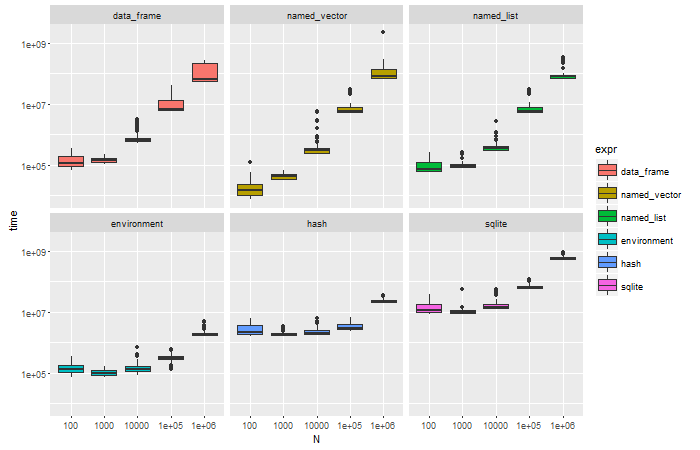
ggplot(result, aes(x=N, y=time, fill=expr)) +
geom_boxplot() + scale_y_log10() + facet_wrap(~expr)
result_mean <- result %>% group_by(N, expr) %>% summarise(mean=mean(time))
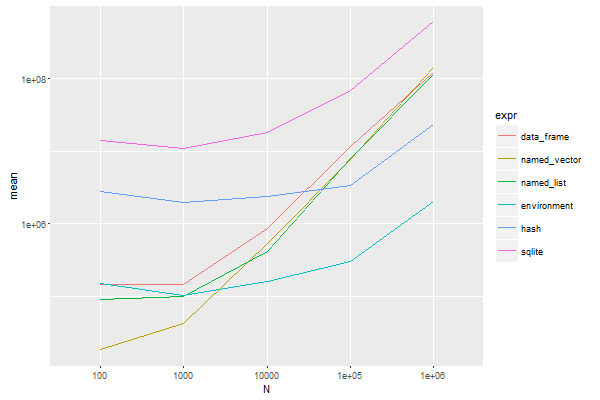
ggplot(result_mean, aes(x=N, y=mean, group=expr, color=expr)) +
geom_line() + scale_y_log10()
テーブルサイズ N が 1000 ぐらいまでは名前付きベクトルが速いが、10000以上になると環境を使った方法が速いようである。
私の今回の用途だと環境を直接用いて実装した方が速くなりそうだ。
hash は便利だが、汎用向けなのか実装がまずいのか、オーバーヘッドが大きいようだ。
Enjoy!