(追記)
PDBIDからパーシステント図を見るWebアプリ。
Protein Holes
(学生時代の研究室サーバに立てているので動かなくなってるかも)
はじめに
今回は、パーシステントホモロジーを用いてタンパク質の穴を可視化してみました。
パーシステントホモロジーとタンパク質
パーシステントホモロジーとは、新しい位相的データ解析手法のことです。
一言で説明すると、データに含まれる「穴」に注目した解析手法です。
詳しくは、「位相的データ解析の基礎と応用」をご覧ください。
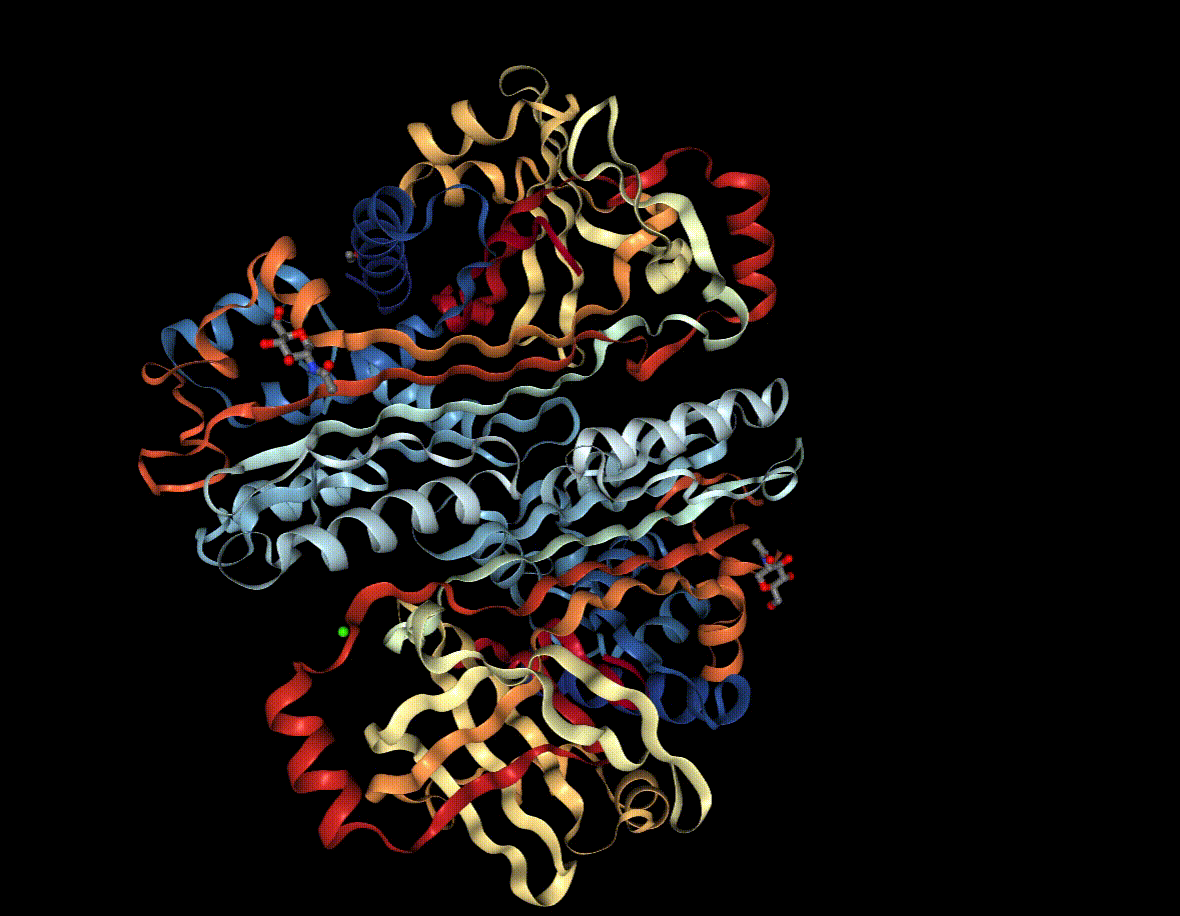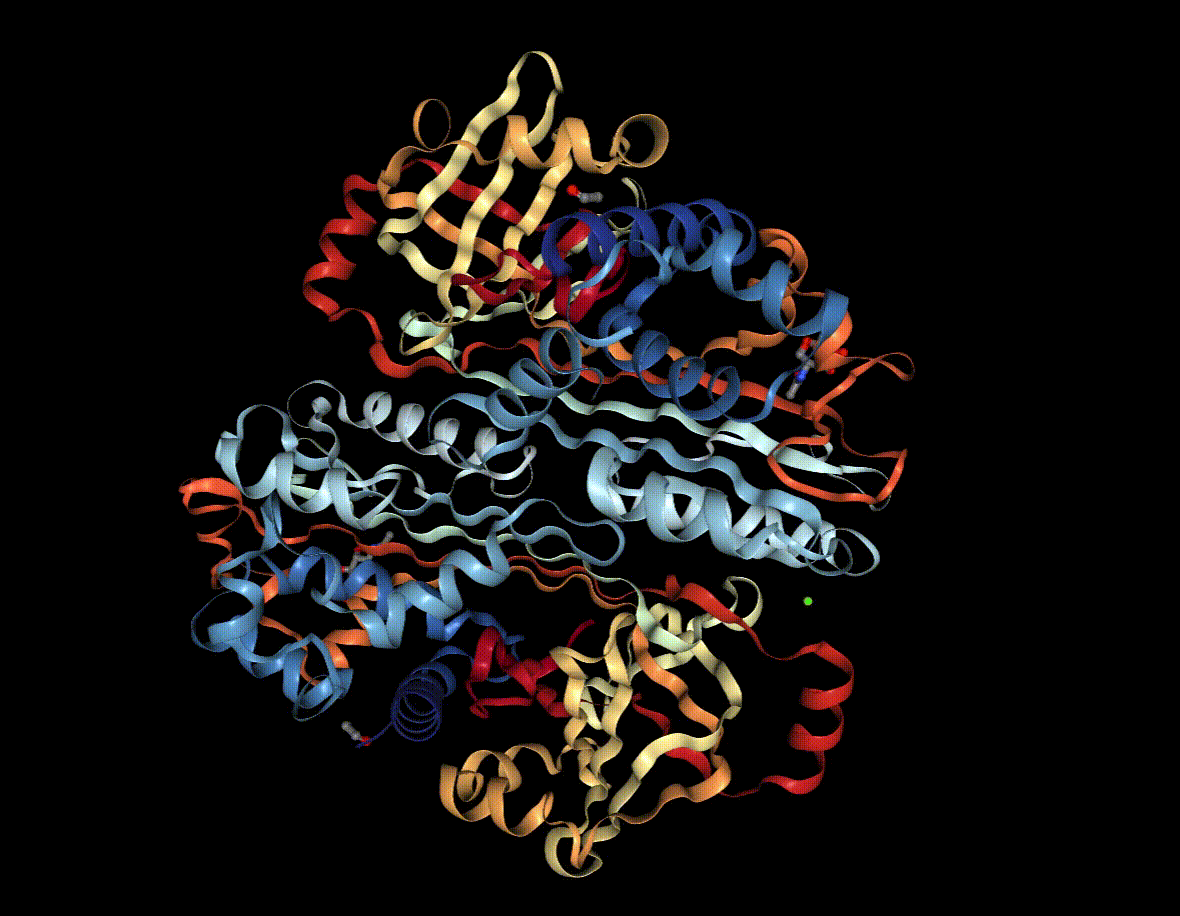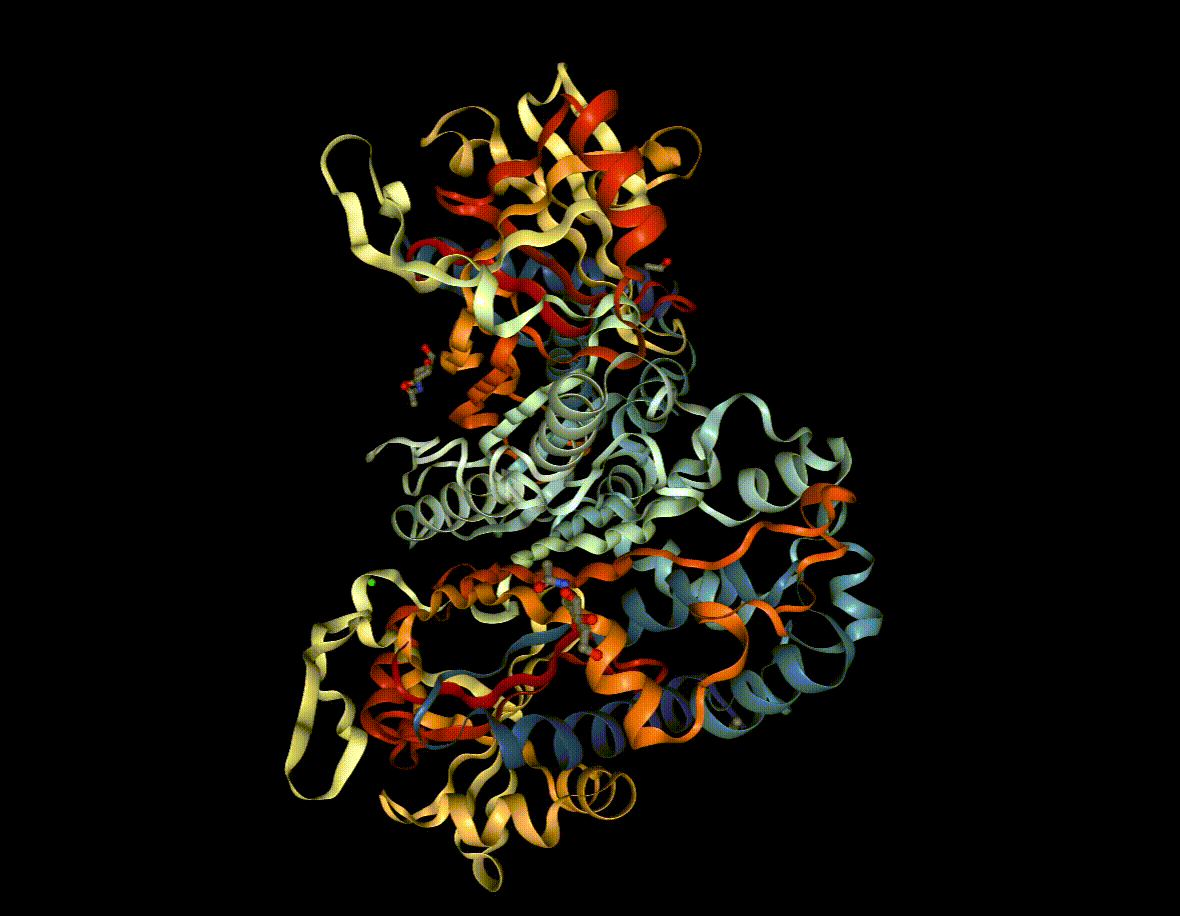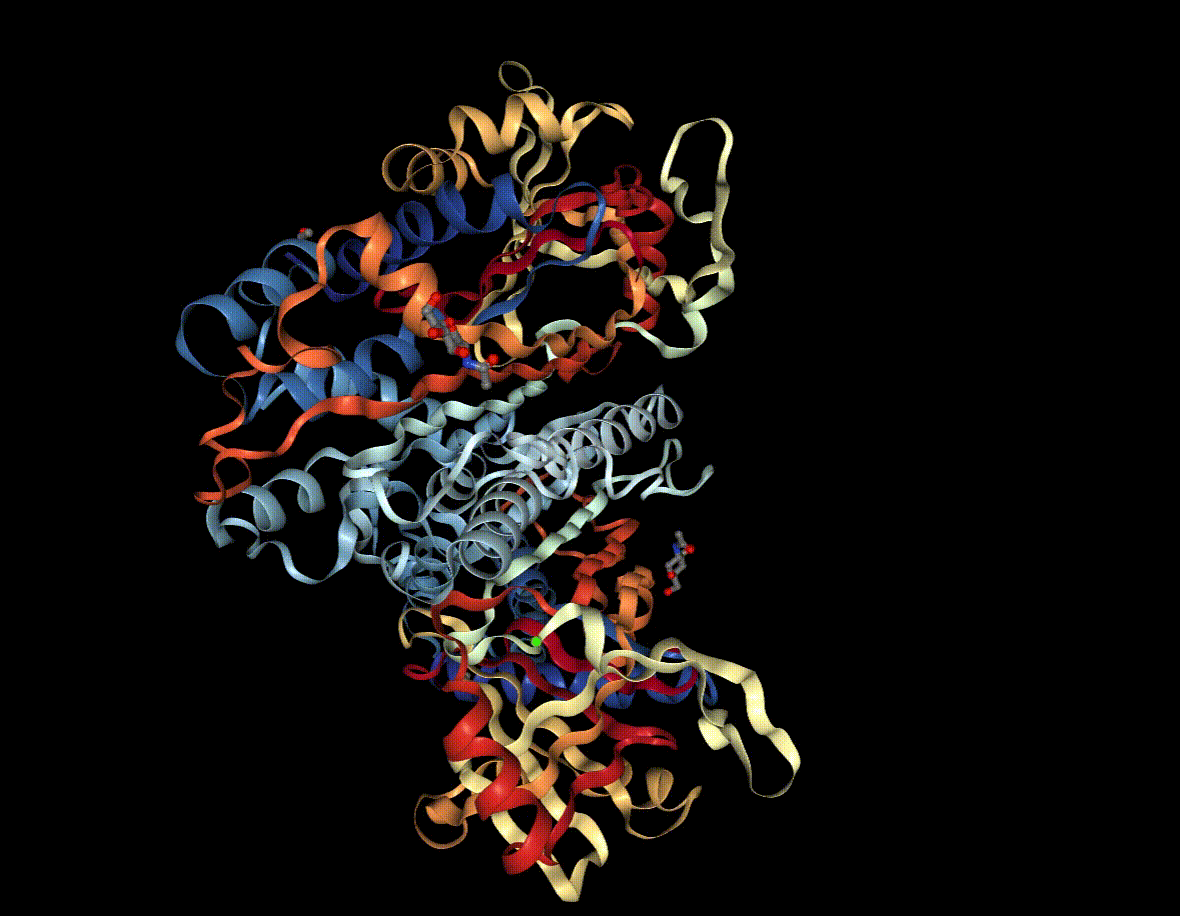
データは、"uncleaved ovalbumin"(1OVA)というタンパク質のアミノ酸主鎖座標(x, y, z)を使いました。
uncleaved ovalbuminは以下のような構造をしています。
このタンパク質に存在する穴をパーシステントホモロジーを使って可視化してみようと思います。
パーシステントホモロジーの計算には、HomCloudを使いました。
PDBファイル(タンパク質構造のファイル:Protein Data Bankから入手可能)からアミノ酸主鎖座標を抜き出す部分のソースコードを公開しています。
ソースコードは、こちらからご覧ください。
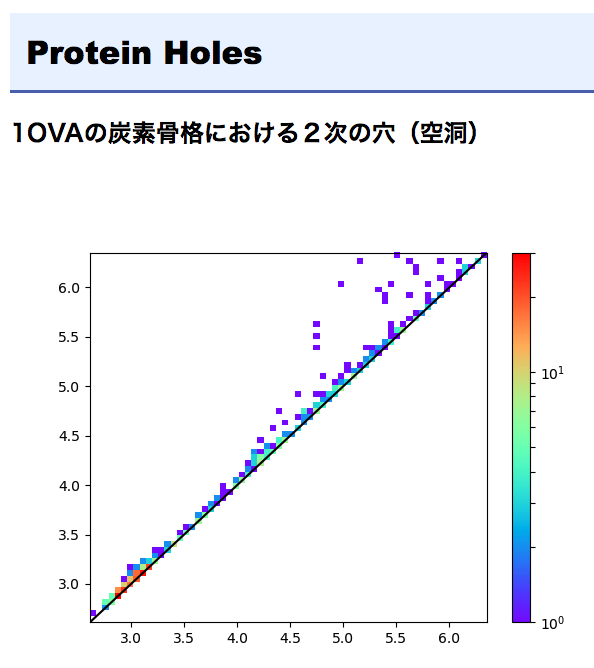
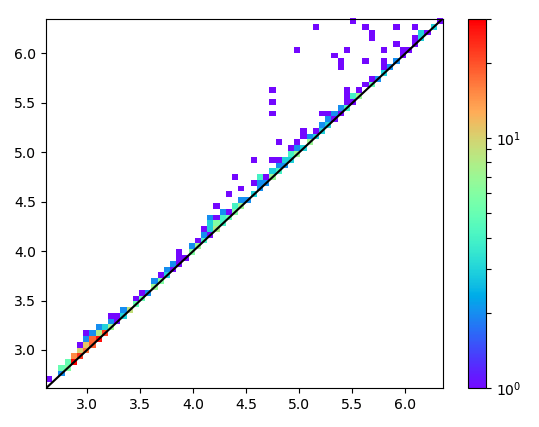
結果
uncleaved ovalbuminの2次(空洞)のパーシステントホモロジーを計算した後のプロット図です。
(追記)
HomCloudが更新されていて、インストール時のエラーが無くなっていました。
インストールもpipで簡単にできるようになりました。
(MacOXでも普通にインストールできました。)
さらに、以前は使えなかった拡張部分が使えるようになっていました。
# HomCloudのインストール時のエラー
HomCloudのインストール時につまずいたところをいくつかあげて起きます。
原因は分かりませんが、インストールはできました。
1、"cmake"ができない
私のPC環境はmac High Sierraですが、"cmake"だけではエラーがでました。
"cmake CMakeList.txt"とすることで解決できました。
2、"pip3 install homcloud-base-x.y.z.tar.gz"ができない
pyenvのpythonを使っていると、なぜかインストールできませんでした。
以下のようにして、systemのpythonに切り替えることでインストールできました。
$ pyenv global system