基本的な表示法
PyMOLで水素結合を表示するには、
GUIのウィンドウから、
[A]ction -> [find] -> [polar contacts]
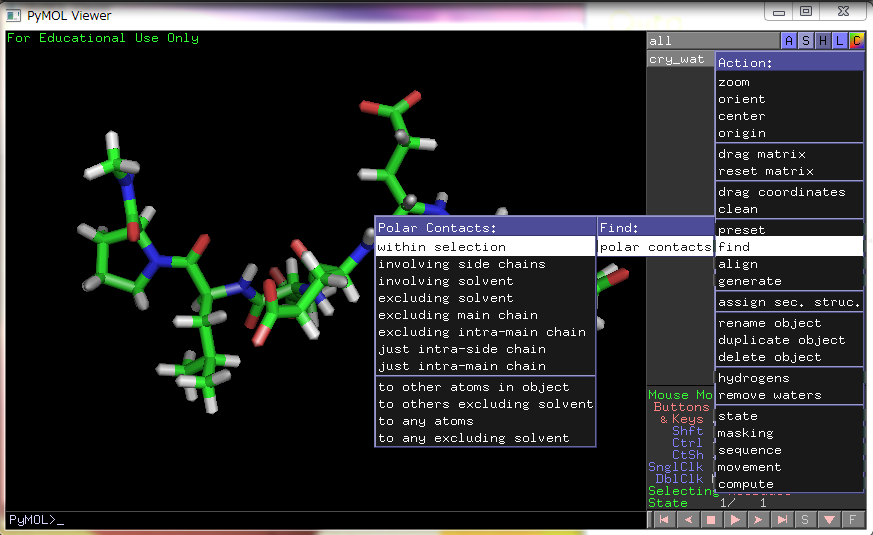
とする方法と、
CUIで、
distance <name>, <selection A>, <selection B>, mode=2

とする方法の2通りある。
ちなみに、CUIでコマンドで打つと距離も表示してくれる。
デフォルトの問題点
デフォルトで水素結合を表示してみると
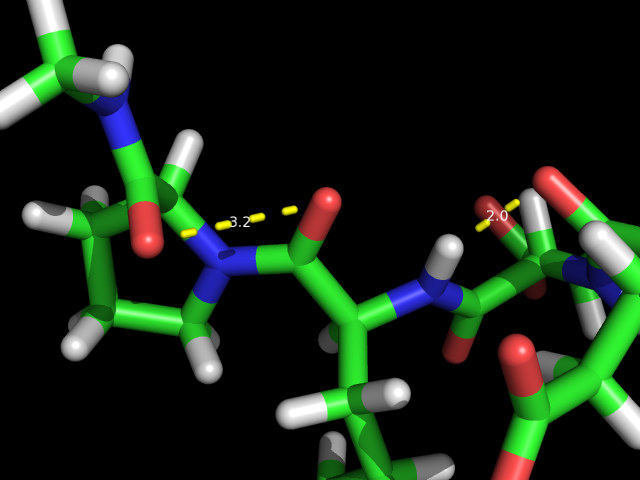
図のように水素結合を示す破線を表示してくれる。
しかしながら、実は、この2つは水素結合ではない。
左のほうは、どちらの酸素原子もカルボニル基の酸素である。つまり二重結合を作っている酸素であるため、間に水素が介在していない。
水素結合(すいそけつごう、英: hydrogen bond)は、電気陰性度が大きな原子(陰性原子)に共有結合で結びついた水素原子が、近傍に位置した窒素、酸素、硫黄、フッ素、π電子系などの孤立電子対とつくる非共有結合性の引力的相互作用である。
(引用:https://ja.wikipedia.org/wiki/%E6%B0%B4%E7%B4%A0%E7%B5%90%E5%90%88)
右のほうは、N-H-O の角度が大きすぎる。
また、水素が付いていない場合(X線結晶構造解析では水素が見えないためPDBに登録されているほとんどに水素がない)、
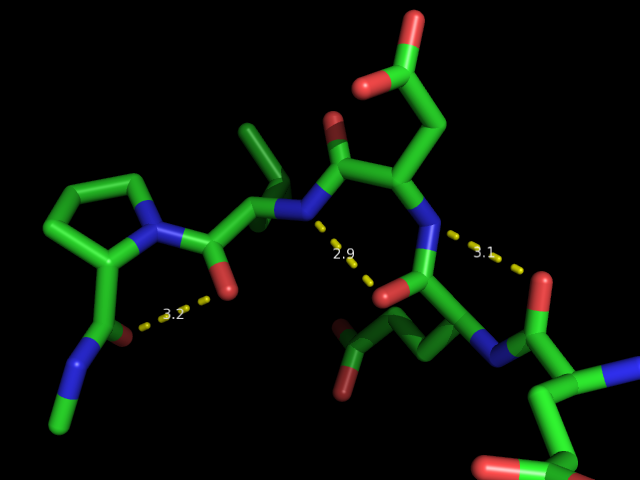
一番右に表示されている破線のように、HがないためにN-H-O の角度を考慮せず、距離だけで、水素結合が引かれてしまう。
水素結合の長さは、結合の強さ、温度、圧力に依存している。結合の強さ自身は、温度、圧力、結合角度、局所的な誘電率などの環境に依存している。典型的な水における水素結合の長さは197 pmである。理想的な結合角度は水素結合供与体の性質に依存している。
(引用:https://ja.wikipedia.org/wiki/%E6%B0%B4%E7%B4%A0%E7%B5%90%E5%90%88)
pymolのデフォルトの設定で引いた水素結合には、けっこう誤りが多い。
水素結合表示するときのパラメータをセット
そこで、
set h_bond_cutoff_center, 3.5 #原子間距離 3.5 Å
set h_bond_max_angle, 30 #最大角 30°
とコマンドを入力することで、設定できる。
それでも、
左の、カルボニル酸素同士に間違った結合が示されている。
ので、手動で消すしかない。
結局、pymolの[find]機能を利用せず、自分の目で見てやるほうがいいかもしれません。
DSSPプログラムで水素結合(主鎖だけ)を調べる。
Linux(ubuntu(Debian))の場合、
sudo apt-get install dssp
でdsspプログラムを使える。
実行方法は、
mkdssp filename.pdb > log.txt
==== Secondary Structure Definition by the program DSSP, CMBI version by M.L. Hekkelman/2010-10-21 ==== DATE=2015-12-21 .
REFERENCE W. KABSCH AND C.SANDER, BIOPOLYMERS 22 (1983) 2577-2637 .
.
COMPND .
SOURCE .
AUTHOR .
9 1 0 0 0 TOTAL NUMBER OF RESIDUES, NUMBER OF CHAINS, NUMBER OF SS-BRIDGES(TOTAL,INTRACHAIN,INTERCHAIN) .
1223.7 ACCESSIBLE SURFACE OF PROTEIN (ANGSTROM**2) .
1 11.1 TOTAL NUMBER OF HYDROGEN BONDS OF TYPE O(I)-->H-N(J) , SAME NUMBER PER 100 RESIDUES .
0 0.0 TOTAL NUMBER OF HYDROGEN BONDS IN PARALLEL BRIDGES, SAME NUMBER PER 100 RESIDUES .
0 0.0 TOTAL NUMBER OF HYDROGEN BONDS IN ANTIPARALLEL BRIDGES, SAME NUMBER PER 100 RESIDUES .
0 0.0 TOTAL NUMBER OF HYDROGEN BONDS OF TYPE O(I)-->H-N(I-5), SAME NUMBER PER 100 RESIDUES .
0 0.0 TOTAL NUMBER OF HYDROGEN BONDS OF TYPE O(I)-->H-N(I-4), SAME NUMBER PER 100 RESIDUES .
0 0.0 TOTAL NUMBER OF HYDROGEN BONDS OF TYPE O(I)-->H-N(I-3), SAME NUMBER PER 100 RESIDUES .
0 0.0 TOTAL NUMBER OF HYDROGEN BONDS OF TYPE O(I)-->H-N(I-2), SAME NUMBER PER 100 RESIDUES .
0 0.0 TOTAL NUMBER OF HYDROGEN BONDS OF TYPE O(I)-->H-N(I-1), SAME NUMBER PER 100 RESIDUES .
0 0.0 TOTAL NUMBER OF HYDROGEN BONDS OF TYPE O(I)-->H-N(I+0), SAME NUMBER PER 100 RESIDUES .
0 0.0 TOTAL NUMBER OF HYDROGEN BONDS OF TYPE O(I)-->H-N(I+1), SAME NUMBER PER 100 RESIDUES .
1 11.1 TOTAL NUMBER OF HYDROGEN BONDS OF TYPE O(I)-->H-N(I+2), SAME NUMBER PER 100 RESIDUES .
0 0.0 TOTAL NUMBER OF HYDROGEN BONDS OF TYPE O(I)-->H-N(I+3), SAME NUMBER PER 100 RESIDUES .
0 0.0 TOTAL NUMBER OF HYDROGEN BONDS OF TYPE O(I)-->H-N(I+4), SAME NUMBER PER 100 RESIDUES .
0 0.0 TOTAL NUMBER OF HYDROGEN BONDS OF TYPE O(I)-->H-N(I+5), SAME NUMBER PER 100 RESIDUES .
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 *** HISTOGRAMS OF *** .
0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 RESIDUES PER ALPHA HELIX .
0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 PARALLEL BRIDGES PER LADDER .
0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 ANTIPARALLEL BRIDGES PER LADDER .
0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 LADDERS PER SHEET .
# RESIDUE AA STRUCTURE BP1 BP2 ACC N-H-->O O-->H-N N-H-->O O-->H-N TCO KAPPA ALPHA PHI PSI X-CA Y-CA Z-CA
1 1 P L 0 0 123 0, 0.0 3,-0.0 0, 0.0 0, 0.0 0.000 360.0 360.0 360.0-104.3 2.9 31.1 -12.5
2 2 P P - 0 0 144 0, 0.0 2,-0.0 0, 0.0 0, 0.0 -0.996 360.0-171.6 -70.7 -10.7 1.0 29.0 -11.6
3 3 P G - 0 0 61 2,-0.1 2,-0.1 3,-0.0 3,-0.1 -0.161 38.8 -25.3 83.4 178.8 -1.1 30.2 -14.6
4 4 P E S S- 0 0 194 1,-0.1 2,-0.2 -2,-0.0 0, 0.0 -0.370 76.9 -94.8 -64.2 137.1 -4.6 29.3 -15.8
5 5 P E - 0 0 158 1,-0.1 2,-2.0 -2,-0.1 -1,-0.1 -0.349 33.2-139.8 -52.7 117.9 -5.9 25.9 -14.7
6 6 P D + 0 0 160 -2,-0.2 -1,-0.1 -3,-0.1 -2,-0.0 -0.393 68.0 98.2 -87.9 64.2 -5.2 23.5 -17.6
7 7 P L S S- 0 0 119 -2,-2.0 -2,-0.0 0, 0.0 0, 0.0 -0.977 78.9 -89.9-144.6 150.6 -8.5 21.7 -17.4
8 8 P P 0 0 114 0, 0.0 -2,-0.0 0, 0.0 -3,-0.0 -0.114 360.0 360.0 -62.8 154.8 -11.7 22.1 -19.5
9 9 P G 0 0 151 0, 0.0 0, 0.0 0, 0.0 0, 0.0 0.854 360.0 360.0 -51.8 360.0 -14.6 24.4 -18.6
1 11.1 TOTAL NUMBER OF HYDROGEN BONDS OF TYPE O(I)-->H-N(I+2), SAME NUMBER PER 100 RESIDUES .
より、I残基目のカルボニル酸素と、I+2残基目の窒素原子が水素結合を作っているとわかる。
参考URL