はじめに
この記事では「はじめてのパターン認識」(通称はじぱた)の第4章4.1節のデータの前処理(線形変換)の数値例をPython(numpyのみ)で実装する。
データのロード
Scikit-kearnのload_iris()を使う。
from sklearn.preprocessing import StandardScaler
import numpy as np
from matplotlib import pyplot as plt
from sklearn.datasets import load_iris
from sklearn import preprocessing
iris = load_iris()
import pandas as pd
df = pd.DataFrame(iris.data, columns=iris.feature_names)
df['target'] = iris.target
df.loc[df['target'] == 0, 'target'] = "setosa"
df.loc[df['target'] == 1, 'target'] = "versicolor"
df.loc[df['target'] == 2, 'target'] = "virginica"
データの中身を確認する。
df.describe()
| sepal length (cm) | sepal width (cm) | petal length (cm) | petal width (cm) | |
|---|---|---|---|---|
| count | 150.000000 | 150.000000 | 150.000000 | 150.000000 |
| mean | 5.843333 | 3.057333 | 3.758000 | 1.199333 |
| std | 0.828066 | 0.435866 | 1.765298 | 0.762238 |
| min | 4.300000 | 2.000000 | 1.000000 | 0.100000 |
| 25% | 5.100000 | 2.800000 | 1.600000 | 0.300000 |
| 50% | 5.800000 | 3.000000 | 4.350000 | 1.300000 |
| 75% | 6.400000 | 3.300000 | 5.100000 | 1.800000 |
| max | 7.900000 | 4.400000 | 6.900000 | 2.500000 |
データの統計量を確認する。
df.cov()
| sepal length (cm) | sepal width (cm) | petal length (cm) | petal width (cm) | |
|---|---|---|---|---|
| sepal length (cm) | 0.685694 | -0.042434 | 1.274315 | 0.516271 |
| sepal width (cm) | -0.042434 | 0.189979 | -0.329656 | -0.121639 |
| petal length (cm) | 1.274315 | -0.329656 | 3.116278 | 1.295609 |
| petal width (cm) | 0.516271 | -0.121639 | 1.295609 | 0.581006 |
今回は「petal length」と「petal width」を対象にする。
x=df['petal length (cm)']
y=df['petal width (cm)']
z=df['target']
setosa = list(z.reset_index().query('target == "setosa"').index)
versicolor = list(z.reset_index().query('target == "versicolor"').index)
virginica = list(z.reset_index().query('target == "virginica"').index)
グラフ描画用の関数を作っておく。
def data_plot():
plt.rcParams['font.size'] = 14
plt.rcParams['font.family'] = 'Times New Roman'
#目盛を内側にする。
plt.rcParams['xtick.direction'] = 'in'
plt.rcParams['ytick.direction'] = 'in'
#グラフの上下左右に目盛線を付ける。
fig = plt.figure()
ax1 = fig.add_subplot(111)
ax1.yaxis.set_ticks_position('both')
ax1.xaxis.set_ticks_position('both')
#軸のラベルを設定する。
ax1.set_xlabel('x')
ax1.set_ylabel('y')
#元データ
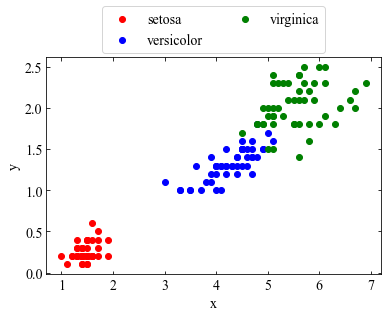
ax1.scatter(setosa_x, setosa_y, label='setosa', c='red')
ax1.scatter(versicolor_x, versicolor_y, label='versicolor', c='blue')
ax1.scatter(virginica_x, virginica_y, label='virginica', c='green')
return ax1
ax1=data_plot()
fig.tight_layout()
ax1.legend(loc="lower center", bbox_to_anchor=(0.5, 1.02,), borderaxespad=0, ncol=2)
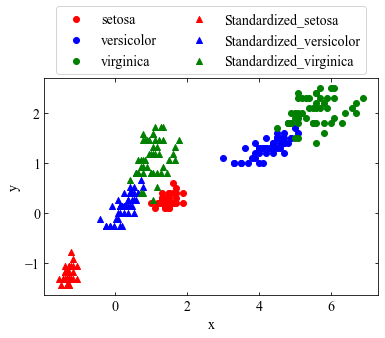
標準化
標準化とは測定単位の影響を取り除くべく、観測データの平均を0、分散を1にする変換のことである。
元のデータ$x$の平均$E\{x\}$を$\mu$、分散$Var\{x\}$を$\sigma$としたとき、線形変換
$$z=\frac{x-\mu}{\sigma}$$
を考えると、
$$E\{z\}=0、Var\{z\}=1$$
となる。
これらの関係が成り立つことを実装して確認する。
# numpyで平均、分散それぞれを計算する。
# 平均
ave_x=np.average(x)
ave_y=np.average(y)
# 分散
stdx=np.std(x)
stdy=np.std(y)
# 標準化の線形変換
nrom_x=[]
norm_y=[]
for i in range(len(x)):
norm_x.append((x[i]-ave_x)/stdx)
norm_y.append((y[i]-ave_y)/stdy)
#setosa, viersicolor, virginicaそれぞれをプロットするために配列を準備
setosa_x=[]
versicolor_x=[]
virginica_x=[]
setosa_y=[]
versicolor_y=[]
virginica_y=[]
n_setosa_x=[]
n_versicolor_x=[]
n_virginica_x=[]
n_setosa_y=[]
n_versicolor_y=[]
n_virginica_y=[]
for i in setosa:
setosa_x.append(x[i])
setosa_y.append(y[i])
n_setosa_x.append(norm_x[i])
n_setosa_y.append(norm_y[i])
for i in versicolor:
versicolor_x.append(x[i])
versicolor_y.append(y[i])
n_versicolor_x.append(norm_x[i])
n_versicolor_y.append(norm_y[i])
for i in virginica:
virginica_x.append(x[i])
virginica_y.append(y[i])
n_virginica_x.append(norm_x[i])
n_virginica_y.append(norm_y[i])
【参考】Scikit-learnを使う場合
# x,yを結合
data = np.c_[x, y]
# 標準化を行う
sc = StandardScaler()
std = sc.fit_transform(data)
グラフを描画する。
ax1=data_plot()
ax1.scatter(n_setosa_x, n_setosa_y, label='Standardized_setosa', marker='^', c='red')
ax1.scatter(n_versicolor_x, n_versicolor_y, label='Standardized_versicolor', marker='^', c='blue')
ax1.scatter(n_virginica_x, n_virginica_y, label='Standardized_virginica', marker='^', c='green')
fig.tight_layout()
ax1.legend(loc="lower center", bbox_to_anchor=(0.5, 1.02,), borderaxespad=0, ncol=2)
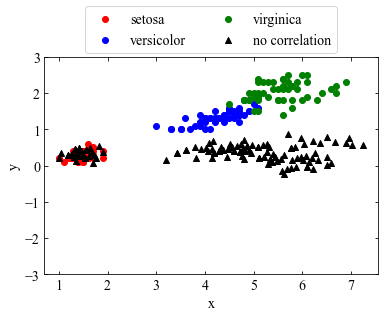
無相関化
無相関化とは、観測データ間の相関をなくす処理のことである。観測データから得られた共分散行列$\Sigma$の固有値問題
$$\Sigma \boldsymbol{s}=\lambda \boldsymbol{s}$$
を解いて得られた固有値を$\lambda_{1} \geqq \lambda_{2} \geqq \cdot\cdot\cdot\geqq\lambda_{d} $、
対応する固有ベクトルを$\boldsymbol{s_{1}, s_{2}, \cdot\cdot\cdot, s_{d}}$として、行列$\boldsymbol{S}$を以下で定義する。
$$\boldsymbol{S}=(\boldsymbol{s_{1}, s_{2}, \cdot\cdot\cdot, s_{d}})$$
観測データ$\boldsymbol{x}$に対して$\boldsymbol{S}^{T}$で線形変換を行う。すなわち、$\boldsymbol{y}=\boldsymbol{S}^{T}\boldsymbol{x}$を考えたとき、以下が成り立つ。
$$E\{\boldsymbol{y}\}=\boldsymbol{S}^{T}\boldsymbol{\mu}, Var\{\boldsymbol{y}\}=\boldsymbol{S}^{-1} \boldsymbol{\Sigma}\boldsymbol{S}=\boldsymbol{\Lambda}$$
$\boldsymbol{S}^{-1} \boldsymbol{\Sigma}\boldsymbol{S}$は$\boldsymbol{\Sigma}$の対角化そのものであるため、対角成分以外は0となる。すなわち、共分散は0となり、相関係数の定義から相関係数は0となることがわかる。
# 分散共分散行列の回転行列Sを求める。
_, S = np.linalg.eig(cov)
S[:,1]=S[:,1]*(-1)
$\boldsymbol{S}^{T}=$$\boldsymbol{S}^{-1}$が成り立つ。すなわち、$S^{T}$$S$が単位行列であることが確認できる。
np.dot(S.T, S)
[[ 1.00000000e+00, -2.53792738e-17],
[-2.53792738e-17, 1.00000000e+00]]
# データを無相関化 ※.Tは転置を表す
pledged_decorr = np.dot(S.T, data.T).T
$\Lambda = \boldsymbol{S}^{-1} \boldsymbol{\Sigma}\boldsymbol{S}$を求める。
lam = np.dot(np.dot(np.linalg.inv(S), cov), S)
はじぱたp.43の式(4.16)と同じ値であることを確認する。
print(lam)
[[3.66123805e+00 2.31134308e-16]
[1.20962250e-16 3.60460707e-02]]
相関係数が0であることを確認する。
print('相関係数: {:.3f}'.format(np.corrcoef(pledged_decorr[:, 0], pledged_decorr[:, 1])[0,1]))
相関係数: 0.000
グラフを描画する。
ax1=data_plot()
ax1.scatter(pledged_decorr[:, 0], pledged_decorr[:, 1], label='no correlation', marker='^', c='black')
plt.ylim(-3,3)
fig.tight_layout()
ax1.legend(loc="lower center", bbox_to_anchor=(0.5, 1.02,), borderaxespad=0, ncol=2)
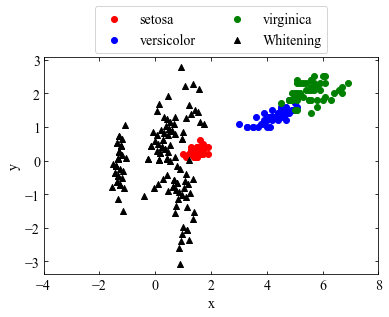
白色化
前述の$\boldsymbol{\Lambda}、\boldsymbol{S}^{T}$、観測データ$\boldsymbol{x}$の平均$\boldsymbol{\mu}$を用いて、線形変換$\boldsymbol{u}=\boldsymbol{\Lambda}^{-\frac{1}{2}}\boldsymbol{S}^{T}(\boldsymbol{x}-\boldsymbol{u})$を考えると、以下が成り立つ。
$$E\{\boldsymbol{u}\}=0$$
$$Var\{\boldsymbol{u}\}=\boldsymbol{\Lambda}^{-\frac{1}{2}}\boldsymbol{S}^{-1}\boldsymbol{\Sigma}\boldsymbol{S}\boldsymbol{\Lambda}^{-\frac{T}{2}}
=\boldsymbol{\Lambda}^{-\frac{1}{2}}\boldsymbol{\Lambda}\boldsymbol{\Lambda}^{\frac{1}{2}}=\boldsymbol{I}$$
ただし、$\boldsymbol{\Lambda}^{-\frac{T}{2}}$は$\boldsymbol{\Lambda}^{-\frac{1}{2}}$の転置行列である。このように観測データの標準偏差を1に正規化し、かつ中心化する変換を白色化と呼ぶ。
データを白色化する。
ave=np.c_[ave_x, ave_y]
lam_1_2=np.linalg.inv(np.sqrt(lam))
u = np.dot(lam_1_2, np.dot(S.T, (df_corr-ave).T))
グラフを描画する。
ax1=data_plot()
plt.xlim(-4,8)
ax1.scatter(u.T[:, 0], u.T[:, 1], label='Whitening', marker='^', c='black')
fig.tight_layout()
ax1.legend(loc="lower center", bbox_to_anchor=(0.5, 1.02,), borderaxespad=0, ncol=2)
最後に白色化後のデータの共分散行列が単位行列になっていることを確認する。
np.cov(u)
確かに単位行列になっている。
array([[ 1.00000000e+00, -6.58744352e-08],
[-6.58744352e-08, 1.00000000e+00]])