RAPIDSとは
RAPIDSは一言でいうとGPUのパワーでデータ処理を高速化するNVIDIAが用意しているライブラリです。
データサイエンスでよく使われるライブラリと対応しています。
| RAPIDS | 対応ライブラリ |
|---|---|
| cuDF | Pandas |
| cuML | scikit-learn |
| cuGraph | NetworkX |
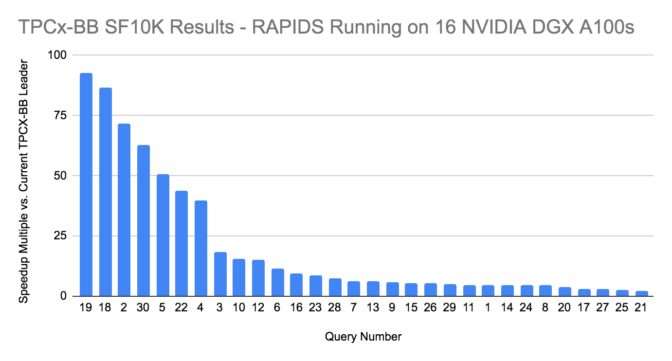
TPCx-BBのベンチマークは、30のクエリでの結果です。16台のDGX A100システム上でRAPIDSを実行することで、10TBのテストでは、クエリごとに上記のような相対的なパフォーマンス向上を実現しています。
Dockerで環境設定
dockerhubにRAPIDSのdocker imageがありますので利用します。
2022年4月3日現在下記コマンドでjupyter labを起動します。
docker pull rapidsai/rapidsai:22.02-cuda11.0-runtime-ubuntu18.04-py3.8
docker run --gpus all --rm -it -p 8888:8888 -p 8787:8787 -p 8786:8786 rapidsai/rapidsai:22.02-cuda11.0-runtime-ubuntu18.04-py3.8
http://localhost:8888/lab をブラウザで開くと下記の通り、あらかじめtutorialなどが格納されたフォルダが用意されています。
試しに動かす
試しに cuml/random_forest_demo.ipynb を動かしてみます。
| 処理 | Scikit-learn Model | cuML Model |
|---|---|---|
| Fit | 38.8s | 1.31s |
| Evaluate | 25.4ms | 306ms |
Evaluete は m秒単位で遅くなっていますが、Fitが秒単位で圧倒的に早いですね。
Benchmarkを動かす
githubにあるBenchmarkを動かす。
Docker起動後に下記でクローンして、データセットをダウンロードしておく。
mkdir rapidsai
cd rapidsai
git clone https://github.com/rapidsai/cugraph.git
cd cugraph/notebooks/cugraph_benchmarks/
sh ./dataPrep.sh
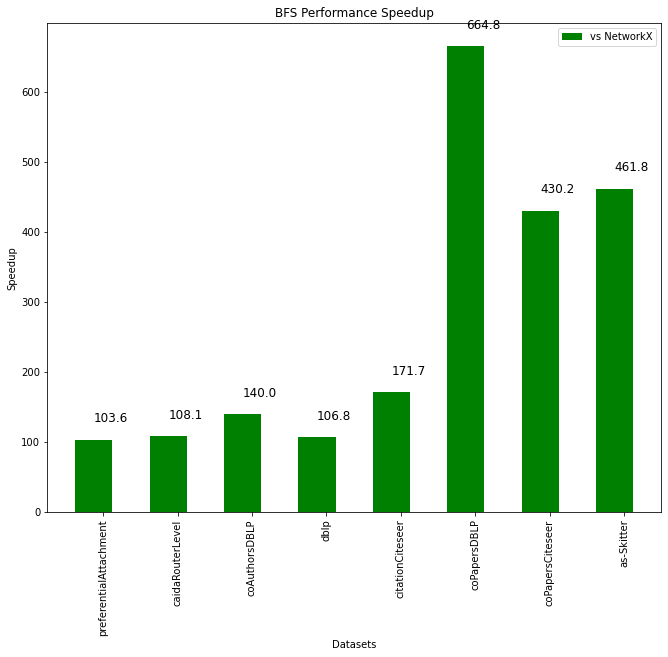
rapidsai/cugraph/notebooks/cugraph_benchmarks/bfs_benchmark.ipynb を実行する。
実行結果として表示されるグラフは下記の通り。
環境にもよるかと思いますが、なんと100~600倍ほどの高速化がされます。
CPUの処理時間がかかるので、処理に20分くらいかかりました。
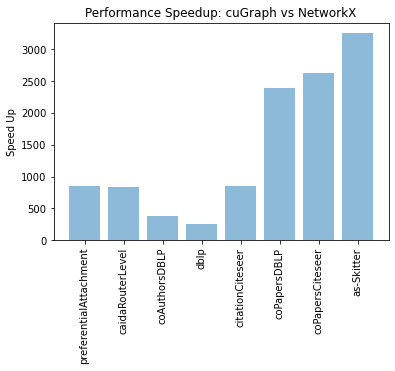
rapidsai/cugraph/notebooks/cugraph_benchmarks/louvain_benchmark.ipynb を実行して、表示されるグラフは下記の通り。
cuGraphはいずれの処理も2秒以内で終わっていますが、CPU側で2時間以上かかりました。
圧倒的じゃないか、わが軍は!
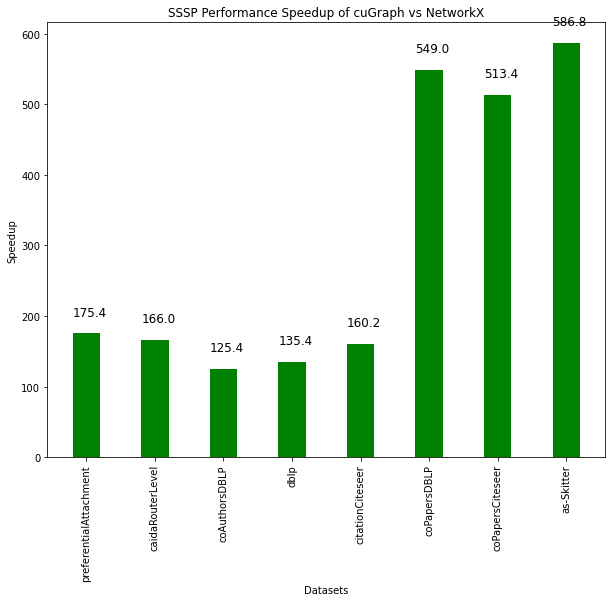
rapidsai/cugraph/notebooks/cugraph_benchmarks/sssp_benchmark.ipynb を実行して、表示されるグラフは下記の通り。
cuGraphはいずれの処理も1秒以内で終わっていますが、CPU側で20分ほどかかりました。
単一始点最短路問題(SSSP)