はじめに
ディープラーニングではエポック毎にミニバッチの学習データをランダムにシャッフルするのが一般的みたいなのでその効果を確かめてみました。
環境
- python: 2.7.6
- chainer: 1.8.0
学習内容
エポック毎にミニバッチの学習データを固定にした際とランダムにした際の違いを確認します。
学習させるのはchainerでsin関数を学習させてみた際と同じsin関数です。
[training data]
- input: theta(0~2π, 1000分割)
- output: sin(theta)
実装
固定 or ランダム
ミニバッチの学習データを固定とランダムに切り替えています。固定時(fixed)のコードはテスト時によく用いられる手法と同じです。
perm = np.random.permutation(N)
sum_loss = 0
for i in range(0, N, batchsize):
if order == "fixed": # 学習する順番が固定
x_batch = x_train[i:i + batchsize]
y_batch = y_train[i:i + batchsize]
elif order == "random": # 学習する順番がランダム
x_batch = x_train[perm[i:i + batchsize]]
y_batch = y_train[perm[i:i + batchsize]]
学習パラメータ
- ミニバッチサイズ(batchsize): 20
- エポック(n_epoch): 500
- 隠れ層の数: 2
- 隠れ層のユニット数(n_units): 100
- 活性化関数: 正規化線形関数(relu)
- ドロップアウト(dropout): なし(0%)
- 最適化: Adam
- 損失誤差関数: 平均二乗誤差関数(mean_squared_error)
パラメータは例のごとく全て適当。
コード全体
# -*- coding: utf-8 -*-
# とりあえず片っ端からimport
import numpy as np
import chainer
from chainer import cuda, Function, gradient_check, Variable, optimizers, serializers, utils
from chainer import Link, Chain, ChainList
import chainer.functions as F
import chainer.links as L
import time
from matplotlib import pyplot as plt
# データ
def get_dataset(N):
x = np.linspace(0, 2 * np.pi, N)
y = np.sin(x)
return x, y
# ニューラルネットワーク
class MyChain(Chain):
def __init__(self, n_units=10):
super(MyChain, self).__init__(
l1=L.Linear(1, n_units),
l2=L.Linear(n_units, n_units),
l3=L.Linear(n_units, 1))
def __call__(self, x_data, y_data):
x = Variable(x_data.astype(np.float32).reshape(len(x_data),1)) # Variableオブジェクトに変換
y = Variable(y_data.astype(np.float32).reshape(len(y_data),1)) # Variableオブジェクトに変換
return F.mean_squared_error(self.predict(x), y)
def predict(self, x):
h1 = F.relu(self.l1(x))
h2 = F.relu(self.l2(h1))
h3 = self.l3(h2)
return h3
def get_predata(self, x):
return self.predict(Variable(x.astype(np.float32).reshape(len(x),1))).data
# main
if __name__ == "__main__":
# 学習データ
N = 1000
x_train, y_train = get_dataset(N)
# 学習パラメータ
batchsize = 10
n_epoch = 500
n_units = 100
# 学習ループ
fixed_losses =[]
random_losses =[]
print "start..."
for order in ["fixed", "random"]:
# モデル作成
model = MyChain(n_units)
optimizer = optimizers.Adam()
optimizer.setup(model)
start_time = time.time()
for epoch in range(1, n_epoch + 1):
# training
perm = np.random.permutation(N)
sum_loss = 0
for i in range(0, N, batchsize):
if order == "fixed": # 学習する順番が固定
x_batch = x_train[i:i + batchsize]
y_batch = y_train[i:i + batchsize]
elif order == "random": # 学習する順番がランダム
x_batch = x_train[perm[i:i + batchsize]]
y_batch = y_train[perm[i:i + batchsize]]
model.zerograds()
loss = model(x_batch,y_batch)
sum_loss += loss.data * batchsize
loss.backward()
optimizer.update()
average_loss = sum_loss / N
if order == "fixed":
fixed_losses.append(average_loss)
elif order == "random":
random_losses.append(average_loss)
# 学習過程を出力
if epoch % 10 == 0:
print "({}) epoch: {}/{} loss: {}".format(order, epoch, n_epoch, average_loss)
interval = int(time.time() - start_time)
print "実行時間({}): {}sec".format(order, interval)
print "end"
# 誤差のグラフ作成
plt.plot(fixed_losses, label = "fixed_loss")
plt.plot(random_losses, label = "random_loss")
plt.yscale('log')
plt.legend()
plt.grid(True)
plt.title("loss")
plt.xlabel("epoch")
plt.ylabel("loss")
plt.show()
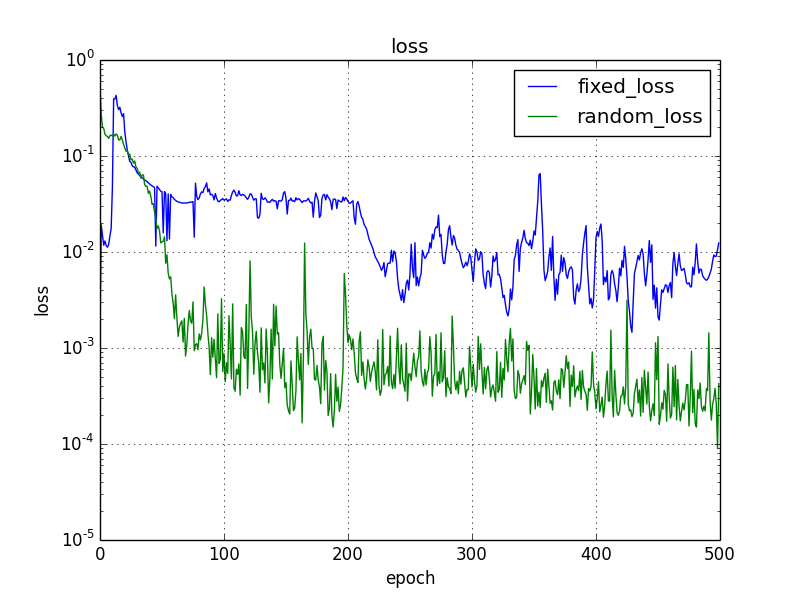
実行結果
誤差
固定時とランダム時で誤差に10倍程度の差がでました。前評判通りランダムにした方がよい結果です。
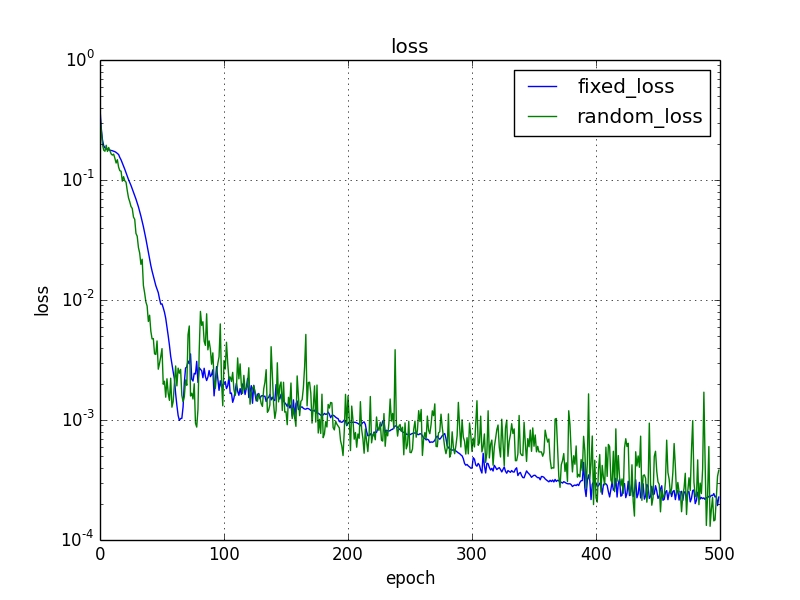
データ全体をシャッフルにした場合
学習データ全体をランダムにシャッフルした場合も確認しました。
学習データ全体をランダムにシャッフルすると固定時もランダム時と遜色がない結果になりました。むしろエポック毎のバラツキは固定時の方がよいです。
def get_dataset(N):
x = 2 * np.pi * np.random.random(N)
y = np.sin(x)
return x, y
まとめ
sin関数を学習する際にミニバッチの学習データをエポック毎にランダムシャッフルするとしないときと比べて誤差が10倍程度よくなりました。ただし、学習データ全体をランダムするとどちらもよい結果になりました。
sin関数などの特定の条件に限るのかもしませんが、データ全体をランダムシャッフル、ミニバッチの学習データを固定する方法も試す価値ありです。