はじめに
流速分布を測定する必要があり,PIV(Particle Image Velocimetry)を実施可能か試してみる.よく知らないが商用版は数十から数百万と言われており,高いため無料のOpenPIVで行う.
方法を簡単に説明すると流体に粒子を分散させてレーザーシートを照射して,粒子からの散乱光を時系列で計測する.粒子の移動量と時間差から速度を求める手法である.粒子の移動量の計算はほとんどのソフトウェアで直接相互相関法とFFT相互相関法の2つです.詳しくは下記のサイトや本を参考にしてください.
PIVとは | カトウ光研株式会社
PIV(粒子画像流速測定法)とは | 日本カノマックス株式会社
OpenPIVとは
OpenPIVは,PIVの画像解析および後処理のためのフリーのオープンソースのソフトウェアです.どちらの方式を採用しているかはよくわからなかった.OpenPIVはMatlab,C++,Pythonの3つが使用可能で,今回はPython版を使ってみる.
OpenPIVの使用,結果
コードは下記に記事を参考に作成した.時間差(dt)やスケール(scaling_factor)は特に設定せずに1とした.
from openpiv import tools, validation, filters, scaling, pyprocess
img_a0 = tools.imread('B003_1.tif')
img_b0 = tools.imread('B003_2.tif')
u0, v0, sig2noise = pyprocess.extended_search_area_piv(img_a0.astype(np.int32), img_b0.astype(np.int32), window_size=32, overlap=20, dt=1, search_area_size=64, sig2noise_method='peak2peak')
x, y = pyprocess.get_coordinates(image_size=img_a0.shape, search_area_size=64, overlap=20)
u1, v1, mask = validation.sig2noise_val(u0, v0, sig2noise, threshold=1.5)
u2, v2 = filters.replace_outliers(u1, v1, method='localmean', max_iter=5, kernel_size=2)
x, y, u3, v3 = scaling.uniform(x, y, u2, v2, scaling_factor=1)
x, y, u3, v3 = tools.transform_coordinates(x, y, u3, v3)
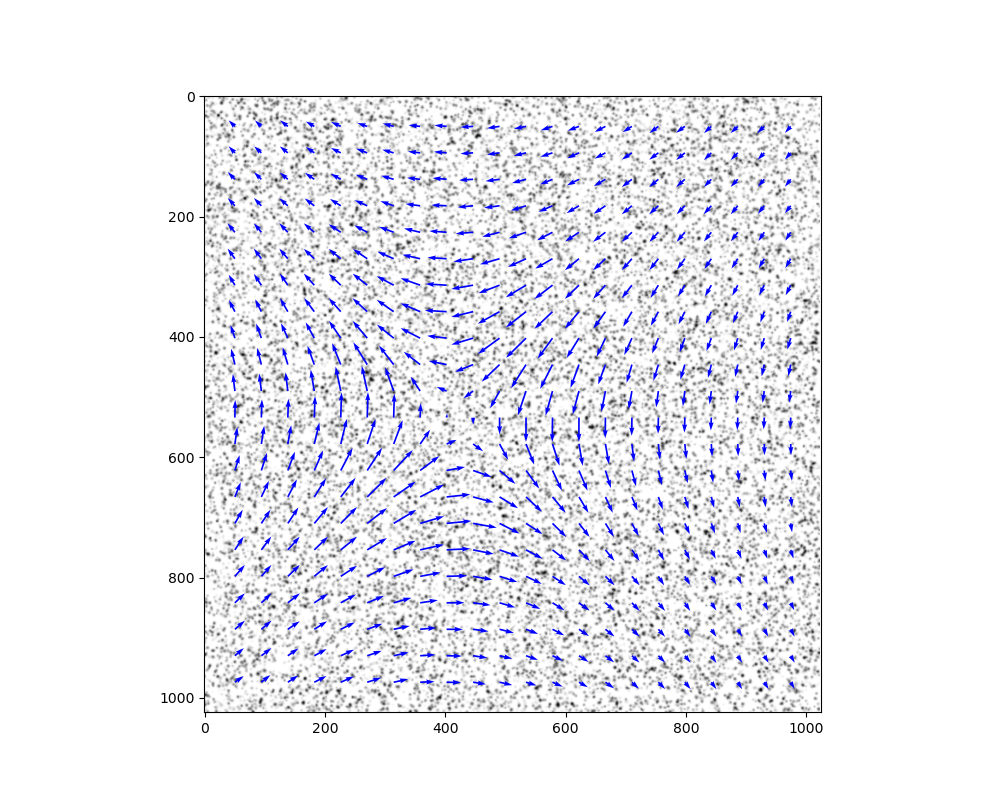
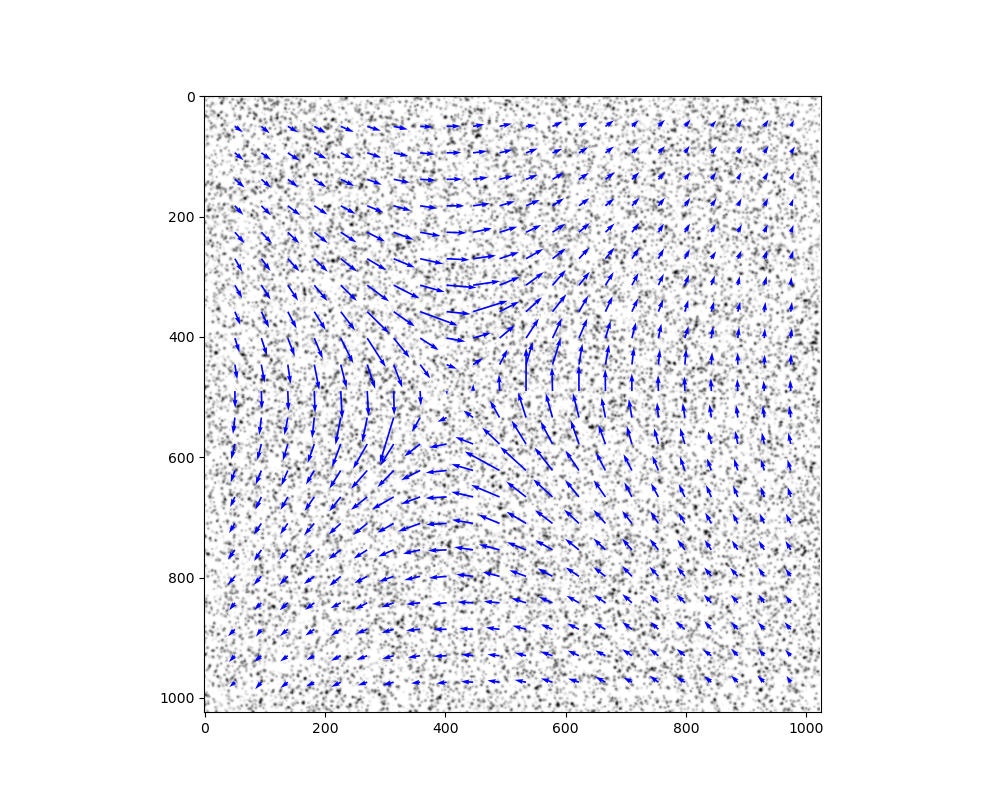
実験は行えないので画像はPIVChallengeのから1st PIV Challenge (Sept.14-15, 2001, Göttingen, Germany) のCase 3: medium particle density and small particle を使用した.実際の実験のデータであるため,今後行う実験に近いと考えて使用した.通常の層流では問題点はわからないと考えて渦のデータを使用した.下記のgifを見ると画像中心部に渦があることをが確認できる.目視の結果が再現できれば簡単な速度場ならOpenPIVが業務に使用できると考えられる.
下記の図がOpenPIVで解析した結果となる.渦ができるはずなのに対向した流れになっている.窓,オーバーラップ,閾値などの値を調整したがうまくいかなかった.コード内部を確認して調整することは時間を要するため,今後は他の方法でPIVを検討する.
追記(2023/01/31/10:30,14:50)
記事を参考にした@fiftystorm36様からコメントをいただき処理の誤りを確認した.
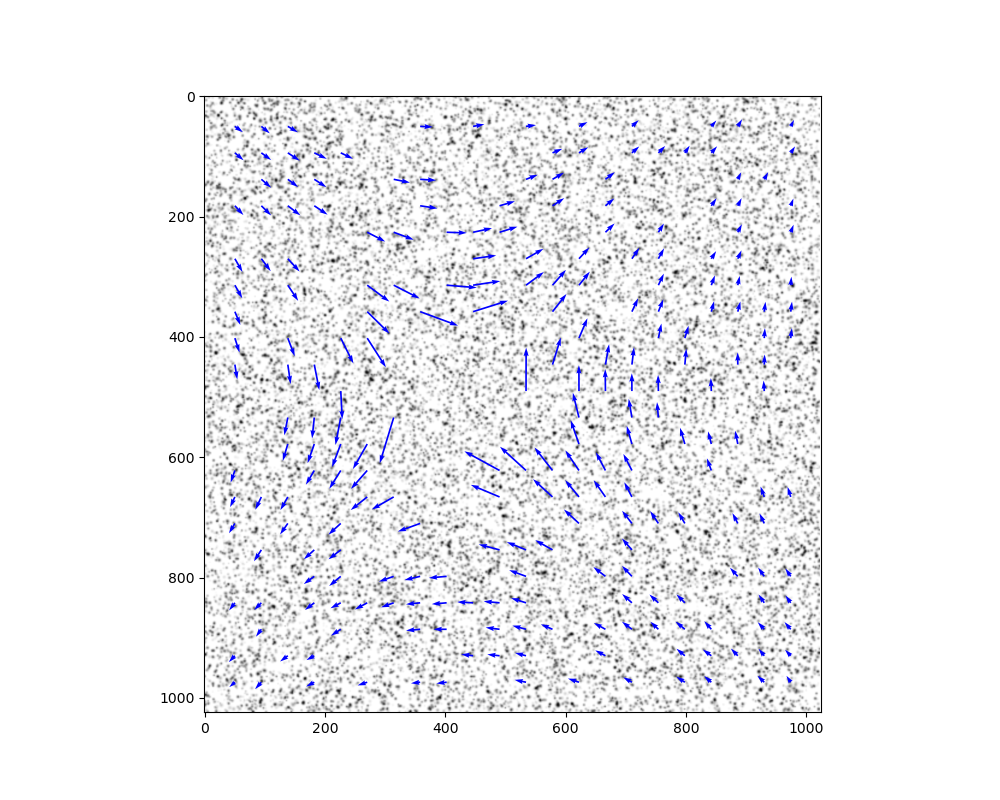
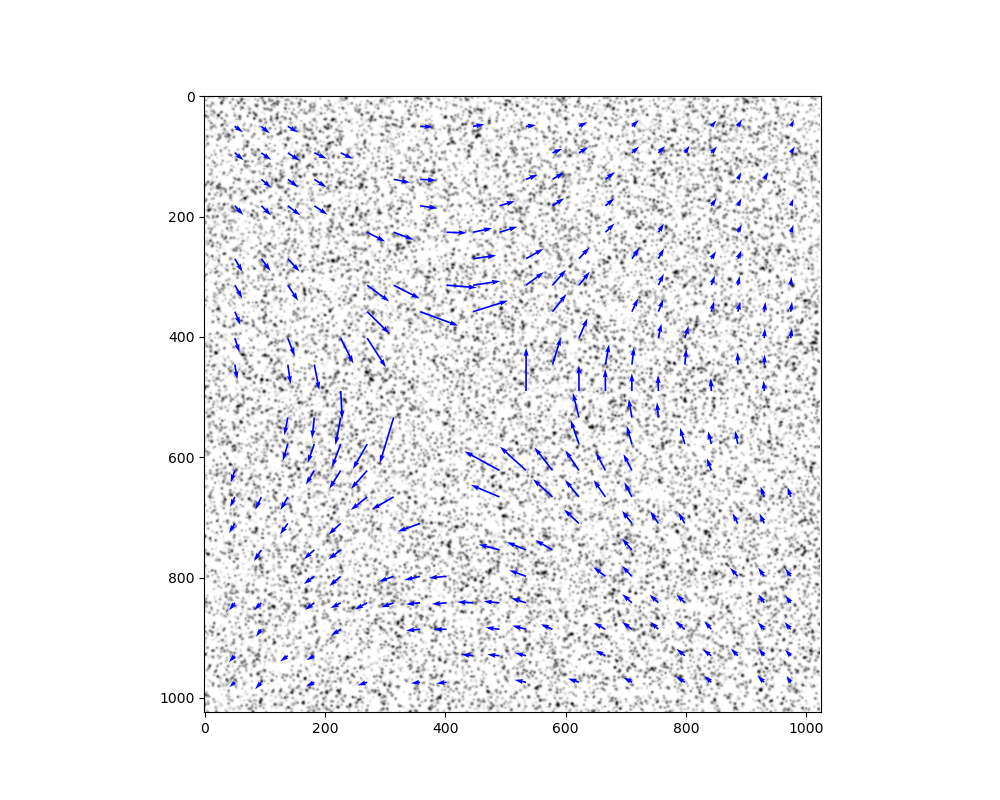
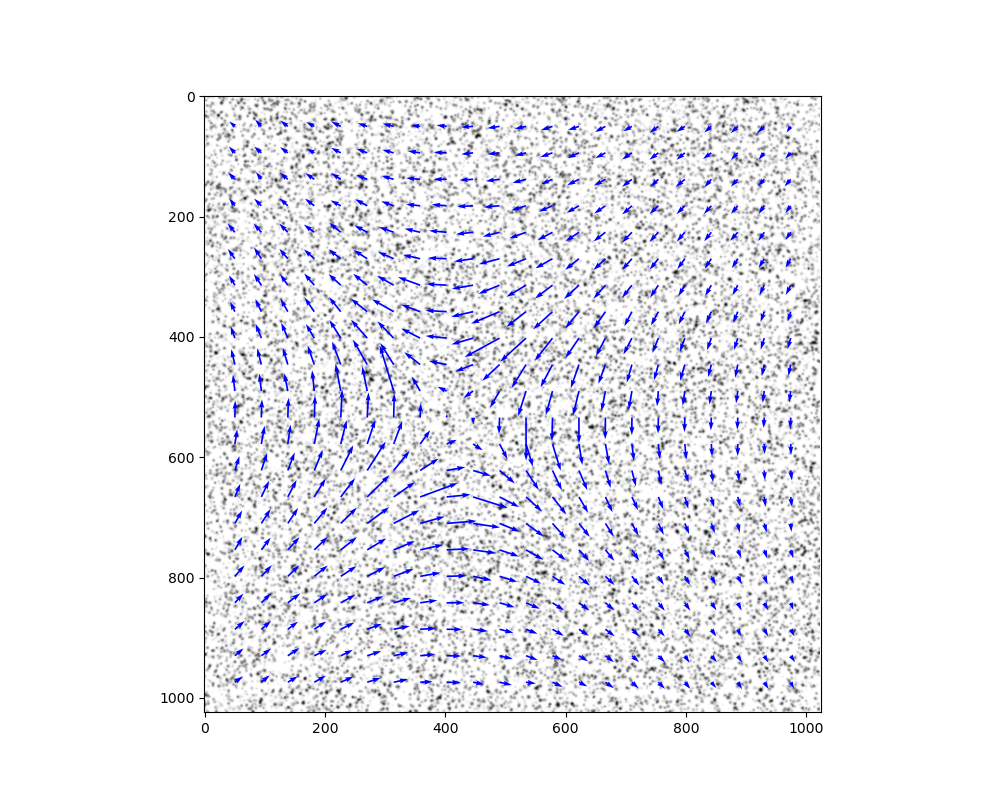
下記の画像を見ると指摘の通りx方向の反転やtools.transform_coordinatesで画像の左上基準から左下基準に座標変換が起きていた.
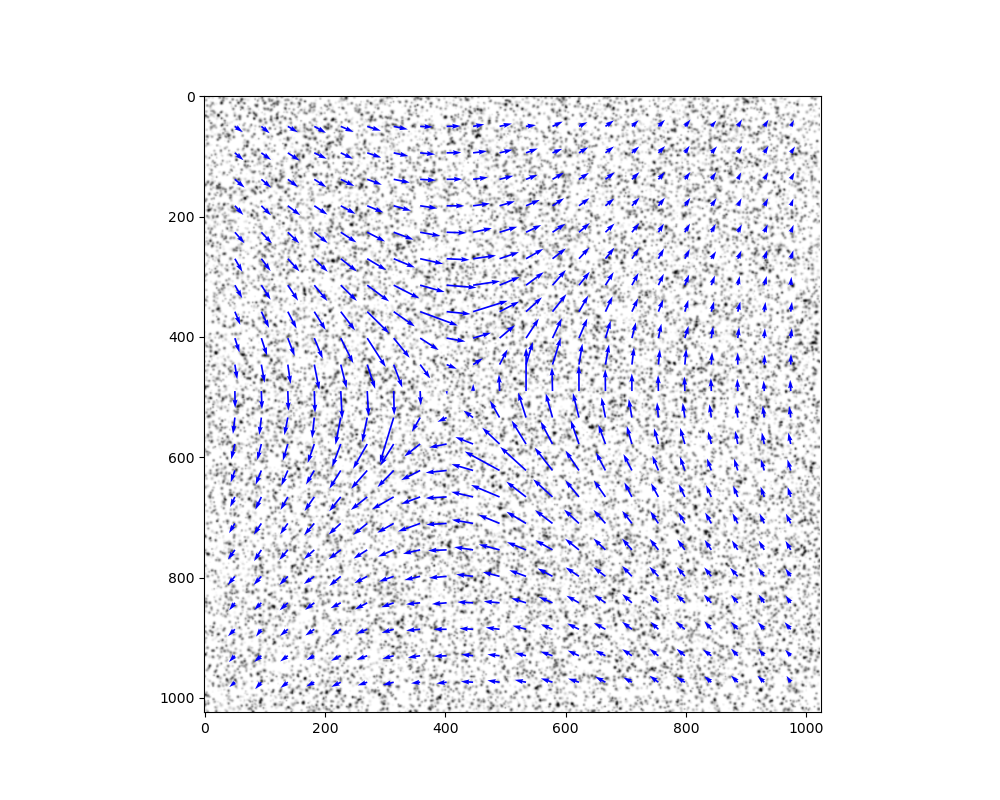
scaling.uniformに-vで入れると綺麗な速度場が表示された.
中心付近に渦が存在していたため座標変換には気づかなかった.
x, yの方向,基準座標には気をつけたい.
今回はmatplotlibのplt.quiver(x, y, u, v)で可視化していたための問題かもしれない.
@fiftystorm36様 確認いただきありがとうございます.
u0, v0, sig2noise = pyprocess.extended_search_area_piv(img_a0.astype(np.int32), img_b0.astype(np.int32), window_size=32, overlap=20, dt=1, search_area_size=64, sig2noise_method='peak2peak')
x, y = pyprocess.get_coordinates(image_size=img_a0.shape, search_area_size=64, overlap=20)
plt.quiver(x, y, u0, v0)
plt.show()
u0, v0, sig2noise = pyprocess.extended_search_area_piv(img_a0.astype(np.int32), img_b0.astype(np.int32), window_size=32, overlap=20, dt=1, search_area_size=64, sig2noise_method='peak2peak')
x, y = pyprocess.get_coordinates(image_size=img_a0.shape, search_area_size=64, overlap=20)
u1, v1, mask = validation.sig2noise_val(u0, v0, sig2noise, threshold=1.5)
plt.quiver(x, y, u1, v1)
plt.show()
u0, v0, sig2noise = pyprocess.extended_search_area_piv(img_a0.astype(np.int32), img_b0.astype(np.int32), window_size=32, overlap=20, dt=1, search_area_size=64, sig2noise_method='peak2peak')
x, y = pyprocess.get_coordinates(image_size=img_a0.shape, search_area_size=64, overlap=20)
u1, v1, mask = validation.sig2noise_val(u0, v0, sig2noise, threshold=1.5)
u2, v2 = filters.replace_outliers(u1, v1, method='localmean', max_iter=5, kernel_size=2)
plt.quiver(x, y, u2, v2)
plt.show()
u0, v0, sig2noise = pyprocess.extended_search_area_piv(img_a0.astype(np.int32), img_b0.astype(np.int32), window_size=32, overlap=20, dt=1, search_area_size=64, sig2noise_method='peak2peak')
x, y = pyprocess.get_coordinates(image_size=img_a0.shape, search_area_size=64, overlap=20)
u1, v1, mask = validation.sig2noise_val(u0, v0, sig2noise, threshold=1.5)
u2, v2 = filters.replace_outliers(u1, v1, method='localmean', max_iter=5, kernel_size=2)
x, y, u3, v3 = scaling.uniform(x, y, u2, v2, scaling_factor=1)
plt.quiver(x, y, u3, v3)
plt.show()
u0, v0, sig2noise = pyprocess.extended_search_area_piv(img_a0.astype(np.int32), img_b0.astype(np.int32), window_size=32, overlap=20, dt=1, search_area_size=64, sig2noise_method='peak2peak')
x, y = pyprocess.get_coordinates(image_size=img_a0.shape, search_area_size=64, overlap=20)
u1, v1, mask = validation.sig2noise_val(u0, v0, sig2noise, threshold=1.5)
u2, v2 = filters.replace_outliers(u1, v1, method='localmean', max_iter=5, kernel_size=2)
x, y, u3, v3 = tools.transform_coordinates(x, y, u2, v2)
plt.quiver(x, y, u3, v3)
plt.show()
u0, v0, sig2noise = pyprocess.extended_search_area_piv(img_a0.astype(np.int32), img_b0.astype(np.int32), window_size=32, overlap=20, dt=1, search_area_size=64, sig2noise_method='peak2peak')
x, y = pyprocess.get_coordinates(image_size=img_a0.shape, search_area_size=64, overlap=20)
u1, v1, mask = validation.sig2noise_val(u0, v0, sig2noise, threshold=1.5)
u2, v2 = filters.replace_outliers(u1, v1, method='localmean', max_iter=5, kernel_size=2)
x, y, u3, v3 = scaling.uniform(x, y, u2, v2, scaling_factor=1)
x, y, u3, v3 = tools.transform_coordinates(x, y, u3, v3)
plt.quiver(x, y, u3, v3)
plt.show()
u0, v0, sig2noise = pyprocess.extended_search_area_piv(img_a0.astype(np.int32), img_b0.astype(np.int32), window_size=32, overlap=20, dt=1, search_area_size=64, sig2noise_method='peak2peak')
x, y = pyprocess.get_coordinates(image_size=img_a0.shape, search_area_size=64, overlap=20)
u1, v1, mask = validation.sig2noise_val(u0, v0, sig2noise, threshold=1.5)
u2, v2 = filters.replace_outliers(u1, v1, method='localmean', max_iter=5, kernel_size=2)
x, y, u3, v3 = scaling.uniform(x, y, u2, -v2, scaling_factor=1)
plt.quiver(x, y, u3, v3)
plt.show()
終わりに
OpenPIVでPIVを実施可能か試して業務に使用可能か検討した.最終的には乱流の渦がある場所での解析を行いため,渦の画像を用いて解析した.結果としては渦を再現することはできずに対向した流れになってしまった.今後は自分でPIVのプログラムを作成してみる.
追記(2023/01/31/10:30)
反転に注意すれば問題なく使用できる気がする.
参考
PIVハンドブック,可視化情報学会
PIVとは | カトウ光研株式会社
PIV(粒子画像流速測定法)とは | 日本カノマックス株式会社