Rubyアドベントカレンダー2022の4日目の記事です。
鈴木弘一(@heronshoes)と申します。
Rubyの新しいデータフレームライブラリであるRedAmberをご紹介します。
RedAmberとは、Rubyでデータフレームを扱うためのライブラリです。
とってもざっくりいうと、Pythonにおけるpandas、Rにおけるdata.frame, dplyr/tidyrがやるようなことをRubyでできるようにするライブラリです。
2022年度Rubyアソシエーション開発助成に採択されたプロジェクトとして開発を進めています。
RedAmberのデータフレームとは
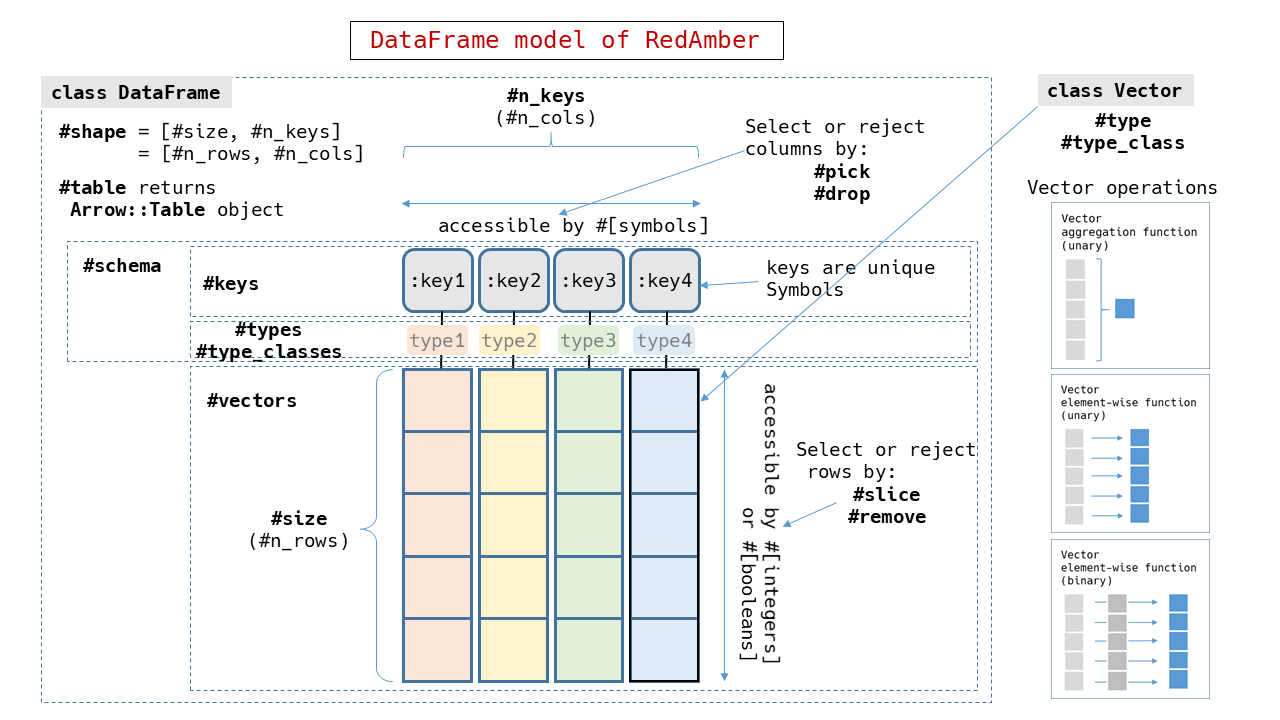
RedAmberでは次のようなものをデータフレームと決めています。
- 同じ型のデータを1次元に並べたものを、Vectorと呼ぶ。
- 同じ長さのVectorを集めてひとまとまりの2次元データとし、DataFrameと呼ぶ
- 各Vectorはユニークなラベルを付けて区別する。ラベルはキーと呼び、Symbolとする。
- 各Vectorとキーをまとめて変数と呼ぶ。変数はふつうは縦に配置されると想像しているので列とも呼ぶ。
- 各行のデータはレコードと呼び、ふつうは互いに関係した一件の観測データなどを置く。
- どんな型のデータでも欠損値として、nilを含むことができる。
Rubyでいうとユニークなラベルがあるという点でArrayを値に持つHashに似ていますが、値の長さと型の制約の点で異なっています。
また、2次元データという点では2次元のNumo::NArrayやArrayのような配列と似ていますが、ラベルと列方向の型の制約の点で異なっています。
列単位でのデータの取り回しと、行方向のデータの関連性を使った検索・集約・抽出に適したデータ構造と言えると思います。SQLのテーブルと類似したところがあります。
RedAmberのデータフレームの構造は次の絵のようになっています。
pandasのデータフレームと違うところは、行や列のインデックスがないという点です。インデックスは特別扱いせず、必要ならば通常の列の一つとして持ちます。これはPolarsの考え方と同じです。暗黙のインデックス(0...size)は常にArrayのように使えます。
RedAmberのクラスは今のところDataFrameとVector、グループ化されたデータフレームを扱うGroupしかありません。(Rubyらしく大クラス主義です)。
代わりにキーの配列はArray、キーとVectorの組はHashで返すなどなるべくRuby標準のコレクションクラスを使うようにしています。そうすることによってよく知ってるRuby標準のやり方で柔軟に処理が書けるはずだからです。
RedAmberはインメモリのカラムナーフォーマットを特徴とするApache ArrowのRuby実装であるRed Arrowを使っています。従ってDataFrame#tableメソッドは内部で使っているArrowのTableを返します。同様にVectorの中身は、ArrowのArrayです。
RedAmberはRed Arrowの一部を使いやすくラップするデータフレームラッパーであるとも言えます。
Apache Arrowを利用したデータ処理については、Rubykaigi-2022の須藤さんのスライドを貼っておきます。RedAmberのことも紹介して頂いています!
入出力
Apache Arrowは特に入出力が速い上に使えるフォーマットの種類も充実しています。csvファイルの読み込みで、Arrowを使うRedAmberと、Ruby標準のCSVを使うRoverを比較してみます。
prelude: |
require 'rover'
require 'red_amber'
penguins_csv = 'tmp/penguins.csv'
unless File.exist?(penguins_csv)
require 'datasets-arrow'
ds = Datasets::Penguins.new
RedAmber::DataFrame.new(ds).save(penguins_csv)
end
benchmark:
'penguins by Rover': Rover.read_csv(penguins_csv)
'penguins by RedAmber': RedAmber::DataFrame.load(penguins_csv)
上のようなyamlファイルでbenchmark-driverを使って比較します。
benchmark-driver csv_load_penguins.yml
Warming up --------------------------------------
penguins by Rover 49.494 i/s - 54.000 times in 1.091036s (20.20ms/i)
penguins by RedAmber 616.594 i/s - 620.000 times in 1.005524s (1.62ms/i)
Calculating -------------------------------------
penguins by Rover 38.195 i/s - 148.000 times in 3.874902s (26.18ms/i)
penguins by RedAmber 574.486 i/s - 1.849k times in 3.218528s (1.74ms/i)
Comparison:
penguins by RedAmber: 574.5 i/s
penguins by Rover: 38.2 i/s - 15.04x slower
念のため言っておくと、Roverでも一旦読み込みさえすればデータフレームの操作はNumo::NArrayを利用しているので十分に速いです。ただ、データが巨大になる程Apache Arrowの優位性が生きてくると考えています。
RedAmberによるデータ分析の例
Rのデータセットから、英国のCOVID-19のデータを元にした Simpson's Paradox: Covidを使ってRedAmberで簡単なデータ分析をやってみます。
Red Data ToolsのRed Datasetsではデータ分析や機械学習で使うための様々なデータが集められていて、Rubyから利用することができます。
require 'red_amber'
require 'datasets-arrow'
ds = Datasets::Rdatasets.new('openintro', 'simpsons_paradox_covid')
df = RedAmber::DataFrame.new(ds)
#<RedAmber::DataFrame : 268166 x 3 Vectors, 0x000000000000f0dc>
age_group vaccine_status outcome
<string> <string> <string>
0 under 50 vaccinated death
1 under 50 vaccinated death
2 under 50 vaccinated death
3 under 50 vaccinated death
4 under 50 vaccinated death
: : : :
268163 50 + unvaccinated survived
268164 50 + unvaccinated survived
268165 50 + unvaccinated survived
データフレームは途中を省略して表示します(irbでは9行まで、Jupyter notebook(iruby)では最大8行まで表示)。次のようにすると一部を取り出して表示できます。
df.head(8) # 先頭の8行を表示
df.tail # 末尾の5行を表示
df[...4, -4..] # 先頭の4行、末尾の4行を表示(RangeのArrayが使える)
# (出力結果省略)
また、@kojix2さん作のred-amber-viewを使うと別ウィンドウで全体の表示ができます。
require 'red_amber/view'
df.view
データフレームの詳細情報(列の型とかキーとかユニークなデータ数の一覧)は、次のようにすると表示できます。
df.tdr
RedAmber::DataFrame : 268166 x 3 Vectors
Vectors : 3 strings
# key type level data_preview
0 :age_group string 2 {"under 50"=>237419, "50 +"=>30747}
1 :vaccine_status string 2 {"vaccinated"=>117114, "unvaccinated"=>151052}
2 :outcome string 2 {"death"=>734, "survived"=>267432}
また、数値データの場合は統計的要約はつぎのようにします。
df.summary
今回は文字列データしかないので結果は空です。
#<RedAmber::DataFrame : 0 x 9 Vectors, 0x000000000000f0f0>
variables count mean std min 25% median 75% max
(Empty Vectors)
データフレームの準備ができたところで、ワクチン接種の有無と生存/死亡で2x2のグループ化をしてそれぞれのデータ数で集約してみましょう。
count = df.group(:vaccine_status, :outcome).count
#<RedAmber::DataFrame : 4 x 3 Vectors, 0x000000000000f104>
vaccine_status outcome count
<string> <string> <int64>
0 vaccinated death 481
1 unvaccinated death 253
2 vaccinated survived 116633
3 unvaccinated survived 150799
列に接種状態、行に生存状態を配置した集計表に変形します。
all_count = count.to_wide(name: :vaccine_status, value: :count)
#<RedAmber::DataFrame : 2 x 3 Vectors, 0x000000000000f118>
outcome vaccinated unvaccinated
<string> <uint32> <uint32>
0 death 481 253
1 survived 116633 150799
ちなみにRedAmberには列ラベルは存在しません。この表を転置する際に、列名を列に変換した際にラベルとして与えることができます。
all_count.transpose(name: :vaccine_status)
# 単に transposeとするとデフォルトの列名(:NAME)が使われる
#<RedAmber::DataFrame : 2 x 3 Vectors, 0x000000000000f12c>
vaccine_status death survived
<string> <uint16> <uint32>
0 vaccinated 481 116633
1 unvaccinated 253 150799
次にそれぞれの値を比率に変換します。
all_count.assign do
{
"vaccinated_%": 100.0 * vaccinated / vaccinated.sum,
"unvaccinated_%": 100.0 * unvaccinated / unvaccinated.sum
}
end
# ブロックの中にある{}は、ハッシュ生成の{}です。
#<RedAmber::DataFrame : 2 x 5 Vectors, 0x000000000000f140>
outcome vaccinated unvaccinated vaccinated_% unvaccinated_%
<string> <uint32> <uint32> <double> <double>
0 death 481 253 0.41 0.17
1 survived 116633 150799 99.59 99.83
このように全体のデータで集計すると、ワクチン接種あり(vaccinated)の方が死亡率(death)が高いことになってしまっていますね。
次に、同じことを年齢のグループ(50歳より上か下か)で分けて集計してみましょう。
今までやったことを並べてメソッドにしておいて、繰り返し使えるようにします。
def make_covid_table(df)
df.group(:vaccine_status, :outcome)
.count
.to_wide(name: :vaccine_status, value: :count)
# assignの中身はこんな風にも書けます。
.assign('vaccinated_%', 'unvaccinated_%') do
[
(100.0 * vaccinated / vaccinated.sum).round(n_digits: 3),
(100.0 * unvaccinated / unvaccinated.sum).round(n_digits: 3)
]
end
end
df[:age_group] == "under 50"のようにすると、条件を満たす行の値がtrueであるようなフィルターがVectorとして得られるので、次のようにすると年齢条件で抽出されたデータフレームが得られます。
df[df[:age_group] == "under 50"]
#<RedAmber::DataFrame : 237419 x 3 Vectors, 0x000000000000f168>
age_group vaccine_status outcome
<string> <string> <string>
0 under 50 vaccinated death
1 under 50 vaccinated death
2 under 50 vaccinated death
3 under 50 vaccinated death
4 under 50 vaccinated death
: : : :
237416 under 50 unvaccinated survived
237417 under 50 unvaccinated survived
237418 under 50 unvaccinated survived
50歳未満では、
make_covid_table(df[df[:age_group] == "under 50"])
#<RedAmber::DataFrame : 2 x 5 Vectors, 0x000000000000f154>
outcome vaccinated unvaccinated vaccinated_% unvaccinated_%
<string> <uint32> <uint32> <double> <double>
0 death 21 48 0.02 0.03
1 survived 89786 147564 99.98 99.97
50歳以上では、
make_covid_table(df[df[:age_group] == "50 +"])
#<RedAmber::DataFrame : 2 x 5 Vectors, 0x000000000000f17c>
outcome vaccinated unvaccinated vaccinated_% unvaccinated_%
<string> <uint16> <uint16> <double> <double>
0 death 460 205 1.69 5.96
1 survived 26847 3235 98.32 94.04
年齢別に分けた集計結果では、直観の通り接種ありの死亡率の方が低くなっていることが確認できます。これはシンプソンのパラドックスと呼ばれる現象で、原因と関連して結果に影響を及ぼすような因子(交絡因子=今回の場合は年齢)があるために因果関係を間違って認識することから生じています。
今回の場合は高齢者の方が死亡リスクが高く、また接種率も高いことが関係しています。
このことを確認するために、年齢別の接種の有無、および死亡数を求めてみると下記のようになります。これは改めて比率に直す必要もないですね。
df.group(:vaccine_status, :age_group).count.to_wide(name: :age_group, value: :count)
#<RedAmber::DataFrame : 2 x 3 Vectors, 0x000000000000f190>
vaccine_status under 50 50 +
<string> <uint32> <uint16>
0 vaccinated 89807 27307
1 unvaccinated 147612 3440
df.group(:outcome, :age_group).count.to_wide(name: :age_group, value: :count)
#<RedAmber::DataFrame : 2 x 3 Vectors, 0x000000000000f1a4>
outcome under 50 50 +
<string> <uint32> <uint16>
0 death 69 665
1 survived 237350 30082
いかがでしたでしょうか。Rの例に比べてRubyらしい書き方でデータの分析ができているのではないかと思います。
RedAmberはまだまだ開発中のライブラリですが、基本操作と重要な機能(Group、Reshape、Join)は使えるようになっていますので、興味がある方は試してみていただけると嬉しいです。インストール方法などはGitHubのRedAmberページで最新情報を参照してください。
今、コード全体の大掃除を行っていて、大掃除なので22年内に終わらせる予定ですが、これによってコードの見通しが良くなることとパフォーマンスが上がることを目指しています。Ruby3.2に合わせてバージョン0.3.0をリリースする予定です。
今はほぼ私一人で作っているのですが、コードはもちろん、使ってみた感想やこんな機能があったらいいなといった提案は大歓迎です。
私のスキル不足の部分で、
- YARDドキュメントを手伝ってくださる方 (現状進捗率30%くらい)
- rbs(型定義情報)を手伝ってくださる方 (今は何もしていない)
- RedAmberのロゴを一緒に考えてくださる方
は特に募集しています。
GitHubで何でも書ける場としてDiscussionsを使えるようにしていますので使ってみてください(日本語オンリーでもOKです)。
RedAmberはRed Data Toolsの成果に助けられています。RedAmberを使っていくことでApache Arrowの開発にも取り組みたいと思ってくださる方が増えるといいなと思っています。興味がある方は Red Data ToolsのGitterにお越しください。
私はつい最近までOSSの開発の初心者で完全な使う側の人間でしたのでまだgitすら使ったことがないというレベルの人に対しても参加のきっかけとなるような活動をしていきたいと思っています。
noteにも別の紹介記事を書いているのでよかったらご覧ください。
今年のRubyアドベントカレンダーでは12/18の枠も予約しています。そちらではRedAmberでやろうとしていることを色々お話ししたいと思います。お楽しみに。