TL;DR
- SageMaker のビルトインアルゴリズム image classification (ResNet-152) を使って COVID-19 陽性・陰性の肺CT画像を分類
- 50エポックほど回すと学習データに対する精度は99%に、バリデーションデータに対する精度は91%に
- ソースコードは github にあげています
データセット
CT画像は肺1つにつき何枚もスライスして撮影するので、高さ・幅・スライスの3次元画像になります。ただ3次元画像をいきなり扱うのはちょっと・・・と思ったので、2次元画像のデータセットを利用しました。医療画像を探すのは初めてだったので、探すのに少し苦労しました。見つけたデータセットのうち、すぐに使えそうなものを以下にメモしておきます。
3次元のCT画像のデータセット
MosMedDataはkeras のチュートリアル にも利用されている3次元CT画像のデータセットです。ライセンスは Creative Commons Attribution-NonCommercial-NoDerivs 3.0 Unported (CC BY-NC-ND 3.0) です。
MosMedData: Chest CT Scans with COVID-19 Related Findings
Sergey Morozov, Anna Andreychenko, Nikolay Pavlov, Anton Vladzymyrskyy, Natalya Ledikhova, Victor Gombolevskiy, Ivan Blokhin, Pavel Gelezhe, Anna Gonchar, Valeria Chernina, Vladimir Babkin
medRxiv 2020.05.20.20100362; doi: https://doi.org/10.1101/2020.05.20.20100362
MosMedDataでは、DOWNLOADからデータセットへのリンク先を取得すると、masks と studies のフォルダが見つかると思います。studies のフォルダには、CT-0, CT-1, CT-2, CT-3, CT-4 のフォルダがあり、それぞれにCT画像が入っています。CT-0は正常データ、CT-1から CT-4 にかけて症状が強く現れているデータセットのようです。ファイル形式は NifTI フォーマットで、nii という拡張子がついています。Python でファイルを読むためには、nibabel という Python のライブラリが必要です。
keras のチュートリアルでは、CT-2とCT-3を異常データとして扱っており、3次元データに対して3D Convolutionを適用しています。チュートリアルの作者が Github 上でデータを再配布しています (リンク)。
2次元のCT画像のデータセット
Mendeley Data 上に2次元の肺CT画像のデータセットが公開されています。ライセンスは Creative Commons Attribution 4.0 International (CC BY 4.0) です。
El-Shafai, Walid; Abd El-Samie, Fathi (2020), “Extensive COVID-19 X-Ray and CT Chest Images Dataset”, Mendeley Data, V3, doi: 10.17632/8h65ywd2jr.3
全体のサイズは4GBほどあり、陽性と陰性をあわせて8000枚ほどの2次元CT画像があります。CT画像以外にX線の画像も含まれています。こちらのファイル形式は jpg または png であり、PIL などのPython のライブラリで読み込むことが可能です。画像はグレースケールとなりますので、RGBの3チャネルではなく1チャネルとなります。
画像分類の実装
Mendeley Data の2次元CT画像を分類するために、Amazon SageMaker のビルトインアルゴリズム Image Classification を利用しました。アルゴリズムは 152層のResNetです。実装の詳細はgithub を見てください。
ビルトインアルゴリズム Image Classification を利用して学習する際、ファイルモードとパイプモードが使えます。ファイルモードは学習用インスタンスにすべての学習データをダウンロードしてから学習を開始し、パイプモードは学習データを逐次ダウンロードして学習を行います。そのためパイプモードは、すべての学習データのダウンロードを待つ必要がなく、すぐに学習を始めることができます。特にデータセットが大規模な場合に有効です。
今回のデータセットは4GBとやや大きかったのでパイプモードを利用しました。パイプモードを利用する場合、データを RecordIO 形式にするか拡張マニフェストファイルを用意するかしなければなりません。今回は拡張マニフェストファイルを利用しました。以下のように、S3の画像データとラベル情報を紐付けたものが、拡張マニフェストファイルです。すでに S3 にデータがあれば、それをそのまま使えるので便利です。
{"source-ref":"s3://image/filename1.jpg", "class":"0"}
{"source-ref":"s3://image/filename2.jpg", "class":"1"}
上記の拡張マニフェストファイルの各行(ファイルのパスとクラス)はシャッフルが必要です。 最初、拡張マニフェストファイルの前半をクラス 0、後半をクラス 1で固めたファイルを作っていたのですが、それだと精度が全くあがりませんでした。
RecordIOは画像データとラベルをバイナリ形式に変換したものなので変換処理が必要になります。しかし、効率的なバイナリ形式であるため、学習時のデータのI/Oの向上が期待できます。
結果
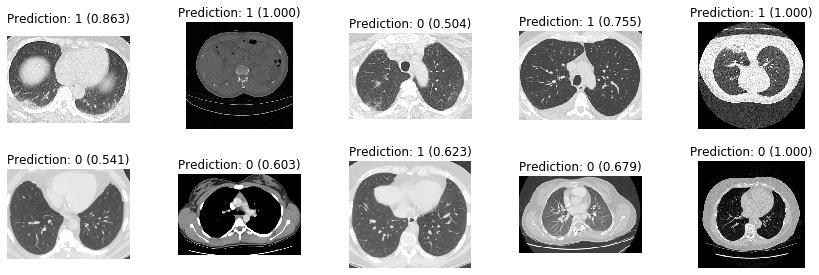
10エポックほど回してテストデータを分類した結果は以下のとおりです。上段はCOVID-19が陽性(ラベル1)、下段は陰性(ラベル0) です。画像の上に推定結果と確率を示しています。素人目には何が何だか分からないですが、予測が結構あたっています。
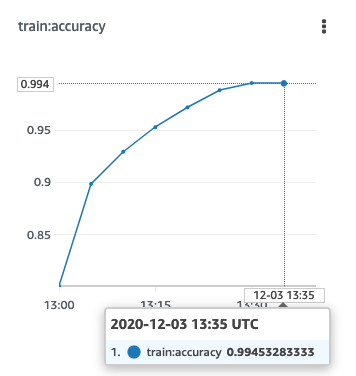
50エポックほどまわしたときの、学習データに対する精度とバリデーションデータに対する精度の推移を示します。ビルトインアルゴリズムの場合、こうした基本的なメトリクスをSageMaker が自動で取得して、コンソールに表示してくれます。
| 学習データに対する精度 | バリデーションデータデータに対する精度 |
|---|---|
 |
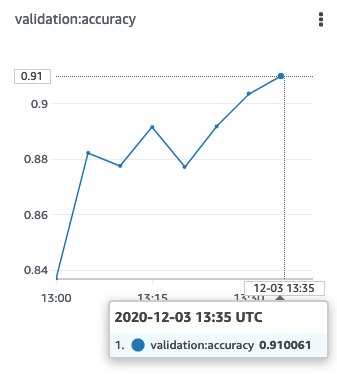 |
なんと学習データに対する精度は99.4%です!バリデーションデータに対する精度も91%となかなかいいですね。今回全くハイパーパラメータなどをいじっていないので、改良の余地はあるかもしれません。
もし時間があれば、3次元画像のほうもやってみたいと思います。ではでは。