to_graphvizメソッド
xgboostにはto_graphvizのメソッドによって作成された木を可視化することができる。
しかしそのグラフのノードには変数名でなく、"f0"や"f1"のように表示されてしまい、これらを照らし合わせながら分析するのは少々煩わしい。
plot_importanceには変数名をkey、そのfeature_importanceをvalueにもつ辞書を渡せば"f1"などと表示されてしまう問題は解決できたのだが、どうやらto_graphvizにはないらしい。
そこでXgboost.poltting.pyの中身をのぞいて"f1"などを変数の名前に置き換えられる関数を作成した。
@DaigoKimuraさんの指摘を受けて修正しました。(2018.12.14)
ありがとうございます!
import re
from graphviz import Digraph
_NODEPAT = re.compile(r'(\d+):\[(.+)\]')
_LEAFPAT = re.compile(r'(\d+):(leaf=.+)')
_EDGEPAT = re.compile(r'yes=(\d+),no=(\d+),missing=(\d+)')
_EDGEPAT2 = re.compile(r'yes=(\d+),no=(\d+)')
def _parse_node(graph, text):
match = _NODEPAT.match(text)
if match is not None:
node = match.group(1)
graph.node(node, label=match.group(2), shape='circle')
return node
match = _LEAFPAT.match(text)
if match is not None:
node = match.group(1)
graph.node(node, label=match.group(2), shape='box')
return node
raise ValueError('Unable to parse node: {0}'.format(text))
def _parse_edge(graph, node, text, yes_color='#0000FF', no_color='#FF0000'):
"""parse dumped edge"""
try:
match = _EDGEPAT.match(text)
if match is not None:
yes, no, missing = match.groups()
if yes == missing:
graph.edge(node, yes, label='yes, missing', color=yes_color)
graph.edge(node, no, label='no', color=no_color)
else:
graph.edge(node, yes, label='yes', color=yes_color)
graph.edge(node, no, label='no, missing', color=no_color)
return
except ValueError:
pass
match = _EDGEPAT2.match(text)
if match is not None:
yes, no = match.groups()
graph.edge(node, yes, label='yes', color=yes_color)
graph.edge(node, no, label='no', color=no_color)
return
raise ValueError('Unable to parse edge: {0}'.format(text))
def to_graphviz(XGB,X_cols,num_trees=0):
tree = XGB._Booster.get_dump()[num_trees]
yes_color = "red"
no_color = "blue"
for i,col in enumerate(X_cols):
tree = tree.replace('f{0}'.format(i),col)
tree = tree.split()
graph = Digraph()
for i, text in enumerate(tree):
if text[0].isdigit():
node = _parse_node(graph, text)
else:
if i == 0:
# 1st string must be node
raise ValueError('Unable to parse given string as tree')
_parse_edge(graph, node, text, yes_color=yes_color,no_color=no_color)
return graph
def plot_tree(XGB,X_cols,num_trees=0,ax=None):
if ax is None:
_,ax = plt.subplots(figsize=(25, 25))
g = to_graphviz(XGB,X_cols,num_trees=num_trees)
s = BytesIO()
s.write(g.pipe(format='png'))
s.seek(0)
img = image.imread(s)
ax.imshow(img)
ax.axis('off')
return ax
xgboost.plottingの必要なところをコピペしてきて、to_graphvizとplot_treeの内容を少々変更。
僕はプロットさえできれば十分なので、argsは省略して色は青と赤に設定。
*変数名に空白があると動かないので注意
tree_plot.plot_treeの引数XGBはxgboost.XGBClassifierもしくはXGBRegressorオブジェクト。
X_colsは変数名の入ったリストで、num_treesは本家と同様、何番目の木をプロットするか。
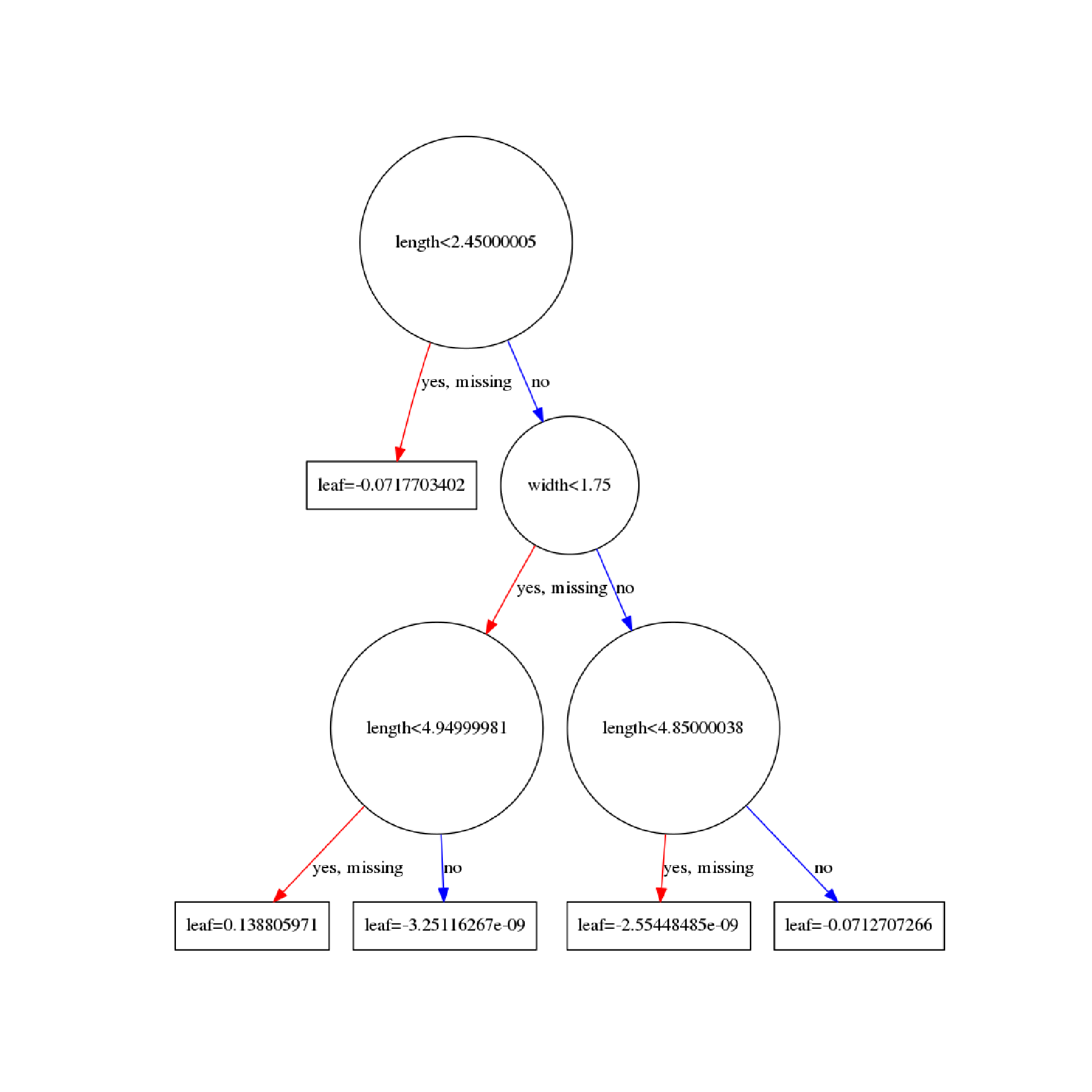
以下のように実行するとちゃんとノードの中に変数名が表示される。
import xgboost as xgb
import matplotlib.pyplot as plt
import sklearn.datasets as sk_data
import tree_plot
%matplotlib inline
X,y,X_cols = sk_data.load_iris()["data"],sk_data.load_iris()["target"],sk_data.load_iris()["feature_names"]
# str.split()を用いるため変数名に空白があるとエラーが出る
X_cols = [i[6:-5] for i in X_cols]
XGB = xgb.XGBClassifier()
XGB.fit(X,y)
tree_plot.plot_tree(XGB,X_cols,num_trees=1)
plt.savefig("plot_tree.png",format="png")
plt.close()