はじめに
ベイジアンネットワークは、データの因果関係を分析する手法の一つで、ある事象の発生が他の事象に及ぼす影響を条件付き確率として評価し、グラフィカルに表現します。変数の依存関係やネットワークの骨格を推論する手法はいくつかありますが、手法の選択に迷うことがあります。
そこで今回は、pgmpyで実装されている複数の手法について比較してみようと思います。
データの準備
今回はWineデータセットを用いて評価を行います。
import pandas as pd
from sklearn.datasets import load_wine
from sklearn.preprocessing import StandardScaler
# Load dataset
wine = load_wine()
df = pd.DataFrame(wine.data, columns=wine.feature_names) # Explanatory variable
df['Wine Class'] = wine.target # Objective variable
# Normalization
scaler = StandardScaler()
df_scaled = scaler.fit_transform(df)
df =pd.DataFrame(df_scaled,columns=df.columns)
今回のデータセットは、「アルコール度数/色の濃さ」などの13種の特徴量に基づき、ワインの品種分類タスク(3クラス)を想定した表形式データセットです。
各特徴量の相関は以下の通りです。
import seaborn as sns
import matplotlib.pyplot as plt
# Feature correlation
corr_matrix = df.corr()
sns.heatmap(corr_matrix,
square=True,
cmap='coolwarm',
xticklabels=corr_matrix.columns.values,
yticklabels=corr_matrix.columns.values)
plt.show()
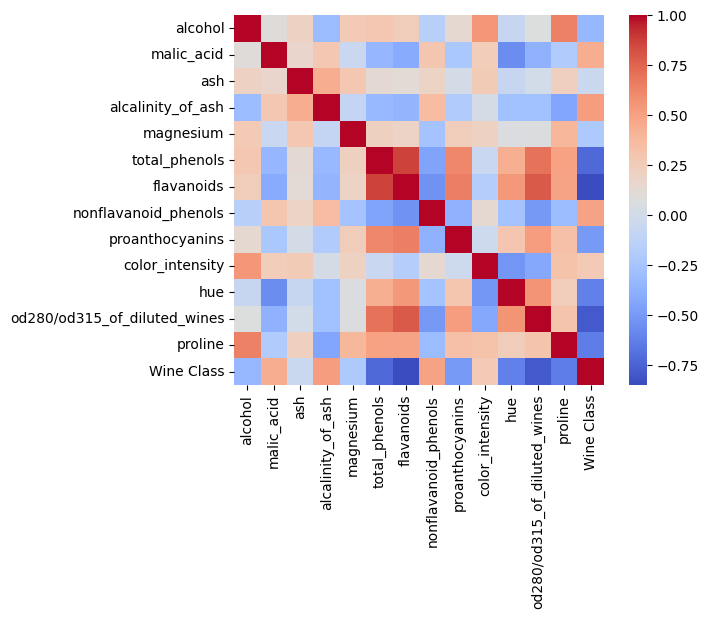
フェノール化合物の一種であるflavanoids(フラボノイド)とtotal_phenols(総フェノール量)が強く相関しているのは妥当そうですね。
さて、今回はアルゴリズムや実行時間の問題からビン分割とよばれる特徴量の離散化を施しています。ビン分割の粒度と推定値の関係は少し気になりますが本記事では深堀しません。
# %% Binning
df_new = df.copy()
for col in wine.feature_names:
df_new[col] = pd.cut(df_new[col], 5, labels=False)
各種手法
今回はpgmpyに実装されている以下の手法を試してみました。networkxを用いてgmlファイルを生成し、Cytoscapeを用いて推定されたネットワークを描画しています。
| 項番 | ページ内リンク |
|---|---|
| 1 | PC (Constraint Based Estimator) |
| 2 | Hill Climb Search (BicScore) |
| 3 | Hill Climb Search (K2Score) |
| 4 | Tree Search |
| 5 | Exhaustive Search |
| 6 | Mmhc Estimator |
1. PC (Constraint Based Estimator)
import networkx as nx
from pgmpy.estimators import PC
network = PC(df_new)
best_network = network.estimate()
# build network
edge_list = list(best_network.edges())
g = nx.DiGraph()
for e in edge_list:
src = e[0]
dst = e[1]
w = abs_adj[src][dst]
g.add_edge(src,dst,weight=w)
nx.write_gml(g,'/save/directory/to/the/obtained/network.gml')
かなり疎なネットワークになりました。情報量は限定的ですが、「ワイン品種→色の濃さ」といった出力は面白いです。右二つの関係性も妥当そうです。
2. Hill Climb Search (BicScore)
from pgmpy.estimators import HillClimbSearch, BicScore, K2Score
network = HillClimbSearch(df_new)
best_network = network.estimate(scoring_method=BicScore(df_new))
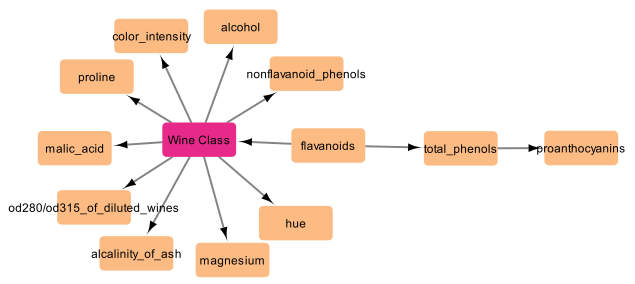
ワインの品種を中心としたネットワークが取得できました。フラボノイドが重要な役割を果たしているようですね。
3. Hill Climb Search (K2Score)
from pgmpy.estimators import HillClimbSearch, BicScore, K2Score
network = HillClimbSearch(df_new)
best_network = network.estimate(scoring_method=K2Score(df_new))
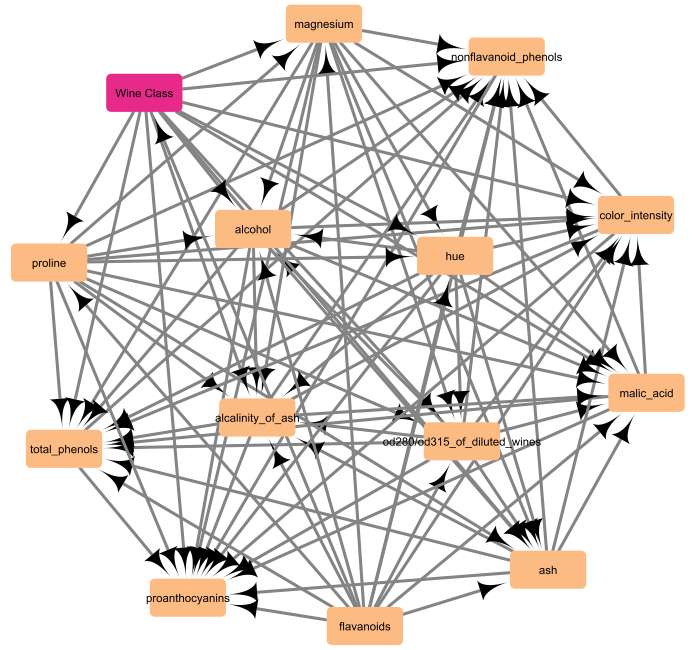
今度はかなり密なネットワークが推定されました。フラボノイドが一貫して上流に位置しているのが興味深いです。一方で、エッジが多くてどこに着目したら良いか分かりづらいです。相関の重みで枝切りしても良いかもしれません。
4. Tree Search
from pgmpy.estimators import TreeSearch
network = TreeSearch(df_new)
best_network = network.estimate()
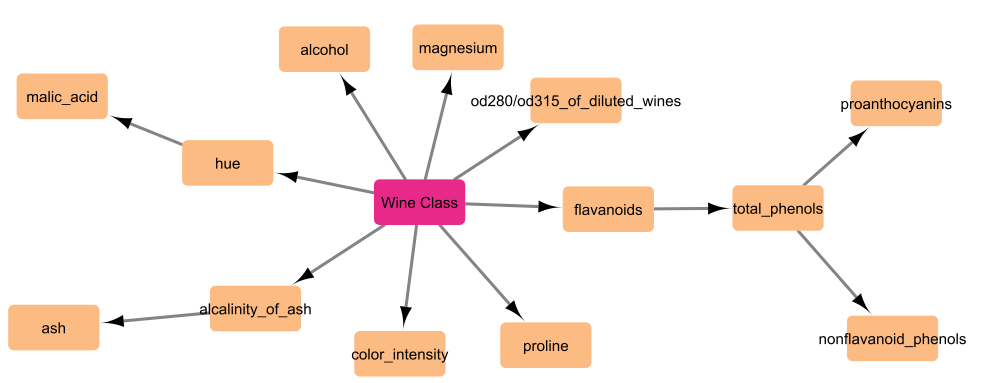
ワインの品種を中心としたネットワークが取得できました。「総フェノール量」周辺の関係性も妥当に思えます。解釈性は結構高そうですね。
5. Exhaustive Search
from pgmpy.estimators import ExhaustiveSearch
network = ExhaustiveSearch(df_new)
best_network = network.estimate()
全探索するため、ノード数が6以上の場合は現実的な時間で推定ができないようです。
6. Mmhc Estimator
from pgmpy.estimators import MmhcEstimator
network = MmhcEstimator(df)
best_network = network.estimate()
空のネットワークが出力され、うまく実行できませんでした。
実行時間の比較
各手法について、骨格推定に要した時間についても比較してみました。
| PC | HC (BicScore) | HC (K2Score) | Tree | Exhaustive | Mmhc |
|---|---|---|---|---|---|
| 18.4 sec | 7.0 sec | 13.5 sec | 0.70 sec | - sec | - sec |
Tree Searchを用いた手法がかなり高速であったことが分かります。今回は特徴量数が14の比較的小さなネットワークの推論だったので、PCやHill Climb Search (K2Score)でも現実的な時間で実行できています。60特徴量の別データに供したところ、Hill Climb Searchでも1日以上推論にかかったので注意してください。
おわりに
今回はワインのデータセットを用いてpgmpyで実装されている各種ベイジアンネットワークの骨格推論を比較しました。今回のデータセットについては、Hill Climb Search (BicScore)とTree Searchの結果が解釈しやすく感じました。コードはGitHubで公開しています。
ワインの味の違いなど自分は何も分かりませんが、「フラボノイドが○○」とか言って、分かっている感を醸し出していこうと思いました。
References