はじめに
ニューラルネットを用いた画像関係の処理に、セグメンテーションと呼ばれるタスクが存在します。セグメンテーションは画像の1ピクセルごとにどのクラスに属するか予測します。代表的な応用事例として、自動運転・医療画像解析が挙げられます。
バイオ関係では、接着細胞の割合を算出することが可能だと思います。
今回はPytorchの習熟とセグメンテーションに対する理解を深めることを目的として、UNetの実装を行いました。
UNet
【参考】セグメンテーションのモデル
【原著論文】U-Net: Convolutional Networks for Biomedical Image Segmentation
セグメンテーションにはいくつかのモデルが存在しますが、UNetはエンコーダ・デコーダ構造の代表的なモデルとなります。
このモデルは序盤(エンコーダ)に畳み込み層を用いた特徴量抽出を行い、終盤(デコーダ)で確率マップを出力します。
UNetの特徴は、skip connectionでデコーダとエンコーダの出力をチャンネル方向に結合することです(下図の灰色矢印)。これにより物体の位置情報を保持して高精度の予測を可能としています。

(原著論文より)
データセット
データセットとしてKaggleの「UW-Madison GI Tract Image Segmentation」のデータセットを使用しました。本コンペティションはMRIの画像から小腸・大腸・胃の領域を予測し、その精度を競うものです。
環境
- Google Colaboratory Pro
コード
モジュールのimport
import pandas as pd
import cv2
from PIL import Image
from matplotlib import pyplot as plt
import numpy as np
from torch.utils.data import DataLoader
from torch.utils.data import Dataset as BaseDataset
import torch
import torch.nn as nn
import torch.nn.functional as F
import torch.optim as optim
from torchvision import transforms
from torchvision.transforms import functional
!pip install -U segmentation-models-pytorch
import segmentation_models_pytorch as smp
データのダウンロード・前処理は省略します。データセットのtrain.csvにラベル情報が含まれており、これをpngファイルとして保存します。
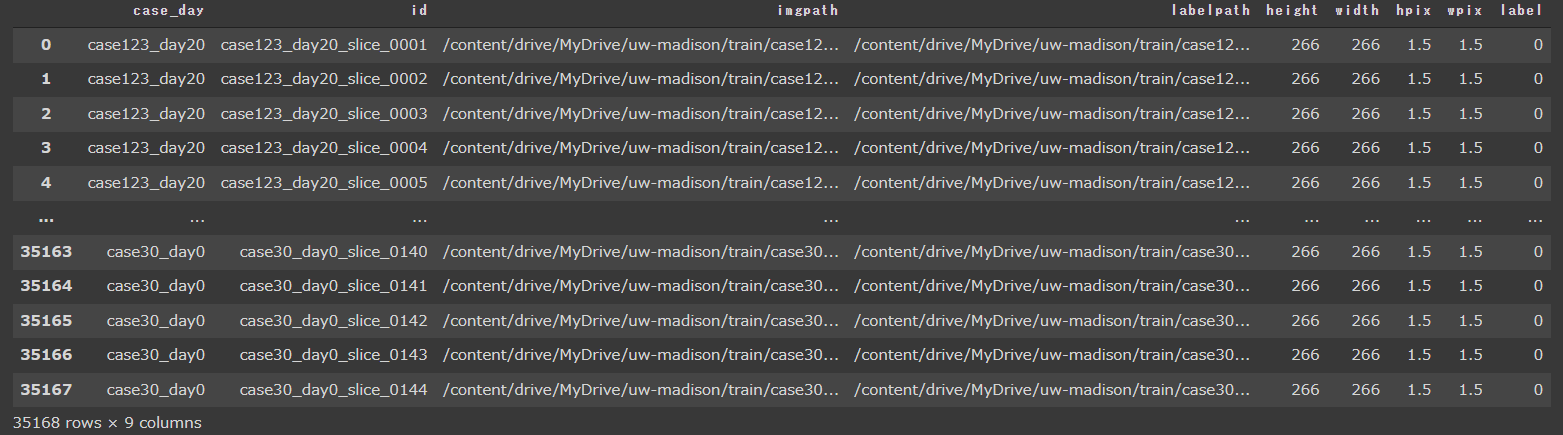
学習用の画像のパスとラベルのpngファイルのパスが分かるように以下のようなデータフレームを作成しました。
train_df = pd.read_csv("/content/drive/MyDrive/uw-madison/train_df.csv")
val_df = pd.read_csv("/content/drive/MyDrive/uw-madison/val_df.csv")
test_df = pd.read_csv("/content/drive/MyDrive/uw-madison/test_df.csv")
train_df
本データセットはラベル有りと無しがほぼ1:1になっています。全てのデータを使用するとうまく学習が進まなかっため、ラベルありのデータのみで学習を行いました。
train_df = train_df[train_df.label == 1].reset_index(drop=True)
val_df = val_df[val_df.label == 1].reset_index(drop=True)
test_df = test_df[test_df.label == 1].reset_index(drop=True)
データローダーを作成します
class Dataset(BaseDataset):
def __init__(
self,
df,
transform = None,
classes = None,
augmentation = None
):
self.imgpath_list = df.imgpath
self.labelpath_list = df.labelpath
def __getitem__(self, i):
imgpath = self.imgpath_list[i]
img = cv2.imread(imgpath)
img = cv2.resize(img, dsize = (256, 256))
img = img/255
img = torch.from_numpy(img.astype(np.float32)).clone()
img = img.permute(2, 0, 1)
labelpath = self.labelpath_list[i]
label = Image.open(labelpath)
label = np.asarray(label)
label = cv2.resize(label, dsize = (256, 256))
label = torch.from_numpy(label.astype(np.float32)).clone()
label = torch.nn.functional.one_hot(label.long(), num_classes=4)
label = label.to(torch.float32)
label = label.permute(2, 0, 1)
data = {"img": img, "label": label}
return data
def __len__(self):
return len(self.imgpath_list)
BATCH_SIZE = 8
train_dataset = Dataset(df)
train_loader = DataLoader(train_dataset,
batch_size=BATCH_SIZE,
num_workers=4,
shuffle=True)
val_dataset = Dataset(val_df)
val_loader = DataLoader(val_dataset,
batch_size=BATCH_SIZE,
num_workers=4,
shuffle=True)
test_dataset = Dataset(test_df)
test_loader = DataLoader(test_dataset,
batch_size=1,
num_workers=4)
Unetを構築します。nn.ModuleListを使用することで短く書くことも可能ですが、可読性が低下するため以下のように書いています。
今回、デコーダーのup-Convolution(高さと幅を2倍にしつつ、チャンネル数を半分にする)については以下の方法で実装しています。
- nn.Upsampleを使用してup-Convolutionを行い、直後にnn.Conv2d(カーネルサイズは2×2を採用していますが、1×1でもよいと思います。)でチャンネル数を半分にする
以下の2つの方法でも実装可能です。
- up-Convolutionの直前のConvolutionブロックでチャネル数を半分にする。その後nn.Upsampleを使用してup-Convolutionを行う。
- nn.ConvTranspose2dを使用してup-Convolutionを行う。
class TwoConvBlock(nn.Module):
def __init__(self, in_channels, middle_channels, out_channels):
super().__init__()
self.conv1 = nn.Conv2d(in_channels, middle_channels, kernel_size = 3, padding="same")
self.bn1 = nn.BatchNorm2d(middle_channels)
self.rl = nn.ReLU()
self.conv2 = nn.Conv2d(middle_channels, out_channels, kernel_size = 3, padding="same")
self.bn2 = nn.BatchNorm2d(out_channels)
def forward(self, x):
x = self.conv1(x)
x = self.bn1(x)
x = self.rl(x)
x = self.conv2(x)
x = self.bn2(x)
x = self.rl(x)
return x
class UpConv(nn.Module):
def __init__(self, in_channels, out_channels):
super().__init__()
self.up = nn.Upsample(scale_factor=2, mode="bilinear", align_corners=True)
self.bn1 = nn.BatchNorm2d(in_channels)
self.conv = nn.Conv2d(in_channels, out_channels, kernel_size = 2, padding="same")
self.bn2 = nn.BatchNorm2d(out_channels)
def forward(self, x):
x = self.up(x)
x = self.bn1(x)
x = self.conv(x)
x = self.bn2(x)
return x
class UNet_2D(nn.Module):
def __init__(self):
super().__init__()
self.TCB1 = TwoConvBlock(3, 64, 64)
self.TCB2 = TwoConvBlock(64, 128, 128)
self.TCB3 = TwoConvBlock(128, 256, 256)
self.TCB4 = TwoConvBlock(256, 512, 512)
self.TCB5 = TwoConvBlock(512, 1024, 1024)
self.TCB6 = TwoConvBlock(1024, 512, 512)
self.TCB7 = TwoConvBlock(512, 256, 256)
self.TCB8 = TwoConvBlock(256, 128, 128)
self.TCB9 = TwoConvBlock(128, 64, 64)
self.maxpool = nn.MaxPool2d(2, stride = 2)
self.UC1 = UpConv(1024, 512)
self.UC2 = UpConv(512, 256)
self.UC3 = UpConv(256, 128)
self.UC4= UpConv(128, 64)
self.conv1 = nn.Conv2d(64, 4, kernel_size = 1)
self.soft = nn.Softmax(dim = 1)
def forward(self, x):
x = self.TCB1(x)
x1 = x
x = self.maxpool(x)
x = self.TCB2(x)
x2 = x
x = self.maxpool(x)
x = self.TCB3(x)
x3 = x
x = self.maxpool(x)
x = self.TCB4(x)
x4 = x
x = self.maxpool(x)
x = self.TCB5(x)
x = self.UC1(x)
x = torch.cat([x4, x], dim = 1)
x = self.TCB6(x)
x = self.UC2(x)
x = torch.cat([x3, x], dim = 1)
x = self.TCB7(x)
x = self.UC3(x)
x = torch.cat([x2, x], dim = 1)
x = self.TCB8(x)
x = self.UC4(x)
x = torch.cat([x1, x], dim = 1)
x = self.TCB9(x)
x = self.conv1(x)
return x
GPU、最適化アルゴリズムの設定を行います。
device = torch.device("cuda:0" if torch.cuda.is_available() else "cpu")
unet = UNet_2D().to(device)
optimizer = optim.Adam(unet.parameters(), lr=0.001)
損失関数を設定します。損失はTversky LossとBCEWithLogits Lossの平均としました。これらの関数は損失関数内でソフトマックス関数を処理する為、UNetの最後にソフトマックス関数を適用していません。
TverskyLoss = smp.losses.TverskyLoss(mode='multilabel', log_loss=False)
BCELoss = smp.losses.SoftBCEWithLogitsLoss()
def criterion(pred,target):
return 0.5*BCELoss(pred, target) + 0.5*TverskyLoss(pred, target)
学習を行います。
history = {"train_loss": []}
n = 0
m = 0
for epoch in range(15):
train_loss = 0
val_loss = 0
unet.train()
for i, data in enumerate(train_loader):
inputs, labels = data["img"].to(device), data["label"].to(device)
optimizer.zero_grad()
outputs = unet(inputs)
loss = criterion(outputs, labels)
loss.backward()
optimizer.step()
train_loss += loss.item()
history["train_loss"].append(loss.item())
n += 1
if i % ((len(df)//BATCH_SIZE)//10) == (len(df)//BATCH_SIZE)//10 - 1:
print(f"epoch:{epoch+1} index:{i+1} train_loss:{train_loss/n:.5f}")
n = 0
train_loss = 0
train_acc = 0
unet.eval()
with torch.no_grad():
for i, data in enumerate(val_loader):
inputs, labels = data["img"].to(device), data["label"].to(device)
outputs = unet(inputs)
loss = criterion(outputs, labels)
val_loss += loss.item()
m += 1
if i % (len(val_df)//BATCH_SIZE) == len(val_df)//BATCH_SIZE - 1:
print(f"epoch:{epoch+1} index:{i+1} val_loss:{val_loss/m:.5f}")
m = 0
val_loss = 0
val_acc = 0
torch.save(unet.state_dict(), f"./train_{epoch+1}.pth")
print("finish training")
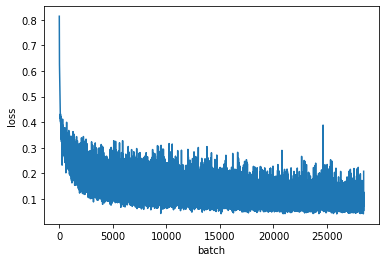
損失の推移をプロットします。学習率の減衰を忘れていました...
plt.plot(history["train_loss"])
plt.xlabel('batch')
plt.ylabel('loss')
testデータに対して予測を行います。
validation用データセットの損失が最も低かったエポックの重みを使用します。
model = UNet_2D()
model.load_state_dict(torch.load("./train_12.pth"))
model.eval()
with torch.no_grad():
data = next(iter(test_loader))
inputs, labels = data["img"], data["label"]
outputs = model(inputs)
loss = criterion(outputs, labels)
print("loss: ",loss.item())
sigmoid = nn.Sigmoid()
outputs = sigmoid(outputs)
pred = torch.argmax(outputs, axis=1)
pred = torch.nn.functional.one_hot(pred.long(), num_classes=4).to(torch.float32)
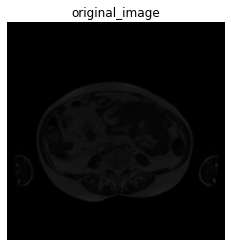
元の画像と予測結果を表示します。
plt.figure()
plt.imshow(data["img"][0,:,:,:].permute(1, 2, 0))
plt.title("original_image")
plt.axis("off")
plt.figure()
classes = ["background","large_bowel","small_bowel","stomach"]
fig, ax = plt.subplots(2, 4, figsize=(15,8))
for i in range(2):
for j, cl in enumerate(classes):
if i == 0:
ax[i,j].imshow(pred[0,:,:,j])
ax[i,j].set_title(f"pred_{cl}")
ax[i,j].axis("off")
else:
ax[i,j].imshow(data["label"][0,j,:,:])
ax[i,j].set_title(f"label_{cl}")
ax[i,j].axis("off")
上段が予測結果になります。精度よく予測できているのではないでしょうか。腸の膜は全く予測できていないのは、ピクセル数が少ないため損失に反映されにくいからだと考えています。