はじめに
2日連続の投稿です。
今回は、前回取得したPubmedデータを利用して、簡単なクラスタリングを行います。
①pubmedの検索結果から、authors listを取得
②authors listにアソシエーション分析を行い、共著が多い人物セットを見出す
③アソシエーション分析結果に対して、スペクトラルクラスタリングを行う
ライブラリ
import time
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns
import chromedriver_binary
from selenium import webdriver
import pyfpgrowth
import networkx as nx
from sklearn import cluster
%matplotlib inline
①pubmedからデータ取得
前回の投稿と同じ関数(get_pubmed_info)を使っているので、関数の内部は割愛します。
解析対象として、研究室内の派閥(グループ)が多そうな教授をチョイスしました。
data = get_pubmed_info("miura masayuki")
author_list = [data[i][2].split(",") for i in range(len(data))]
author_list = [[author.split("(")[0].replace("\n"," ").strip(".").strip()
for author in author_list[i]] for i in range(len(author_list))]
②アソシエーション分析
pyfpgrowthで、先程取得したデータに対して、アソシエーション分析を行います。
patterns = pyfpgrowth.find_frequent_patterns(author_list, 3)
G = nx.Graph()
for key in patterns.keys():
if len(key) == 2:
G.add_path(key)
pr = nx.pagerank(G)
plt.figure(figsize=(20,20))
nx.draw_networkx(G, node_color=list(pr.values()), cmap=plt.cm.Reds, font_size=15, node_size=5000)
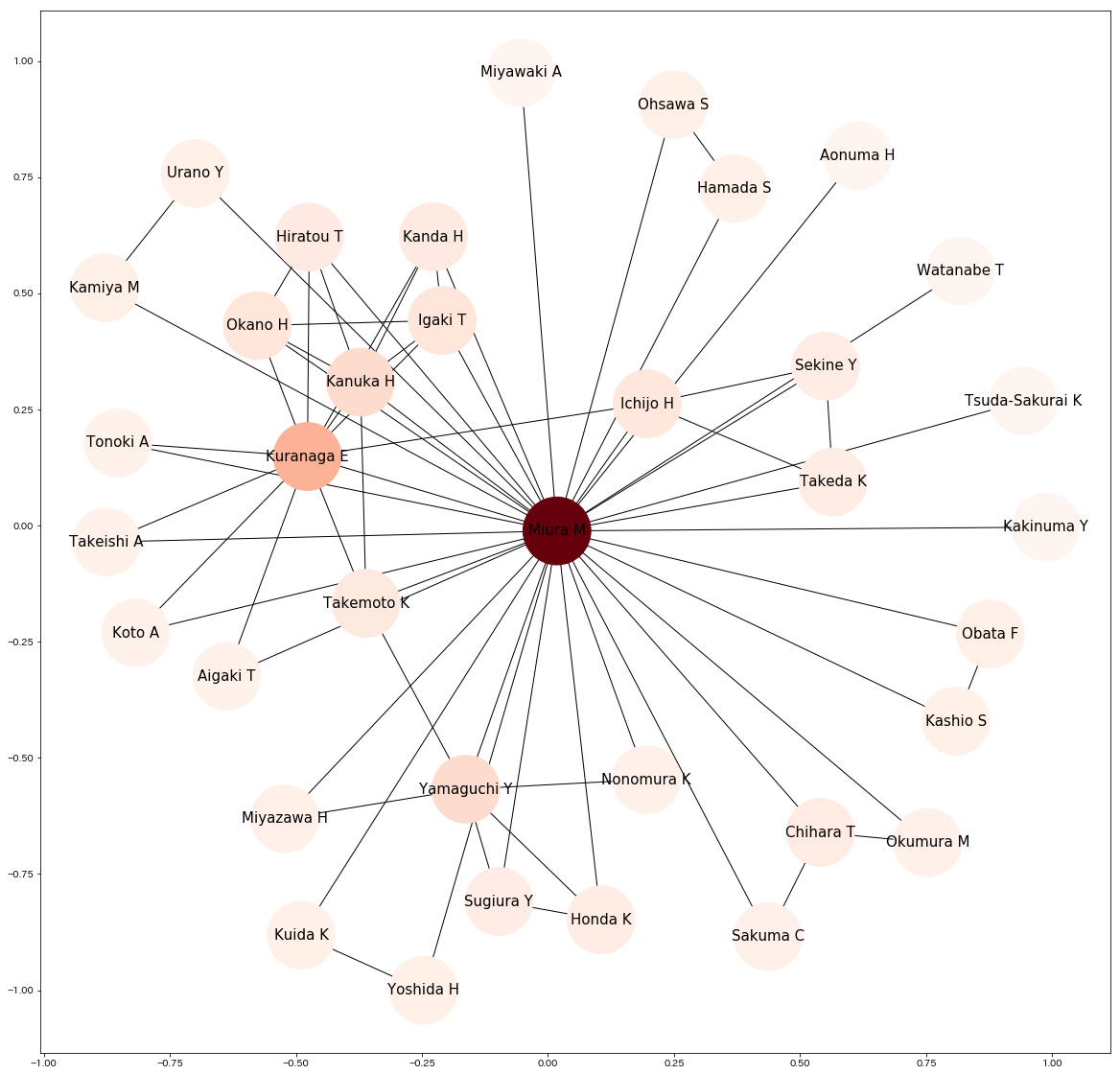
得られた結果をnetworkxで可視化
pagerankで、ネットワークの重要度を色付けしています(色が濃いほど重要)。
このネットワーク図からでも、クラスターの存在は感じられます。
③スペクトラルクラスタリング
pyfpgrowthで得たデータを整形して、スペクトラルクラスタリングを行います。
データ整形過程
key_list = []
value_list = []
for key, value in patterns.items():
if len(key) == 2:
key_list.append(key)
value_list.append(value)
df = pd.DataFrame()
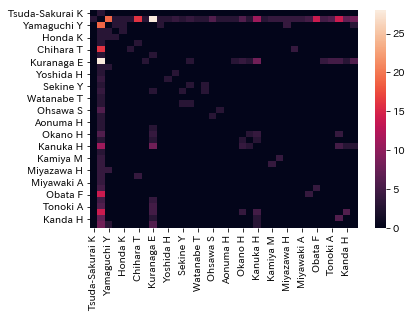
for (i, j), num in zip(key_list, value_list):
df.loc[i, j] = num
df.loc[j, i] = num
df = df.loc[df.columns].fillna(0)
sns.heatmap(df)
いざスペクトラルクラスタリング!
spectral = cluster.SpectralClustering(n_clusters=9, affinity="precomputed")
result = spectral.fit(df.values)
result_se = pd.Series(result.labels_, index=df.index)
plt.figure(figsize=(20,20))
nx.draw_networkx(G, node_color=result_se, cmap=plt.cm.Accent, font_size=15, node_size=5000)
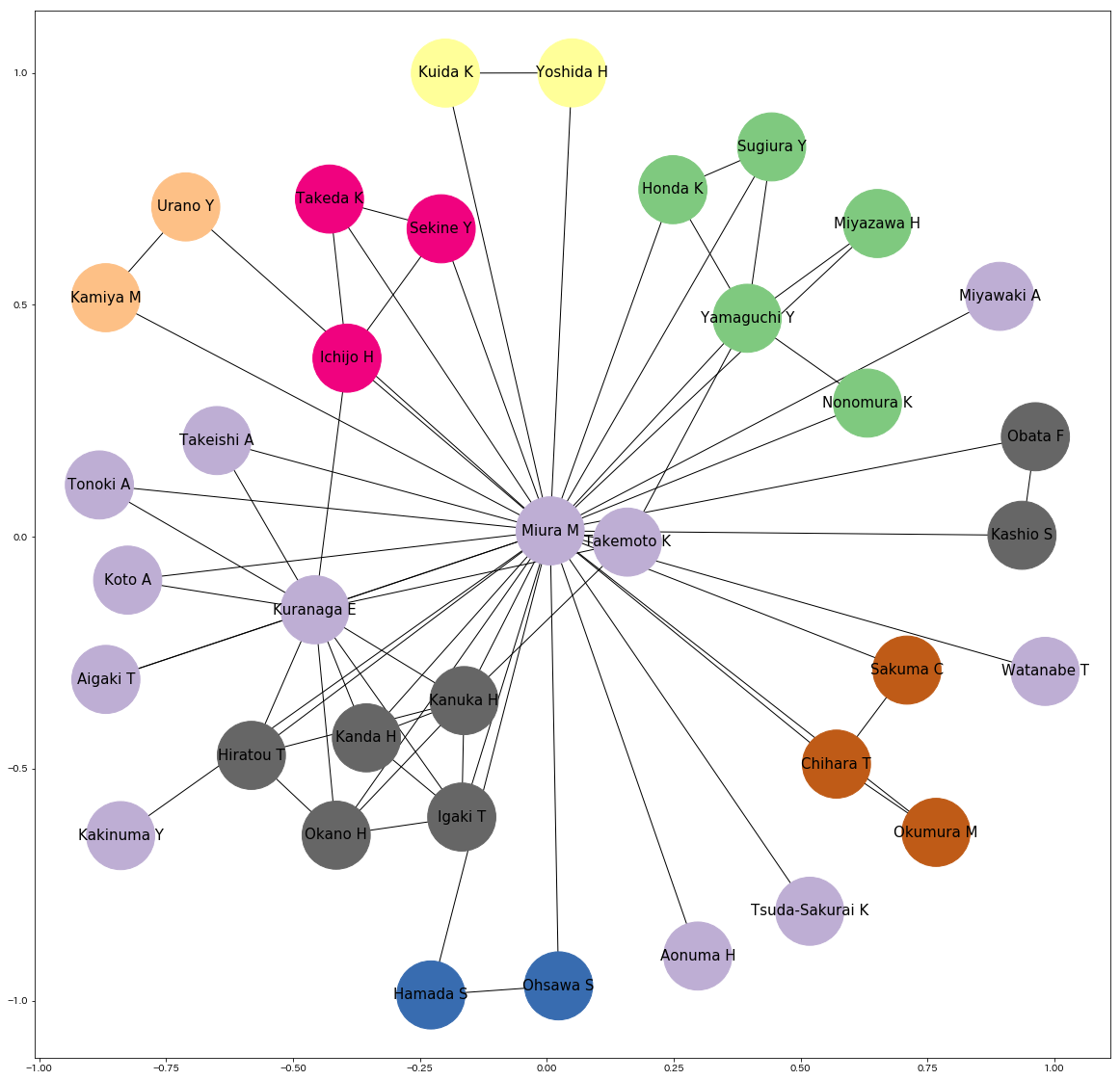
クラスタリング結果を先程のネットワーク図に反映させました。
キレイに研究グループごとに分かれていることがわかります。
濃い緑は哺乳類系、茶色はショウジョウバエ神経系、ピンク色は外部の共同研究先といった具合になっています。
参考
http://www.f.u-tokyo.ac.jp/~genetics/
終わりに
かなりきれいにクラスタリング出来ました。