はじめに
上の記事を参考にしてMaterial ProjectというデータベースのAPIと、Material Projectと連動した物質解析のためのライブラリであるPymatgenを利用して、バンドギャップの予測に挑戦してみました。
検証内容
Material Projectのバンドギャップに関するデータを見てみると、ナローバンドギャップ系の材料はバンドギャップが0となっている場合が多いため、今回は説明変数に電子密度、目的変数をバンドギャップとしてRandom Forest Regressionを用いてナローバンド半導体として有名なInSbのバンドギャップを予測してみました。
動作環境
Python 3.6.5:: Anaconda
scikit-learn 0.19.1
pymatgen 2018.6.11
Material Projectからデータを取得
Material ProjectのAPIの取得にはMaterial Projectに登録(無料)し、ダッシュボードの”Generate API key”から取得。また取得するデータはバンドギャップがゼロ以外の材料系
import csv, itertools
from pymatgen import MPRester
API_KEY = 'Your API key'
all_symbols = ["H", "He", "Li", "Be", "B", "C", "N", "O",\
"F", "Ne", "Na", "Mg", "Al", "Si", "P", "S",\
"Cl", "Ar", "K", "Ca", "Sc", "Ti", "V", "Cr",\
"Mn", "Fe", "Co", "Ni", "Cu", "Zn", "Ga", "Ge",\
"As", "Se", "Br", "Kr", "Rb", "Sr", "Y", "Zr", "Nb",\
"Mo", "Tc", "Ru", "Rh", "Pd", "Ag", "Cd", "In", "Sn",\
"Sb", "Te", "I", "Xe", "Cs", "Ba", "La", "Ce", "Pr",\
"Nd", "Pm", "Sm", "Eu", "Gd", "Tb", "Dy", "Ho", "Er",\
"Tm", "Yb", "Lu", "Hf", "Ta", "W", "Re", "Os", "Ir", "Pt",\
"Au", "Hg", "Tl", "Pb", "Bi", "Po", "At", "Rn", "Fr", "Ra",\
"Ac", "Th", "Pa", "U", "Np", "Pu", "Am", "Cm", "Bk", "Cf", "Es",\
"Fm", "Md", "No", "Lr"]
allBinaries = itertools.combinations(all_symbols, 2)
with MPRester(API_KEY) as m:
with open('data.csv', 'w', newline='') as f:
fieldnames = ['Density', 'Band Gap']
writer = csv.DictWriter(f, fieldnames=fieldnames)
writer.writeheader()
count = 0
# writing bandgap and density in 'data.csv'
for system in allBinaries:
if count > 500:
break
systems = m.get_data(system[0] + '-' + system[1], data_type='vasp')
for material in systems:
if material['band_gap'] != 0.0:
count = count + 1
writer.writerow({'Density': material['density'], 'Band Gap': material['band_gap']})
break
Random Forest Regression
gete_data.pyから得られるdata.csvよりランダムフォレスト回帰を適用
import numpy as np
import matplotlib.pyplot as plt
import pandas as pd
# Importing the dataset
dataset = pd.read_csv('data.csv')
X = dataset.iloc[:,0:1].values
y = dataset.iloc[:,1].values
# Splitting the dataset into the Training set and Test set
from sklearn.model_selection import train_test_split
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size = 0.2, random_state = 0)
# Fitting the Regression Model to the dataset
from sklearn.ensemble import RandomForestRegressor
regressor = RandomForestRegressor(n_estimators=100, random_state=0, )
regressor.fit(X_train, y_train)
# Visualising the Regression results for higher and smoother curve
X_grid = np.arange(min(X_train), max(X_train), 0.01)
X_grid = X_grid.reshape(len(X_grid), 1)
plt.scatter(X_train, y_train, color = 'red')
plt.plot(X_grid, regressor.predict(X_grid), color='blue')
plt.title('Random Forest Regression')
plt.xlabel('Density [eV]')
plt.ylabel('Band Gap [eV]')
plt.show()
回帰してみた結果はこんな感じ↓
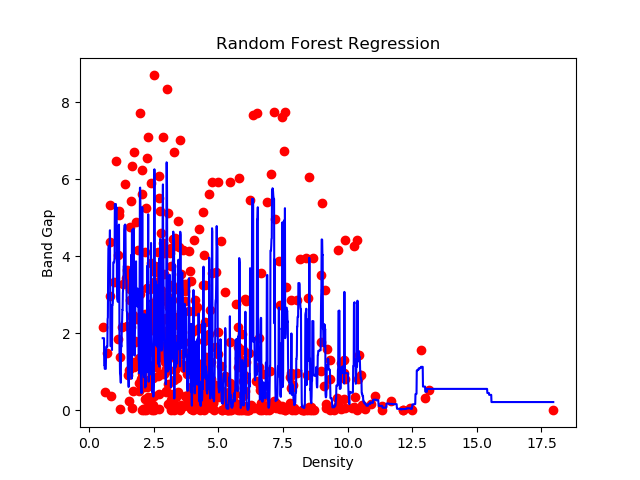
また、InSbの電子密度は5.772 g/cm3 (Wikipedia )をテストに用いる説明変数に使用。
結果は予測したバンドギャップが 1.90 eVに対して、Wikipedaでは 0.17 eVです。全然、違いますね。。。そもそもモデルが単純すぎるというのと、特徴量の選択範囲がおかしいですよね。。興味があれば、pymatgenの公式のドキュメントを読んでみてください!