この記事は、http://d.hatena.ne.jp/fukuit/20151031/1446295539 からの転載です。
GEOのデータを使って、特定の条件での特定遺伝子の発現量を求める方法を何人かに聞かれたので、需要があるのかな?と思って、書いておく。
DataSetsを使いこなす
DataSetsとは、NCBIによってcurationされたGEOデータのことだそうだ。
http://www.ncbi.nlm.nih.gov/geo/ を開いたら、まずはDataSetsのところをクリックする。
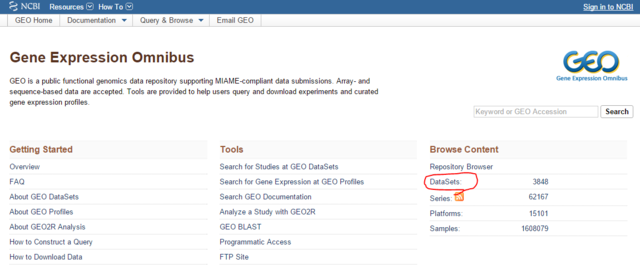
すると、登録されているDataSetsの一覧画面になる。ここで、自分の欲しい条件で検索する。
とりあえず、Triple Negative Breast Cancerで検索してみたら、2件ヒットした。
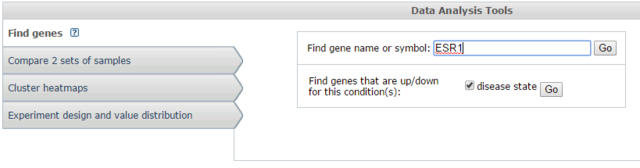
このうちのGDS4069の方を見ることにして、Data Analysis Toolsというところをクリックする。
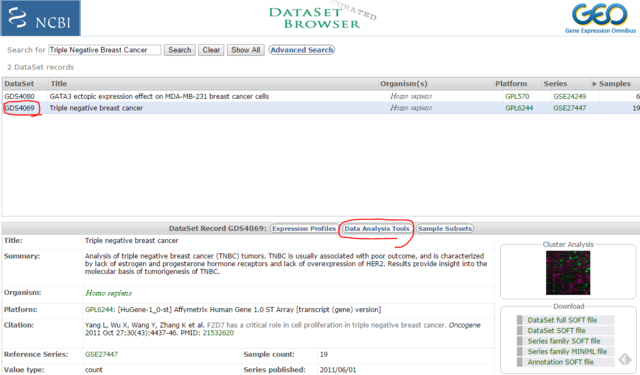
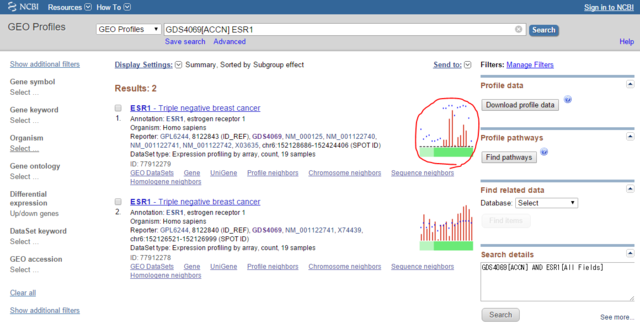
ここで遺伝子の発現量を検索できるので、試しにESR1を検索してみる。
2件ヒットしたようだ。
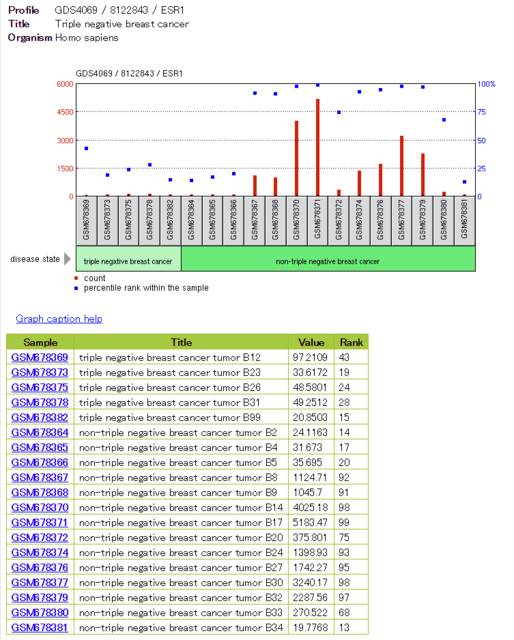
すると、triple negativeではほとんど発現していないが、non-triple negativeの一部では高発現でした、ということが分かった。
実に簡単だ。簡単すぎる。もし、自分が考えている実験条件に近いものがDataSetsとして登録されていたら、実験をする必要はないかもしれない。事前検討を減らせるかもしれない。
という具合に、研究室で「ちょっとPCに詳しい人」程度のBioInformaticianは失業の危機である。