この記事は、http://d.hatena.ne.jp/fukuit/20151101/1446341605 からの転載です。
Seriesの方を使う例も挙げておく。
Seriesを使いこなす。
論文を書いたときに、マイクロアレイの発現データを寄託しておくよう求められるが、その寄託先の一つがGEOであり、その実験ごとのまとめがSeriesである。
Webブラウザでの作業
Expression Profilinig by arrayと、Homo Sapiensをクリックして、絞り込みを行う。
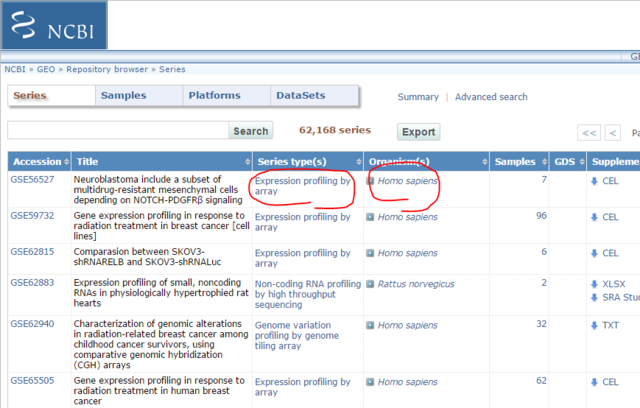
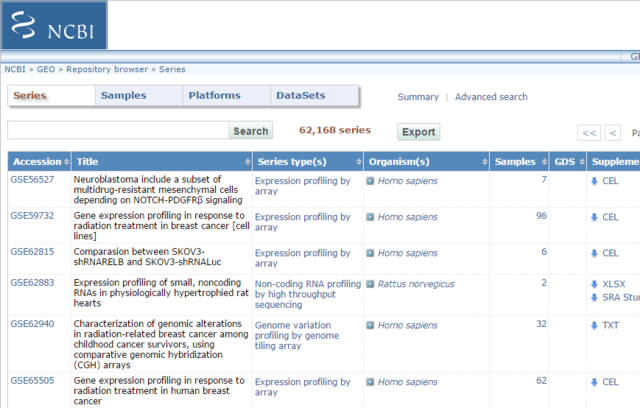
例として、breastを検索すると、958 sampleも登録されている。
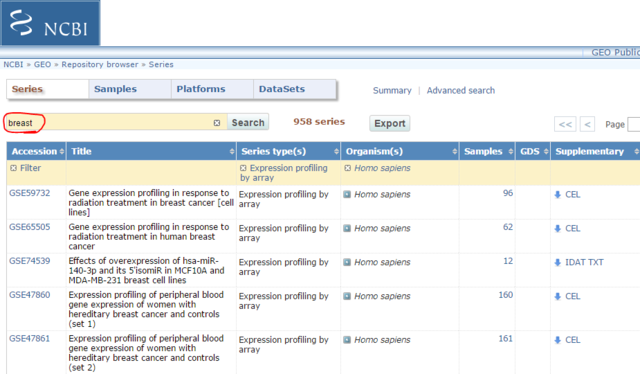
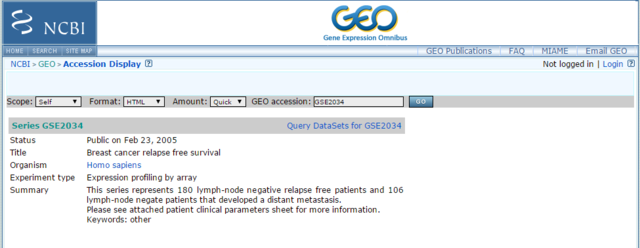
GSE2034に予後のデータがあるっぽいので、これを選択。
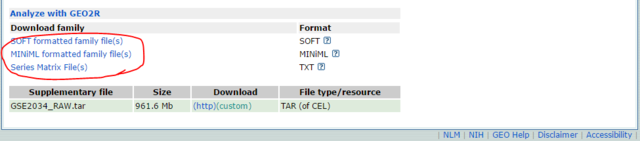
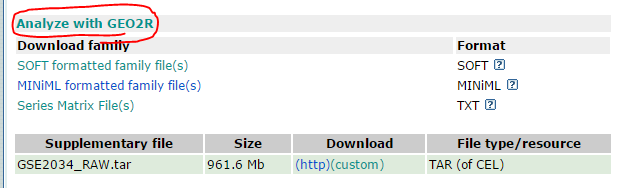
SOFT Formatted FileとSeries Matrixファイルをダウンロードしておく。
ちなみに、gzipで圧縮されているので、これを解凍する。Windowsならば、Lhaplus辺りを使えばいい。CygwinやMingWのgzipを使いこなせる人は、もちろんどうぞ。
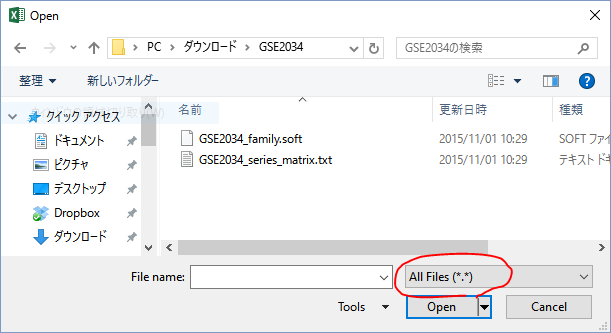
以降はExcelでの作業
SOFTファイルは、用いられたarrayのアノテーションファイルである。このファイルがないと、どのプローブにどんなIDが付与されているのか?が分からないので、解析ができない。
Series Matrixファイルは、解析済の発現量が記載されている。このファイルがあれば、目的の遺伝子の発現量を取り出すことができるが、そのためには、SOFTファイルを参照して、目的の遺伝子には、どのようなIDが振られたprobeが設定されているか?を知る必要がある。
SOFTファイルもSeries Matrixファイルもテキストファイルなので、Excelで開くことができるが、注意点はExcelの「ソレっぽい表記は日付と自動判断する機能」を使わないようにすること、である。遺伝子名(GENE SYMBOL)には、OCT4、DEC1のようなものがあり、Excelはこれをそれぞれ10月4日、12月1日に自動変換する。まだ、これらは推測可能だから良い。問題なのは、MARCH1遺伝子もMARC1遺伝子も双方とも3月1日に変換してしまうので、どっちがどっちだか分からなくなってしまうのである。なので、これは要注意。
このGSE2034のSOFTファイルの場合、23247行目以降がmicroarrayのアノテーション情報であり、K列にGENE SYMBOLが入っているので、ソコを注意する。
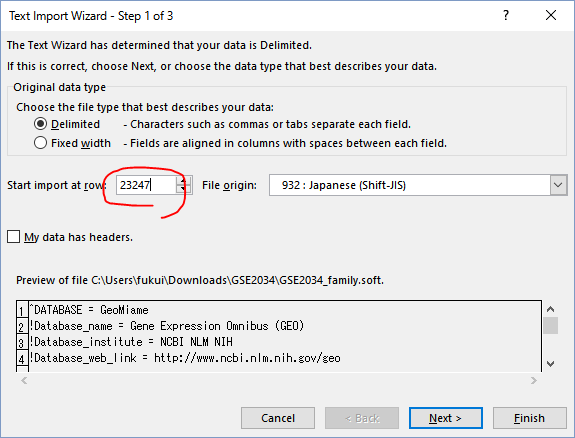
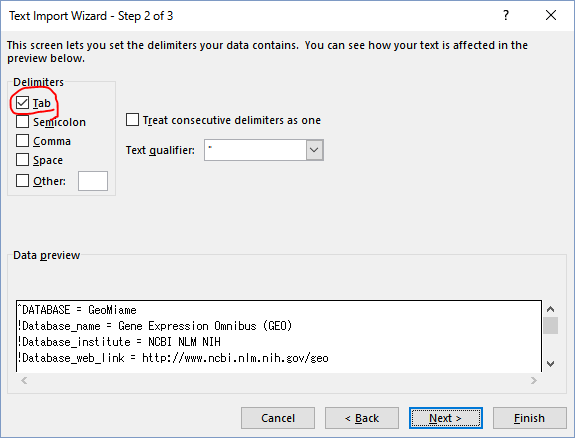
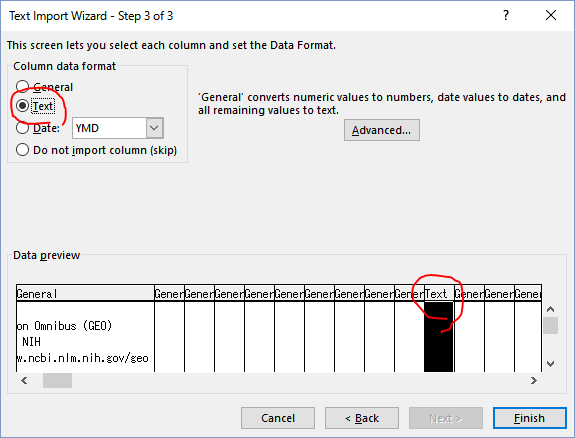
ExcelでSOFTファイルを開いたら、例えば関心のある遺伝子HIF1AをGENE SYMBOLの列で検索すると、IDが200989_atであることが分かった。
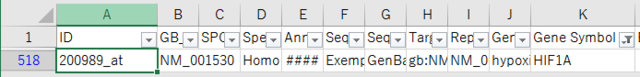
このID: 200989_atをメモっておいて、次はSeries MatrixファイルをExcelで開く。
そして、一列目ID_REFが"200989_at"になっているものを検索する。
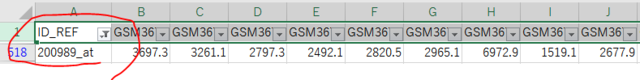
この行が、GSE2034におけるHIF1Aの発現だ。
このデータを使うと、再発有無の違いでHIF1Aの発現量がどうだったか?を調べることもできる。
さて、以上の行為をGEOのGEO2RのWeb画面上で実施することもできる。例えば、以下のようなグラフを描画するとか。
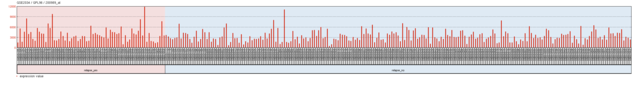
GEO2R
GSE2034の画面の下の方に「Analyze with GEO2R」というリンクがある。これをクリックする。
すると、GEO2Rの画面が開く。
これはGEO上でRを使って解析してくれて、その結果を戻してくれる便利機能だ。
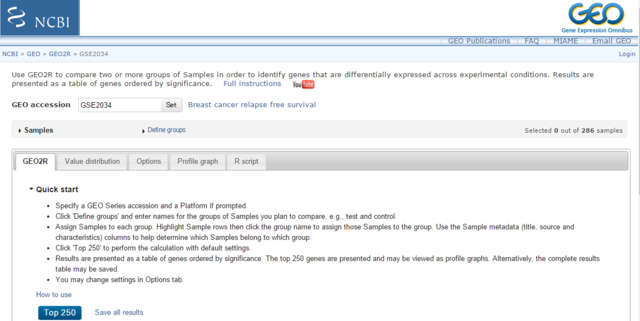
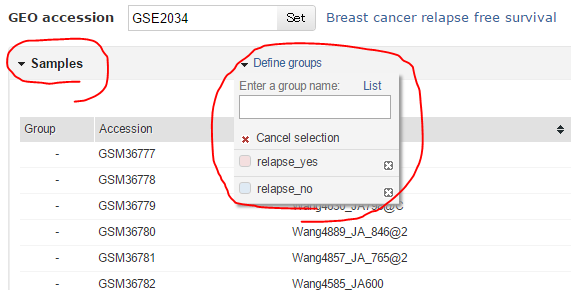
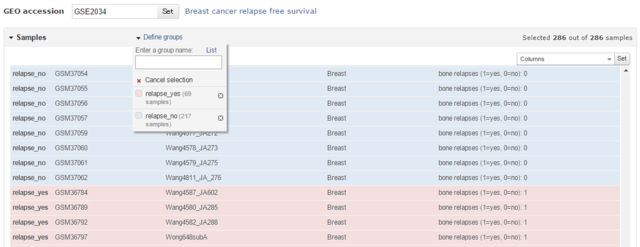
この画面で「Samples」をクリックすると、サンプルの一覧が表示される。Groupsをクリックして、自分でグループ名を定義したうえで、各サンプルにグループを割り当てる。
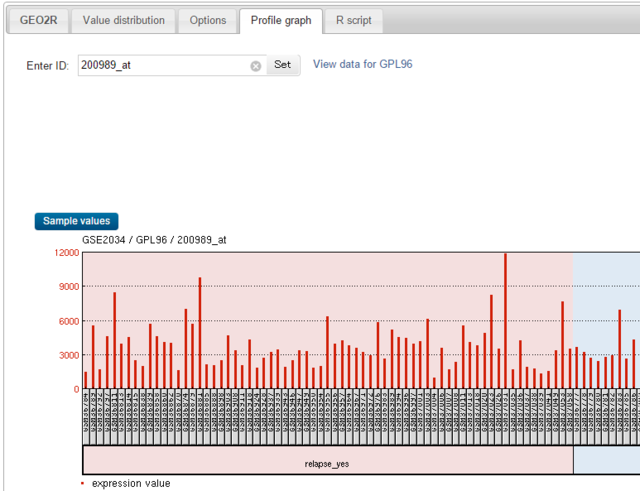
「Profile Graph」のタブをクリックして、そこに例えばさっきのHIF1A遺伝子プローブのID「200989_at」を入力する。
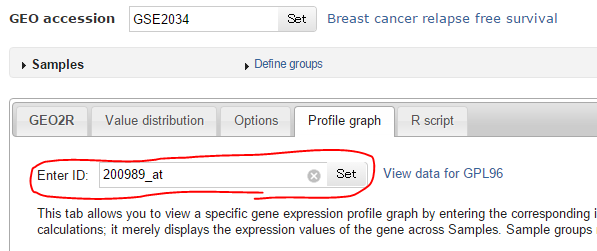
「Set」ボタンを押すと、グループ別に色分けされて、発現量のグラフが表示される。複数プローブに対応していないのが残念だが、複数のプローブを同時に表示したグラフを描画したいのであれば、上述のようにSeries Matrixファイルをダウンロードして自前でExcelでやればいい。
また、上記の処理を実行するのに使ったRのscriptを見ることもできる。
いや、ホントに「PCにちょっと詳しいだけのBioInformaticsの人」とか、失業モンですよ。