機械学習ってなんだろうっていうと、思いつくままに書くと、こんな感じかな。
- 分類
- 回帰
- 判別
他にもあるかもしれないけれど、思いつかないからいいや。
Rでこの辺りのチュートリアルをすすめる時には、irisデータを使うんだけれど、Pythonにはあるのかな。→あるらしい。
Rで機械学習
分類&判別の例
まずは、Rでやってみる。RでSVMやらNeuralNetやらNaiveBayseやらRandomForestやらを使って識別器を作るのは、なんていうか作るだけならほとんど何も考える必要がない。それぞれのクラスタリング手法を使って、トレーニングセットを使って識別器を作り、テストセットがその識別器で正しく分類できるかどうかを評価するという流れだ。
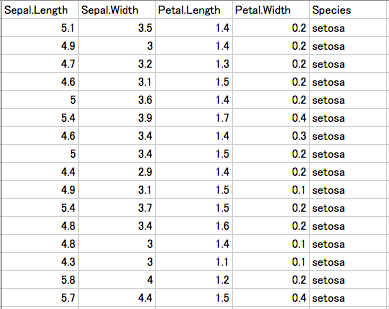
Rではdata(iris)すれば、ショウブだかアヤメだかカキツバタだかのデータを利用できる。
これは、萼長、萼幅、花弁長、花弁幅を説明変数に、目的変数にirisの品種(setosa , versicolor , virginica )を持った全150 sampleのdata.tableだ。
まずは、sample()で半数をトレーニングセットに、残りをテストセットになるように、ランダムに分割する。
## irisデータの読み込み
data(iris)
## 半分をトレーニングセットにするために、ランダムに選択する
train_ids <- sample(nrow(iris), nrow(iris)*0.5)
## トレーニングセットの作成
iris.train <- iris[train_ids,]
## 残り半分をテストセットに
iris.test <- iris[-train_ids,]
ところで、irisの3品種、setosa , versicolor, virignicaは以下のような花だ。みんな同じやん!コレを花びらの形状で区別するとか、罰ゲームですやん。
### SVM実行
library(kernlab)
iris.svm <- ksvm(Species~., data=iris.train)
svm.predict <- predict(iris.svm, iris.test)
### 結果表示
table(svm.predict, iris.test$Species)
### neuralnet実行
library(nnet)
iris.nnet<-nnet(Species ~ ., data = iris.train, size = 3)
nnet.predict <- predict(iris.nnet, iris.test, type="class")
### 結果表示
table( nnet.predict, iris.test$Species)
### naivebayes実行
library(e1071)
iris.nb <- naiveBayes(Species~., iris.train)
nbayes.predict <- predict(iris.nb, iris.test)
### 結果表示
table(nbayes.predict, iris.test$Species)
### randomforest実行
library(randomForest)
iris.rf <- randomForest(Species~., iris.train)
rf.predict <- predict(iris.rf, iris.test)
### 結果表示
table(rf.predict, iris.test$Species)
実行してみると、どんな手法を使っても、だいたい正解率73/75くらいで識別できるようになる。百発百中とはいかないんだけれど、それはパラメーター調節とかでなんとかなるのかもしれない(ならないのかもしれない)。
さて、同じことをPythonでやってみるためには、scikit-learnを使う。詳しいことは、scikit-learnのチュートリアル に書いてある。
Pythonで識別
PythonでSVMをできるようになってみる
そもそも「上記のようにRで簡単にできたこと」をなぜ自分にとって未知のPythonでやるのか?というと、Pythonでやってみたいからとしか言いようがない。ソコに山があったら登るし、水たまりがあったらハマるし、据え膳があったら喰うのである。
Anacondaインストール済ならscikit-learnはインストール済なので、importする。importするんだけれど、ライブラリ名はsklearnだ。このライブラリにはdatasetsとして、irisもloadできるようになっている。
from sklearn import svm, datasets
iris = datasets.load_iris()
中身については、print(iris.data)とか、print(iris.target)とかすると分かるんだけれど、iris.dataの方に説明変数が、iris.targetの方に目的変数が入っている。これを、トレーニング用のデータセットと、テスト用のデータセットに分割する。Rだとsample()関数を使ったけれど、scikit-learnの場合には、sklearn.cross_varidationにtrain_test_split()というメソッドがある。これで、Rでやった時と同様に、半分をトレーニングセット(iris_data_train, iris_target_train)と、テストセット(iris_data_test, iris_target_test)に2分割した。
from sklearn import svm, datasets
from sklearn.cross_validation import train_test_split
import numpy as np
iris = datasets.load_iris()
iris_data_train, iris_data_test, iris_target_train, iris_target_test = train_test_split(iris.data, iris.target, test_size=0.5)
このトレーニングセットを使って識別器を作る。SVMにもいろいろあって、線形か?非線形か?とかパラメータをいろいろ設定する必要があるんだけれど、ここは全部デフォルトで。
svc = svm.SVC()
svc.fit(iris_data_train, iris_target_train)
svc.predict(iris_data_test)
fit()とpredict()は2行に分けて書いたけれど、初期化して、トレーニングデータ食わせて、テストデータを食わせるだけなので、1行で書いてもいい。
iris_predict = svm.SVC().fit(iris_data_train, iris_target_train).predict(iris_data_test)
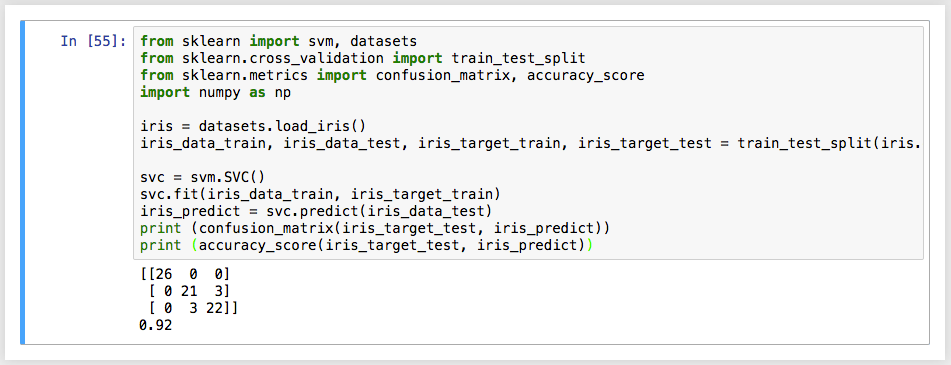
このsvc.predict()の結果(iris_predict)と、iris_target_testとの一致率を見れば良い。Rでは、table(svm.predict, iris.test$Species) のように表形式にして出力したので、同じようにやってみる。この表を confusion_matrix という。accuracy_scoreで、正解率を手っ取り早く出すこともできる。
from sklearn.metrics import confusion_matrix, accuracy_score
print (confusion_matrix(iris_target_test, iris_predict))
print (accuracy_score(iris_target_test, iris_predict))
confusion_matrix()は、公式ドキュメントによると、第一引数が真値で、第二引数が識別器による判別値になっているようなので、それぞれそのように渡す。
Rよりは単純にタイプ量が多いけれど、同じような結果が得られるようにはなった。
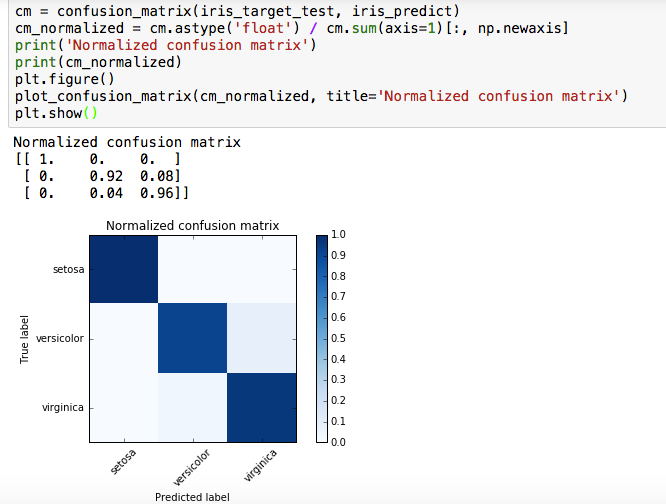
上記のドキュメントによると、これを図に示すこともできる。まず、confusion_matrixの各行が合計1になるように正規化する。この式をパッと自分では書けないな。
cm = confusion_matrix(iris_target_test, iris_predict)
cm_normalized = cm.astype('float') / cm.sum(axis=1)[:, np.newaxis]
この正規化したconfusion_matrixを使って、matplotlibの機能を使ってheatmap化する。ちなみに、何回もsvc.predict()をやって、そのたびに結果が微妙に異なるので、上図と下図はconfusion_matrixの中身が若干異なる。
def plot_confusion_matrix(cm, title='Confusion matrix', cmap=plt.cm.Blues):
''' confusion_matrixをheatmap表示する関数
Keyword arguments:
cm -- confusion_matrix
title -- 図の表題
cmap -- 使用するカラーマップ
'''
plt.imshow(cm, interpolation='nearest', cmap=cmap)
plt.title(title)
plt.colorbar()
tick_marks = np.arange(len(iris.target_names))
plt.xticks(tick_marks, iris.target_names, rotation=45)
plt.yticks(tick_marks, iris.target_names)
plt.tight_layout()
plt.ylabel('True label')
plt.xlabel('Predicted label')
今回は、svc=svm.SVC()という具合に全部デフォルトだったんだけれど、もちろん分類のためのkernelはいろいろ使えるはず。
scikit-learnの公式サイトのExamplesのページには、いろいろなデモとかチュートリアルとか載ってるので、いざ必要なときには、ここからヒントを探ることができそうだ。
Rでやったように、じゃあNeuralNetならnaiveBayesならrandomForestならどうなのか、というのも気になるけれど、多分、だいたい同じ(調べてないけど)。
今回の結論
機械学習の一端に触れた気分なんだけれど、結局irisってのがショウブなのかアヤメなのかカキツバタなのかは分からずじまいなのであった。
追記
当初「Pythonで機械学習のさわりをやってみる」というタイトルでしたが、「さわりは最も盛り上がる所の意味だから、タイトルはふさわしくない」とのことだったので、修正しました。
「PythonでSVMで判別器を作れる」のって、個人的には盛り上がるポイントだと思うんだけど、独りよがりだったみたいです。SVMごときで機械学習を語ってすみませんでした。出直してきます。